PyMCでベイズ統計を始める
PyMCによるベイズ統計の始め方
Peter D. Hoff 著, 入江薫 菅澤翔之助 橋本真太郎 訳
標準ベイズ統計学 (A First Course in Bayesian Statistical Methods)
2022-06-01
https://www.asakura.co.jp/detail.php?book_code=12267
Osvaldo Martin 著, 金子武久 訳
Pythonによるベイズ統計モデリング (Bayesian Analysis with Python)
2018-06-26
https://www.kyoritsu-pub.co.jp/book/b10003944.html
PRMLや標準ベイズ統計学を読み進めている時に感じた、初学者として戸惑った観点などを忘れないうちに書き留めておきます。
ベイズの考え方から初めて、実際にPyMCでデータを記述していきます。
学部初年次の確率統計の講義でよくやる頻度論的な仮説検定で統計恐怖症になった人でも、ベイズを使うと直感的に検定を理解できるのでベイジアンに改宗すると世界が広まります。
この記事では、まず完全初心者のための「ツールとしての式の読み方」について話してから、「標準ベイズ統計学(FCBSM)」の1~2章と「Pythonによるベイズ統計モデリング(BAwP)」の1~3章を読みながらデータ分析の初歩に必要なベイズ統計のおさらいをします。
確率統計の書籍を読む前に、入り口でつまずいた人向けの「感覚」の解説メモを置いておきます。
次の2点ふわっとした雰囲気を押さえてあげれば、PRMLなどを読み始めてもある程度耐えられると思います。
ツールとしての確率分布の読み方
右も左も分からなかった頃、中学から使っていた関数
特に式の引数と返り値について補足すると、関数は「引数(y is equal to f of x の
上の例を似た扱いになるよう書き換えると、以下のように書ける。
よく見る形としては、正規分布
あながちこの話はいい加減ではなく、ベイズ的な回帰を考える時、予測分布は
頻度論とベイズでの母数の考え方
母数
本来は母集団の持つ統計量、つまり(頻度論的には)観測したデータを生成しているはずの真の分布の持つパラメータのこと。
頻度論的な統計とベイズ統計との根本的な考え方の違いは次のところが大きい。
- 頻度論: データは確率変数で、その背後にある真の分布(仮説)からサンプルされて現れたものと考える
- ベイズ統計: 得られている定まったデータに対して、最も合う分布(信念)を確率変数として考える
言い換えると、頻度論は「母数は不変で、データは確率的に変わりうる」という考えで、ベイズ統計は「観測したデータは不変で、母数はデータに条件付けられた確率変数として存在する」という考えといえる。
どちらが誤っているという訳では無いが、正直なところ頻度論の統計は初心者が雑に扱うのは難しすぎる気がする... 仮説検定などは特にロジックが錯綜していて、国語力がないと「自分の足を撃つ」手法がベイズのそれより多いと思う。あと単純によくわからない。
# この記事で使うライブラリ
import numpy as np
from scipy import stats
import matplotlib.pyplot as plt
import seaborn as sns
import pymc as pm
import arviz as az
import xarray as xr
az.style.use("arviz-darkgrid")
信念と単純な例題
ここではベイズ推論に関する初歩的な解説と、PyMCで実装した簡単な例を見ることで、ベイズ統計がどんなふうに動くのか確認してみる。
確率は信念(belief)を数値的に表現するいい方法になる。
A,B,C が重複可能な事象、たとえば「おばばが小豆入りの餅を焼く」「室温が20度以上である」「正月である」という事象に対し、信念関数
-
b(A) > b(B) -
b(A|C) > b(B|C) -
b(A|B) > b(A|C)
この信念を表すためには以下の公理を満たしてほしいという要求がある。
信念の公理
-
b(\texttt{not} C | C) ≤ b(A|C) ≤ b(C|C) b(\texttt{not} C | C) b(C|C) -
b(A \texttt{or} B | C) ≥ \max(b(A|C) , b(B|C)) -
b(A \texttt{and} B | C) b(B|C) (A | B \texttt{and} C) - Cが真の時のBを判断する。
- Bが真ならば、BとCが真の時のAを判断する。
確率の公理
0 = p(\texttt{not} C | C) ≤ p(A|C) ≤ p(C|C) = 1 A \cap B = ∅ \implies p(A \cup B|C) = p(A|C) + p(B|C) p(A \cap B | C) = p(B|C) p(A|B \cap C)
上を見ればなんとなくそんな感じがすると思うが、確率の公理は信念の公理を満たしていて、ベイズ推論では信念を確率として表現する妥当性がわかると思う。
ベイズ推論
ベイズ推論(Bayesian inference)は集団の特性を表現するパラメータ
BAwPの言い方を借りれば、
以下では、標本や信念に関して考えて、ベイズの学習の過程を見ていく。
-
\mathcal{Y} \{ y_n \} -
\mathcal{Θ}
-
y, θ \mathcal{Y}, \mathcal{Θ}
-
p(θ) θ \in \mathcal{Θ} -
p(y|θ) θ y θ
- データを観測して
y
-
p(θ|y) y θ
上の話は次の式で表せる。分母の周辺尤度(エビデンス)は規格化定数なのでツールを使う上ではあんまり気にしなくて良い。
ちなみに尤度原理という考えがあり、「データは全て尤度に入っている必要がある」というもので、今回の例でも式を辿ればデータ
複雑な統計の問題に頻度論的な推論方法は、往々にして自明でない。しかしベイズルールでは同じような感じで推論を構成できて、性能比較に頻度論的な方法を併用することもできる。便利で上手くいく。
稀な事象の発生確率を推定する
FCBSM 1.2.1 で例示されている問題をPyMCで実装して、実際に見てみる。
ある町の感染症の有病率について、20人の町民に対して調査を行った。
関心があるのは町内の感染者の割合
まず、尤度と事前分布を決める。
尤度に関して、全体
そして、
ここで、尤度として2項分布を定めたことで、事前分布としてβ分布(beta distriburion)を定めると便利になる。というのも、尤度である2項分布とβ分布の積は、β分布に戻ってくれて、その分布は
β分布に従うθは期待値
# まずモデリングに使った分布を可視化してみる
fig, axes = plt.subplots(1, 2, figsize=(10,5))
N = 20
# fig Bin(x|n,p)
p_list = [0.1, 0.2, 0.3, 0.5, 0.8]
x = np.arange(N)
for p in p_list:
y = stats.binom(n=N, p=p).pmf(x)
axes[0].plot(x, y, "-o", label=f"p = {p}")
axes[0].legend()
axes[0].set_xticks([0,5,10,15,20], [0,5,10,15,20])
# fig Beta(x|a,b)
a_list = [0.2, 1, 2, 2, 1]
b_list = [0.5, 1, 1, 3, 3]
x = np.linspace(0, 1, 20)
for a, b in zip(a_list, b_list):
y = stats.beta(a=a, b=b).pdf(x)
axes[1].plot(x, y, label=f"a = {a}, b = {b}")
axes[1].legend()

以上のモデリングが終われば、後はデータを入手してベイズ推論を計算するだけで、この町の有病率に対する信念がどれだけ合っていたか、データを見た後どのような信念に変わったかを確認することができる。今回は町内に感染者がいなかった(
PyMCではpm.Model内に分布を登録してサンプリングによる近似で周辺尤度などをいい感じに計算して事後分布を出してくれる。
事前分布とobservedでデータを観測する分布(つまり尤度)を与えて、最後にpm.sampleで「推論ボタン」を押せば勝手に計算してくれる。(といいつつ今回のモデルは手計算で事後分布が求まるのであまり有効ではない)
# 観測データ
N = 20
y = np.zeros(N).sum() # 感染者数
alpha = 2
beta = 20
# PyMCによる計算
with pm.Model() as model:
theta = pm.Beta("theta", alpha=alpha, beta=beta)
y_pred = pm.Binomial("y_pred", n=N, p=theta, observed=y)
trace = pm.sample(2000, chains=4, cores=1)
trace = trace.sel(draw=slice(200, None)) # burn-in
az.plot_trace(trace) # 事後分布の可視化
az.plot_autocorr(trace, combined=False) # サンプリング時の自己相関
az.summary(trace) # 計算結果の要約
# mean sd hdi_3% hdi_97% mcse_mean mcse_sd ess_bulk ess_tail r_hat
# theta 0.048 0.033 0.002 0.108 0.001 0.0 2254.0 2296.0 1.0

計算結果はtraceに格納されていて、pm.sampleで渡したサンプリング回数drawsと試行回数chainsの分だけデータが入っている。いくつか図を表示したが、これらは計算結果を確認するのに役立つ。
-
az.plot_trace: サンプリング結果のKDEと、サンプリング過程で得られた素のサンプル値。サンプル値は変に規則性がない方が良い。 -
az.plot_autocorr: サンプリング過程の各値の自己相関。自己相関が高いのは良くない。 -
az.summary: 計算結果の要約を出してくれる。r_hatはGelman-Rubinテストの値で、1.1未満ならいい感じに計算が収束している。
PyMCではデータの受け渡しにxarrayという名前付きのNumPy配列を扱う。
今回のtraceの出力は主に「Data variables」、「Coordinates(chain, draw)」の軸(次元)があり、InferenceData.posterior["parameter"]のようにアクセスできる。
> trace
Inference data with groups:
> posterior
> sample_stats
> observed_data
> trace.posterior
<xarray.Dataset>
Dimensions: (chain: 4, draw: 1800)
Coordinates:
* chain (chain) int64 0 1 2 3
* draw (draw) int64 200 201 202 203 204 205 ... 1995 1996 1997 1998 1999
Data variables:
theta (chain, draw) float64 0.03886 0.0366 0.04764 ... 0.04298 0.0689
Attributes:
created_at: 2024-01-21T08:16:52.272755
arviz_version: 0.13.0
inference_library: pymc
inference_library_version: 5.9.0
sampling_time: 1.9531362056732178
tuning_steps: 1000
> trace.posterior["theta"]
<xarray.DataArray 'theta' (chain: 4, draw: 1800)>
array([[0.03886133, 0.03659619, 0.04764218, ..., 0.03289611, 0.01826896,
...
0.00704132, 0.00761786, 0.01328384, ..., 0.06280299, 0.0429803]])
Coordinates:
* chain (chain) int64 0 1 2 3
* draw (draw) int64 200 201 202 203 204 205 ... 1995 1996 1997 1998 1999
Burn-in: 可視化ライブラリArvizのtutorialのように、traceの最初の方のサンプル結果を消すことがよくある。これはplot_autocorrの図をみれば分かる通り、最初の方は自己相関が高いのでうまい結果ではないからである。今回の例では最初の200サンプルを削っているので、上のdrawが200から始まっているのがわかる。
ちなみに、InferenceData.sel(draw=slice(100, 200))のようにすれば、drawの軸を100以前と200以降を捨てることを意味する。
結果をもう少しよく見てみる。
最高密度区間(Highest Density Interval; HDI): よく使われるのは95%HDIや98%HDI。その区間にパラメータが存在する確率。ベイズの信用区間(credible interval)を意味する。
実質同値域(region of partical equivalence; ROPE): 予め定めておく仮説区間。ROPEとHDIの重複ぐあいで「仮説が妥当か、そうでないか」を論じることができる。
今回の問題では、他の町での有病率は0.10で、データを取った町でも大体0.08から0.12くらいの間になるだろうと考えられた。以下の右の図は事前分布(水色)とサンプリングで計算した事後分布(青)、その事後分布をβ分布で引き直した補助線(赤)を示していて、ROPEは[0.09, 0.11]の区間を示しており、オレンジ色の直線が0.10のラインである。
fig, axes = plt.subplots(1, 2, figsize=(15,5))
# chainごとの最高密度区間を表示
az.plot_forest(trace, var_names=["theta"], ax=axes[0])
# 事前分布(cyan)と事後分布(blue)、HDI、ROPEを表示
az.plot_posterior(trace, var_names=["theta"], rope=[0.09, 0.11], ref_val=0.10, ax=axes[1])
t = np.linspace(0, 0.26, 50)
axes[1].plot(t, stats.beta(a=alpha, b=beta).pdf(t), color="c")
axes[1].plot(t, stats.beta(a=2, b=40).pdf(t), color="r", alpha=0.8)

θの事後分布をみればわかる通り、事前分布よりも小さな有病率を信じるようになったことがわかる。定めたROPEはHDIを含んで入るものの少しだけはみ出しており、有病率の仮説の正誤は保留となっている。
最後に事後分布と事前分布からデータをサンプルしたらどうなるか見てみる。
spost = pm.sample_posterior_predictive(trace, model=model)
az.plot_ppc(spost, num_pp_samples=50, kind="scatter")

感度分析
事後分布を使った計算により、事前知識が事後分布にどのように影響を与えるか見ることもできる。
先程の議論で出てきた事後分布の閉形式
つまり、β分布の事後分布の平均は、データで決まる項と事前分布で決まる項に分解できて、
この式による計算を意思決定へ活かす事もできる。
例えば保険機関が市民にワクチン接種を推奨する広告を打つか打たないか判断しようと思った時、現在の感染率が0.10より大きいと強く確信しているならば広告を打てばいい。自信がない(
頻度論的な方法との比較
母集団比率
信頼区間はcorrect asymptotic frequentist covrage、つまり
また、調整Wald区間というものがよく使われるようになっていて、その式はベイズ的な味方をすれば事前分布
以上のように、ただ標本平均を取るだけではサンプル数が少ない時、母集団平均
階層モデルによるベイズ推論
前の章では頻度論的な不確実性の表現よりもベイズ推論が自然に不確実性を表現できていて、信念などの扱いで事前知識と結果の意思決定に影響を与えることができることを見てきた。
次は、前章の問題を便利に解決できる階層モデルについて見てから、複数のグループに跨るデータの分析を行う。
階層モデル
事前分布についてのパラメータを更に事前分布で束縛することで、パラメータが多いモデルもロバストな推論ができるようにする。というのも、パラメータは少ない方が解釈性や汎化性能がいい(バイアスとバリアンスの問題)が、多いほうが複雑な事象をモデリングできるので、どっちも捨てがたいという課題があり、階層モデルではこのパラメータ数からくる複雑さも最適化できるという嬉しさがある。
具体的にいうと、まずデータ
これにより事前分布の情報が不十分で多義的な解釈ができてしまう場合でも、多パラメータについてロバストに推論できるようになる。実際の応用では、ハイパラ
以下では、前章の町の有病率についての問題を階層ベイズで推論したPyMCのコードで、パラメータ
# 観測データ
N = 20
y = 8 # 感染者数 今回は 8/20人 が感染したことにする
with pm.Model() as model_nomal:
alpha = 2
beta = 20
theta = pm.Beta("theta", alpha=alpha, beta=beta)
y_pred = pm.Binomial("y_pred", n=N, p=theta, observed=y)
trace_nomal = pm.sample(2000, chains=2, cores=1)
trace_nomal = trace_nomal.sel(draw=slice(100, None))
az.plot_trace(trace_nomal)
plt.show()
az.plot_posterior(trace_nomal, var_names=["theta"])
plt.show()
with pm.Model() as model_hierarchical:
alpha = pm.HalfCauchy("alpha", beta=5)
beta = pm.HalfCauchy("beta", beta=5)
theta = pm.Beta("theta", alpha=alpha, beta=beta)
y_pred = pm.Binomial("y_pred", n=N, p=theta, observed=y)
trace_hierarchical = pm.sample(2000, chains=2, cores=1)
trace_hierarchical = trace_hierarchical.sel(draw=slice(100, None))
az.plot_trace(trace_hierarchical)
plt.show()
az.plot_posterior(trace_hierarchical, var_names=["theta", "alpha", "beta"])
plt.show()


今回の設定ではデータ上の有病率は0.40%で、最初に事前知識を強く与えたモデルでは
他にも、前章と同じ設定の0人で行った場合も階層モデルの方がデータに近い推測をしてくれる、言い換えれば設定した事前分布の効果を強く受けすぎないという便利さがある。これは良いことばかりでもないが、特に予想に自信がもてない場合は、頻度論よりは不確実性をある程度説明してくれながら、データにfitした推論をしてくれる、という点で使いやすいモデルを作れると思う。
複数グループに跨るデータ分析
階層モデルの簡単な挙動について見たところで、もっと有益な性質について見ていく。
縮小(shrinkage): 各グループのデータがハイパー事前分布を通じて情報を共有することで、共通の平均値に向かって動くこと。
以下のコードでは次の問題を扱うことで、階層モデルによる収縮を確認する。
問題として、ある都市の3つの川の汚染度が、やばい(1)か、やばくない(0)かを、1つの川の30箇所からサンプルすることで、河川の状況を調べたいというシチュエーションを考える。それぞれの川の合計汚染度をベルヌーイ分布(2項分布の各試行の2値版)で尤度をモデリングし、β分布による各川の汚染度を推測する。
以下のコードでは、3つの川の汚染サンプルから総合的な汚染度%を推定するものである。
def make_data(g_sample, n_sample):
n = len(n_sample)
groupe_idx = np.repeat(np.arange(n), n_sample)
data = []
for i in range(3):
data.extend(np.repeat([1,0], [g_sample[i],n_sample[i]-g_sample[i]]))
return data, groupe_idx # データ数はn_sampleの合計、データ内容はg_sample個の汚染と(n_sample-g_sample)個の非汚染
def infer(data, groupe_idx):
with pm.Model() as model:
alpha = pm.HalfCauchy("alpha", beta=5)
beta = pm.HalfCauchy("beta", beta=5)
theta = pm.Beta("theta", alpha=alpha, beta=beta, shape=3)
y_pred = pm.Bernoulli("y_pred", p=theta[groupe_idx], observed=data)
trace = pm.sample(2000, chains=2, cores=1, progressbar=False)
trace = trace.sel(draw=slice(100, None))
az.plot_posterior(trace, var_names=["theta"])
plt.show()
return trace
n_sample = [30, 30, 30] # データに含まれる、グループ毎のサンプル数
g_sample = [18, 18, 18] # グループに含まれる、汚染がやばいサンプルの個数
n = len(n_sample)
data, groupe_idx = make_data(g_sample, n_sample)
# groupe_idx = [
# 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0
# 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1
# 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2
# ]
# data = [
# 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 0 0 0 0 0 0 0 0 0 0 0 0
# 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 0 0 0 0 0 0 0 0 0 0 0 0
# 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 0 0 0 0 0 0 0 0 0 0 0 0
# ]
infer(data, groupe_idx) # サンプル中の汚染 [18, 18, 18] の場合
g_sample = [3, 3, 3]
data, groupe_idx = make_data(g_sample, n_sample)
infer(data, groupe_idx) # サンプル中の汚染 [ 3, 3, 3] の場合
g_sample = [18, 3, 3]
data, groupe_idx = make_data(g_sample, n_sample)
infer(data, groupe_idx) # サンプル中の汚染 [18, 3, 3] の場合
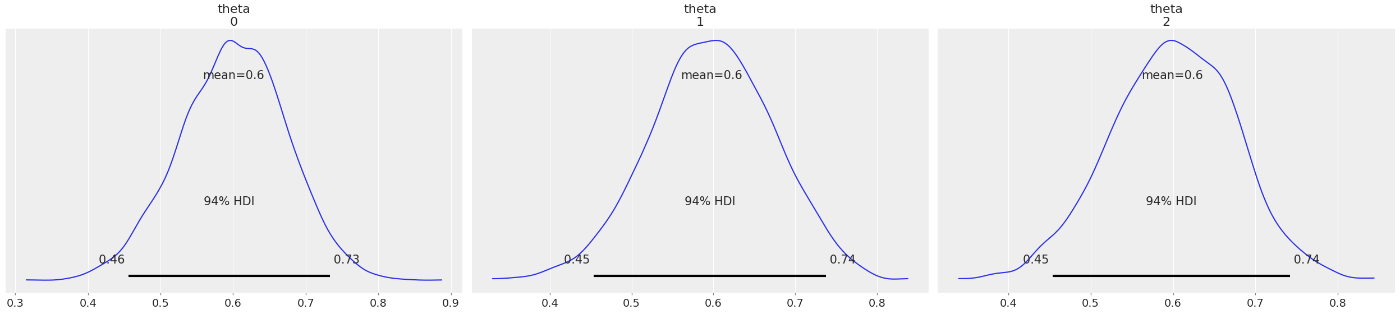


結果の通り、各川の汚染サンプルが[18, 18, 18]の場合の
収縮に関する数学的な話が見当たらなかったので深煎りしないが、この性質を使うことで、データセット内のあるグループにデータがほんの少ししかないような状況が発生した時でも、そのグループにも確からしい推測ができるようになる、つまり情報が伝播してグループ単位のロバスト性が現れる、という良さをもたらしてくれるように思う。
ロバスト推論
以上の話題が理解できて使えるようになれば、もうベイズっぽい本を読み始める準備はできたと思う。内容はかなり薄まってしまっているが、FCBSMの1章とBAwPの1~3章の内容はだいたいこんな感じの話を詳しくしてくれている。
FCBSM 1章の最後では線形回帰モデルにおけるスパース回帰などを題材に予測分布について触れられている。ここではBAwP 3.2.2のロバスト推定について少し詳しく扱って、spike-and-slab回帰の話題は文字面だけ紹介しておく。
StudentT分布による推論
StudentT分布: 正規分布の裾を重くしたような形の分布で、ガウス分布よりも外れ値に引っ張られない推論ができる。例えば上の水質調査の例で言えば、1箇所だけ水質が悪くても他が全部綺麗だったら、その1箇所は無視して推測してしまおうという挙動が起こる。PRML 2.3.7に詳しく載っている。
奇っ怪な式だが、これは正規分布とその分散の共役事前分布であるガンマ分布の階層モデルを周辺化して、あと変数をゴネゴネすると出てくる割と正統派な分布である。自由度
これを使って、正規分布との違いを見てみる。
nu = [100, 1, 0.1]
x = np.linspace(-10,10,200)
fig, axes = plt.subplots(1, 2, figsize=(10,5))
for n in nu:
x_pdf = stats.t(n).pdf(x)
axes[0].plot(x, x_pdf)
x_pdf = stats.norm(0, 1).pdf(x) # 正規分布
axes[1].plot(x, x_pdf)
x_pdf = stats.cauchy().pdf(x) # コーシー分布
axes[1].plot(x, x_pdf)

# データ作成
data = np.concatenate([stats.norm(0, 1).rvs(45), stats.norm(10, 1).rvs(5)])
# 推論 正規分布
with pm.Model() as model:
mu = pm.Normal("mu", mu=0, sigma=5)
sigma = pm.HalfNormal("sigma", sigma=5)
y_pred = pm.Normal("y_pred", mu=mu, sigma=sigma, observed=data)
trace = pm.sample(2000, chains=2, cores=1, progressbar=False)
az.plot_posterior(trace)
plt.show()
g_mu = trace.posterior["mu"].values.mean() # 推論結果の分布の期待値を点で求める
g_sigma = trace.posterior["sigma"].values.mean()
# 推論 StudentT分布
with pm.Model() as model:
mu = pm.Normal("mu", mu=0, sigma=5)
sigma = pm.HalfNormal("sigma", sigma=5)
nu = pm.Exponential("nu", lam=1/30)
y_pred = pm.StudentT("y_pred", mu=mu, sigma=sigma, nu=nu, observed=data)
trace = pm.sample(2000, chains=2, cores=1, progressbar=False)
trace = trace.sel(draw=slice(100, None))
az.plot_posterior(trace)
plt.show()
t_mu = trace.posterior["mu"].values.mean()
t_sigma = trace.posterior["sigma"].values.mean()
t_nu = trace.posterior["nu"].values.mean()
# データにfitしているか確認
fig, axes = plt.subplots(1, 2, figsize=(10,5))
sns.kdeplot(data, ax=axes[0])
axes[0].vlines(g_mu, 0, 0.2, color="m", linestyles="dotted")
axes[0].vlines(t_mu, 0, 0.2, color="c", linestyles="dotted")
x_pdf = stats.norm(g_mu, g_sigma).pdf(x)
axes[1].plot(x, x_pdf, color="m")
axes[1].vlines(g_mu, 0, 0.3, color="m", linestyles="dotted")
x_pdf = stats.t(t_nu, t_mu, t_sigma).pdf(x)
axes[1].plot(x, x_pdf, color="c")
axes[1].vlines(t_mu, 0, 0.3, color="c", linestyles="dotted")
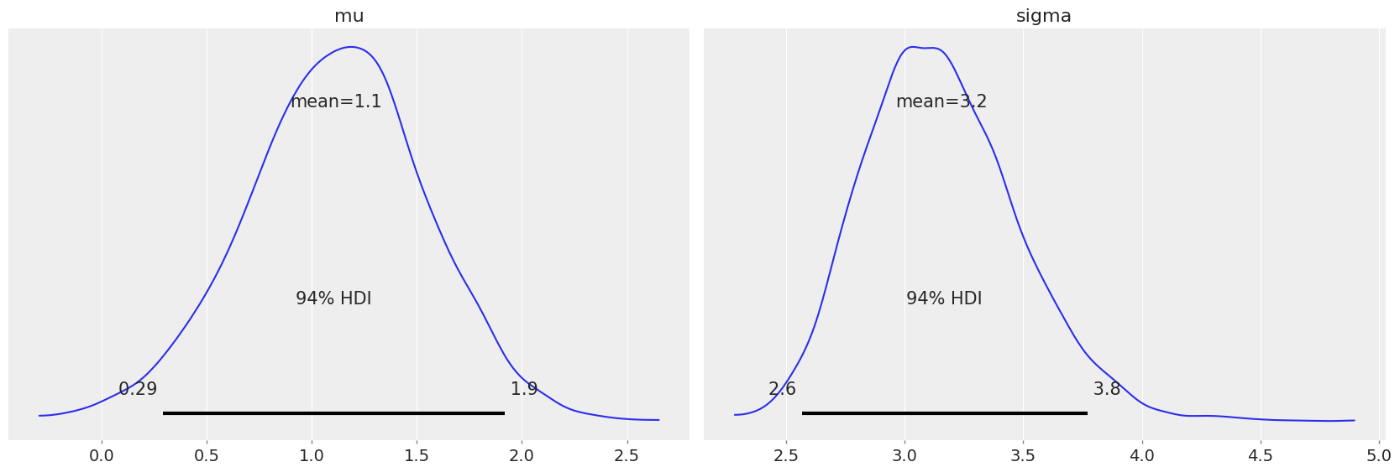


結果を見る通り、正規分布では小さなコブに引っ張られて平均
この記事を通して、推論で出た分布の平均を取ってグラフを書いているので、あまりベイズのご利益を受けていないことに注意。ベイズ的な方法は、「事前に持っている信念が、データを受けてどのように変化したか、その不確かさが確率分布として得られる」ことであり、高度なことをするようになって初めて嬉しさがわかってくるものだとも言える。
このロバスト性は、回帰などで言うRedgeやLassoなどに近い感覚で、正則化項の強さを事前分布に語らせることで変数選択を暗黙的に行っている(?)感じの計算から来ている。もとより、正規分布を事前分布として使った線形回帰は、式展開するとRedge回帰になっていることがわかり、Lassoなどもラプラス分布を事前分布にした方法として導ける(らしい)。モデル比較の文脈でも出てくるが、バイアス-バリアンス分解や前述の調整Wald区間など、ベイズ的観点からモデルを扱うことで正則化 に関する知識がたくさん得られる。
FCBSM 1.2.2 ではspike-and-slab事前分布 を使ったスパースな(つまり変数選択を正則化とした)線形モデルによる重回帰を例示している。(最初から飛ばしすぎな内容なので最初読む分には完全に理解する必要はないかもしれない...)
FCBSMの方は読み進めている途中だが、データサイエンティストで有名なTJOさんが書評を出していた。
宣伝
以下の記事ではPyMCによる一般化線形モデル、モデル比較、ガウス過程というようなベイズ初心者3点セットの勉強ノートを残している。線形回帰より後の記事はある程度正確だと思うので、勉強の順番の目次として活用してもらえたらうれしい。
Discussion