Bayesian Analysis with PyMC 勉強ノート 5 分類モデル
Osvaldo Martin 著, 金子武久 訳
Pythonによるベイズ統計モデリング (Bayesian Analysis with Python)
2018-06-26
https://www.kyoritsu-pub.co.jp/book/b10003944.html
Christopher M. Bishop 著, 元田浩 栗田多喜夫 樋口知之 松本裕治 村田昇 監訳
パターン認識と機械学習 ベイズ理論による統計的予測 (Pattern Recognition and Machine Learning)
2012-01-20
https://www.maruzen-publishing.co.jp/item/?book_no=294524
Pythonによるベイズ統計モデリングを読んだので学習メモを整理します。学習中のノートなので正確ではないかもしれません。使われているコードをPyMC3からPyMC5に書き直しました。
また、せっかくなのでPRMLの本を参照しながら読むことにしました。式の文字の扱いなどはPRML風になるよう頭に収めようと思っています。
ロジスティック回帰
次のライブラリを使用する。
import matplotlib.pyplot as plt
import numpy as np
import pandas as pd
import seaborn as sns
import pymc as pm
import arviz as az
az.style.use("arviz-darkgrid")
0. 線形回帰を題材にしたこれまでの復習
教師データ
ベイズ線形回帰を扱うと、通常の最尤推定による回帰モデルで起こる過学習を避け、モデルの複雑さを自動的に決定できる嬉しさがある。
MCMCを使える今回は代数計算による必要がないので、モデルパラメータ
PRML3.3の例では、以下の設定の問題を解いている。
- 単回帰モデル
y(x,\bold{w}) = w_0 + w_1 x -
f_{\texttt{real}}(x,\bold{a}) = a_0 + a_1 x a_0 = -0.3, a_1 = 0.5 -
x_n \sim \mathcal{U}(x|y(-1, 1) \{ x_n \} f_{\texttt{real}}(x_n,\bold{a}) -
ε \sim \mathcal{N}(ε|0, 0.2^2) t_n = x_n + ε \{ t_n \} -
\{ t_n \} \bold{a} - PRMLでは精度
β = (1/0.2)^2
N = 20
a0_real = -0.3
a1_real = 0.5
e_real = np.random.normal(0, 0.2, N) # loc: μ, scale: σ
x = np.random.uniform(-1.0, 1.0, N)
y_real = a0_real + a1_real * x
y = y_real + e_real
plt.plot(x, y_real)
plt.scatter(x, y)
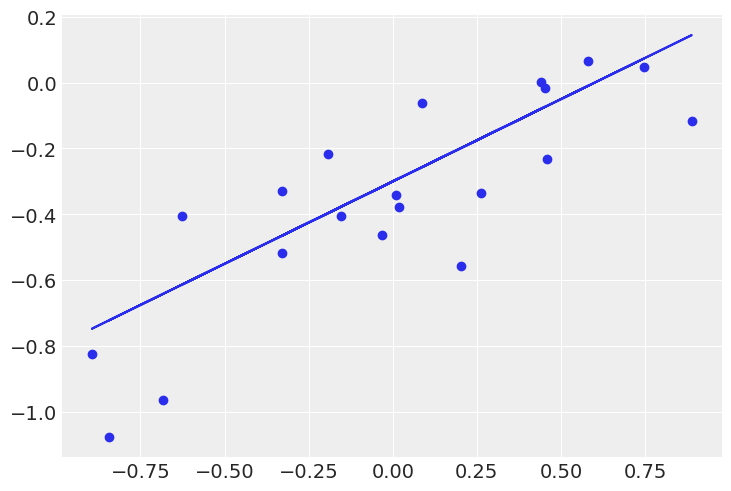
with pm.Model() as model:
w0 = pm.Normal("w0", mu=0, sigma=1)
w1 = pm.Normal("w1", mu=0, sigma=1)
e = pm.HalfCauchy("epsilon", 5)
mu = pm.Deterministic("mu", w0 + w1 * x)
y_pred = pm.Normal("y_pred", mu=mu, sigma=e, observed=y)
trace = pm.sample(1000, chains=2, cores=1)
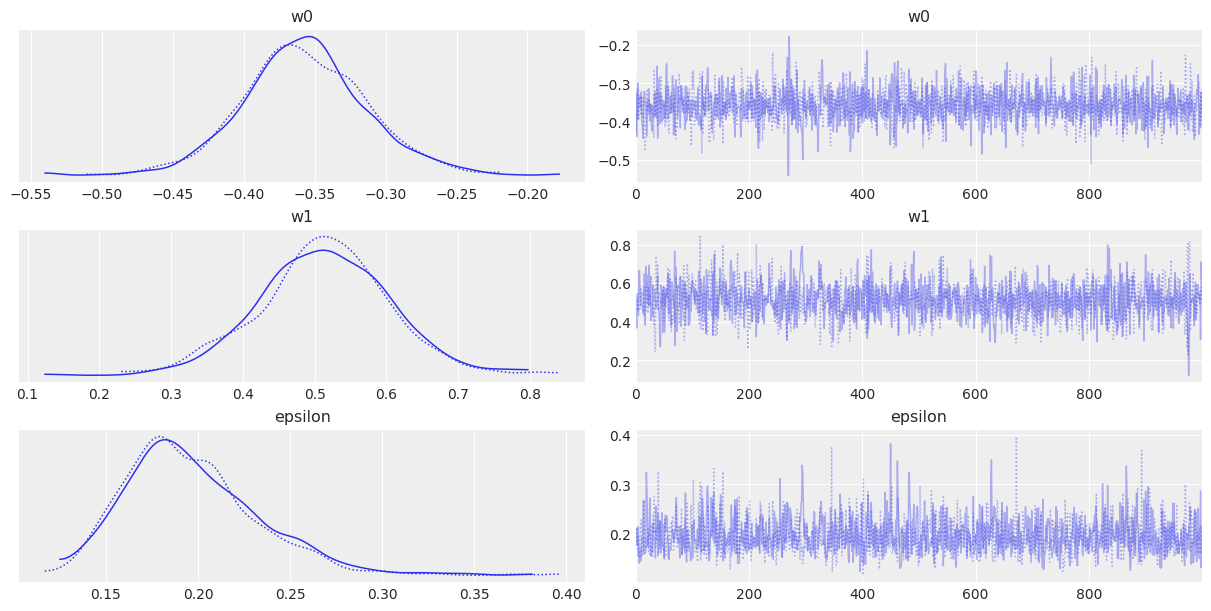
az.plot_trace(trace, var_names=["w0", "w1", "epsilon"])
az.plot_autocorr(trace, var_names=["w0", "w1", "epsilon"], combined=True)
az.summary(trace, var_names=["w0", "w1", "epsilon"])
mean sd hdi_3% hdi_97% mcse_mean mcse_sd ess_bulk ess_tail r_hat
w0 -0.357 0.043 -0.436 -0.273 0.001 0.001 2326.0 1482.0 1.0
w1 0.514 0.091 0.333 0.674 0.002 0.002 1921.0 1314.0 1.0
epsilon 0.197 0.036 0.136 0.262 0.001 0.001 1669.0 1042.0 1.0
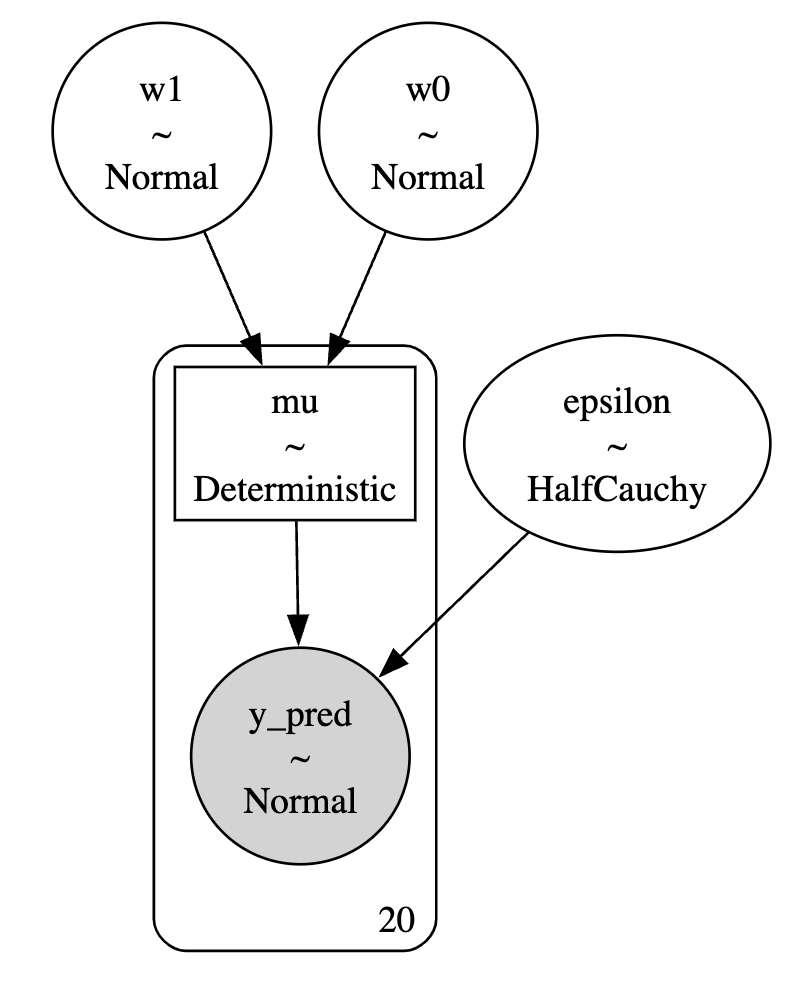
graphvizをインストールしてあると、グラフィカルモデルを可視化できる。
# brew install graphviz
# pip install graphviz
pm.model_to_graphviz(model=model)
事前分布と事後分布について確認する。
# https://www.pymc.io/projects/docs/en/latest/api/generated/pymc.sampling.forward.sample_prior_predictive.html
# Sampling: [epsilon, w0, w1, y_pred]
myprior = pm.sample_prior_predictive(samples=50, model=model)
# Inference data with groups:
# > prior
# > prior_predictive
# > observed_data
# https://www.pymc.io/projects/docs/en/latest/api/generated/pymc.sampling.forward.sample_posterior_predictive.html
# Sampling: [y_pred]
myposterior = pm.sample_posterior_predictive(trace, model=model)
# print(myposterior)
# Inference data with groups:
# > posterior_predictive
# > observed_data
sns.kdeplot(x=myprior.prior["w0"].values.reshape(-1), y=myprior.prior["w1"].values.reshape(-1), cmap=sns.light_palette("blue", as_cmap=True))
sns.kdeplot(x=trace.posterior["w0"].values.reshape(-1), y=trace.posterior["w1"].values.reshape(-1), cmap=sns.light_palette("red", as_cmap=True))
plt.xlim([-1.0, 1.0])
plt.ylim([-1.0, 1.0])
plt.show()

もともと広く適当に設定していた事前分布が、データ観測によって最適化された事後分布になったことがわかる。
予測分布
実際的にはパラメータそのものではなく新しい
ただし
- 目標変数の条件付き分布
p(t | \bold{x}, \bold{w}, β) = \mathcal{N}(t | y(\bold{x}, \bold{w}), β^{-1}) - パラメータ
\bold{w} p(\bold{w} | \bold{t}, α, β) -
α, β
MCMCで扱えば代数的に計算しなくても可視化できる。
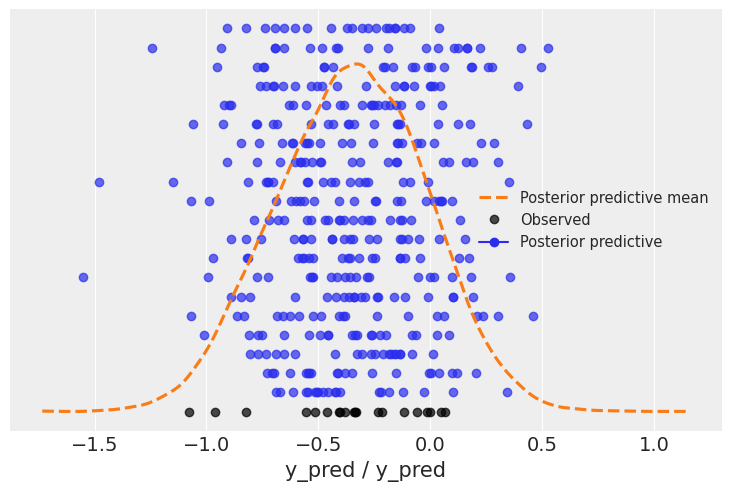
# y_predが観測データを再現できるか確認
# https://python.arviz.org/en/stable/api/generated/arviz.plot_ppc.html
az.plot_ppc(myposterior, num_pp_samples=20, kind="scatter")
plt.show()
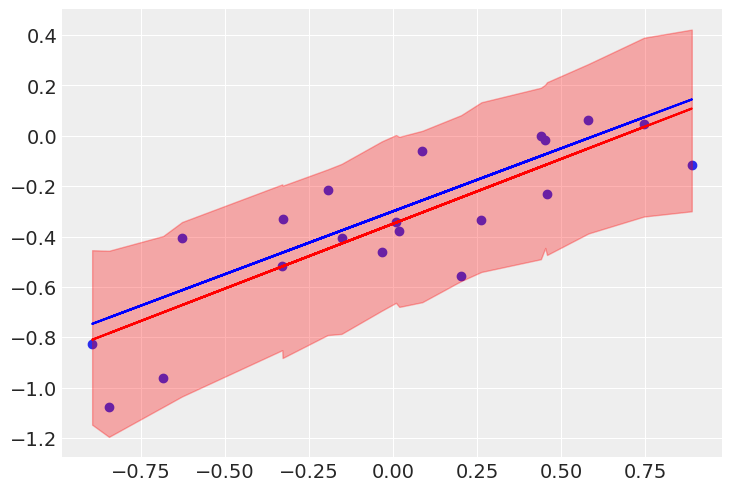
# 予測パラメータのサンプリングによるモデルの不確かさの可視化
idx = np.argsort(x)
post_w0 = trace.posterior["w0"].values.reshape(-1)
post_w1 = trace.posterior["w1"].values.reshape(-1)
sig = az.hdi(myposterior.posterior_predictive, hdi_prob=0.90)["y_pred"].values[idx] # az.hdi(trace.posterior) -> <xarray.Dataset> [4(=w0+w1+eps+mu), draw]
plt.scatter(x, y)
plt.plot(x, np.mean(post_w0[idx])+np.mean(post_w1[idx])*x, c="r") # 予測分布の期待値による回帰直線
plt.plot(x, a0_real + a1_real * x, c="b") # データ生成元の直線
plt.fill_between(x[idx], y1=sig[:,0], y2=sig[:,1], color="r", alpha=0.30) # 信頼区間
plt.show()
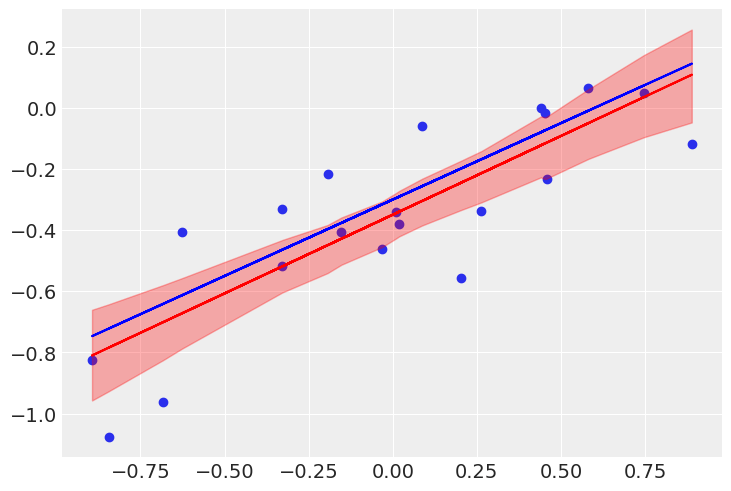
# 予測パラメータの最高密度区間(ベイズ信頼区間)によるモデルの不確かさの可視化
sig = az.hdi(trace.posterior, hdi_prob=0.90)["mu"].values[idx]
plt.scatter(x, y)
plt.plot(x, np.mean(post_w0[idx])+np.mean(post_w1[idx])*x, c="r")
plt.plot(x, a0_real + a1_real * x, c="b")
plt.fill_between(x[idx], y1=sig[:,0], y2=sig[:,1], color="r", alpha=0.30)
plt.show()
逐次推論
同じ問題設定においてデータを逐次的に投下した場合、まず事前分布に対してデータ空間を観測しパラメータの事後分布を得ることになる。ベイズ推論ではデータが増えた時に減少するパラメータの不確かさを見ることもできる。PRML p.155の図の通り、
 [1]より、逐次推論における事後分布と尤度の推移
[1]より、逐次推論における事後分布と尤度の推移
また予測分布に関しても、PRML p.157 の通りデータ観測によって定まっていく。
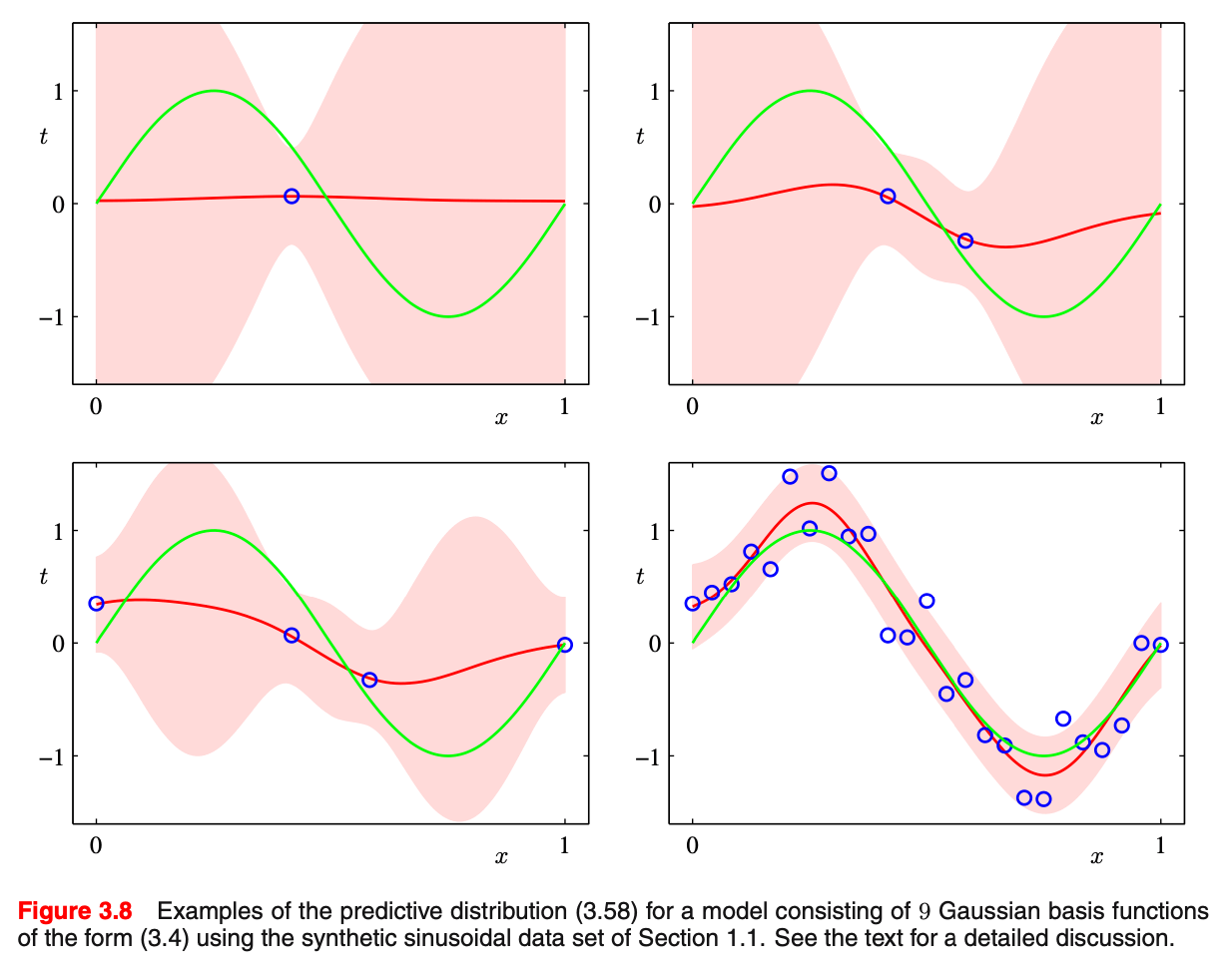 [1]より、逐次推論における予測分布の推移
[1]より、逐次推論における予測分布の推移
1. ロジスティック回帰
PRML 4.3.2
活性化関数:
連結関数: 活性化関数の逆関数。すなわち、活性化関数は逆連結関数ともいえる。
単純な線形モデルでは活性化関数が恒等射として扱っているが、一般化線形モデル (generalized linear model; GLM)の枠組みになるともはやパラメータすら線形ではない。
この活性化関数としてシグモイド(logistic sigmoid)を用いることで、GLMで分類モデルを作ることができる。
データセットとしてirisを使う。4つの独立変数と3つの種別があり、今回は簡単に1つの独立変数と2つの種別の分類について考える。
iris = sns.load_dataset("iris")
sns.pairplot(iris, diag_kind="kde", hue="species")
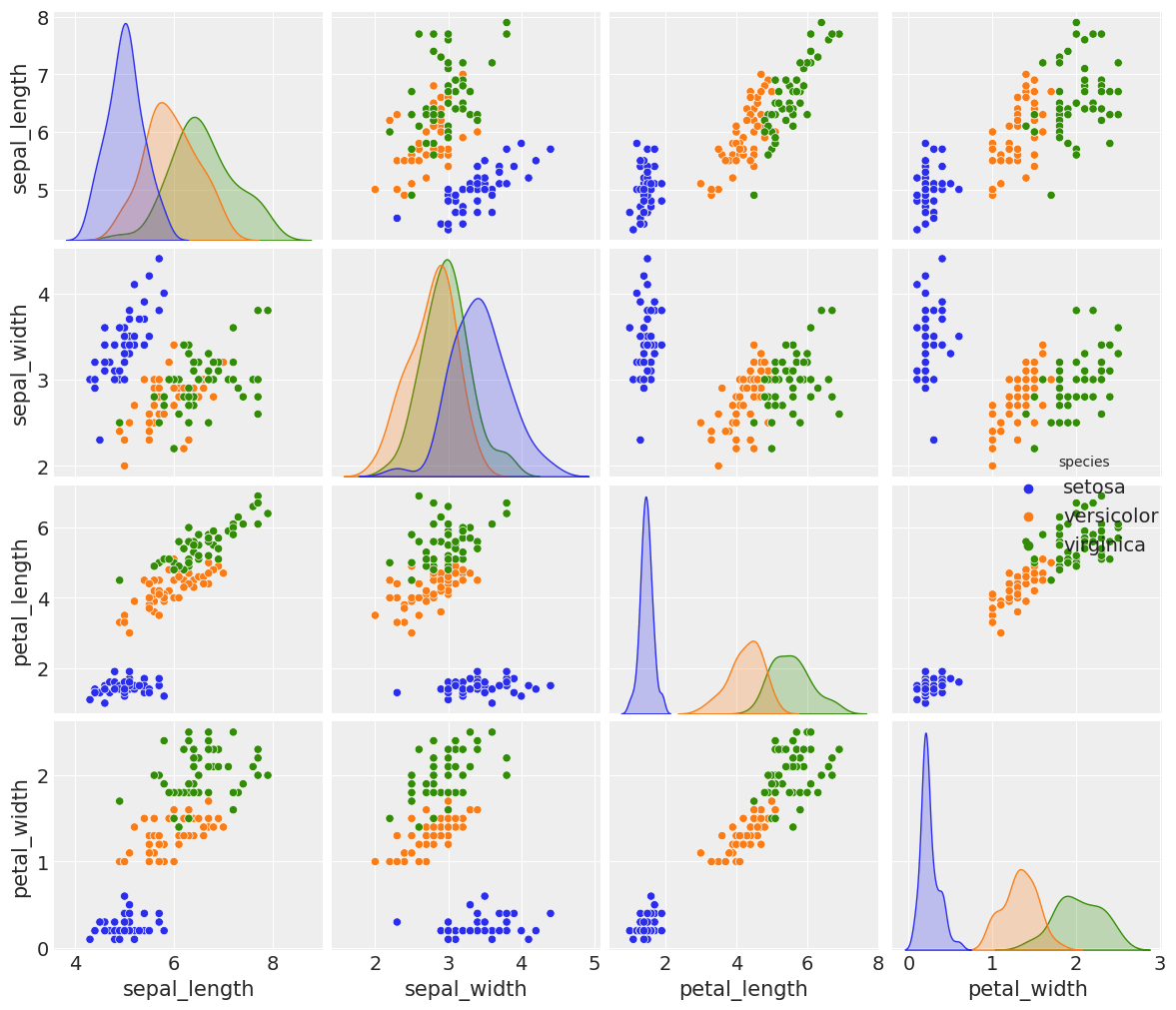
dataset = pd.concat([iris[iris["species"] == "versicolor"], iris[iris["species"] == "setosa"]])
y = pd.Categorical(dataset["species"]).codes
x = dataset["sepal_length"].values
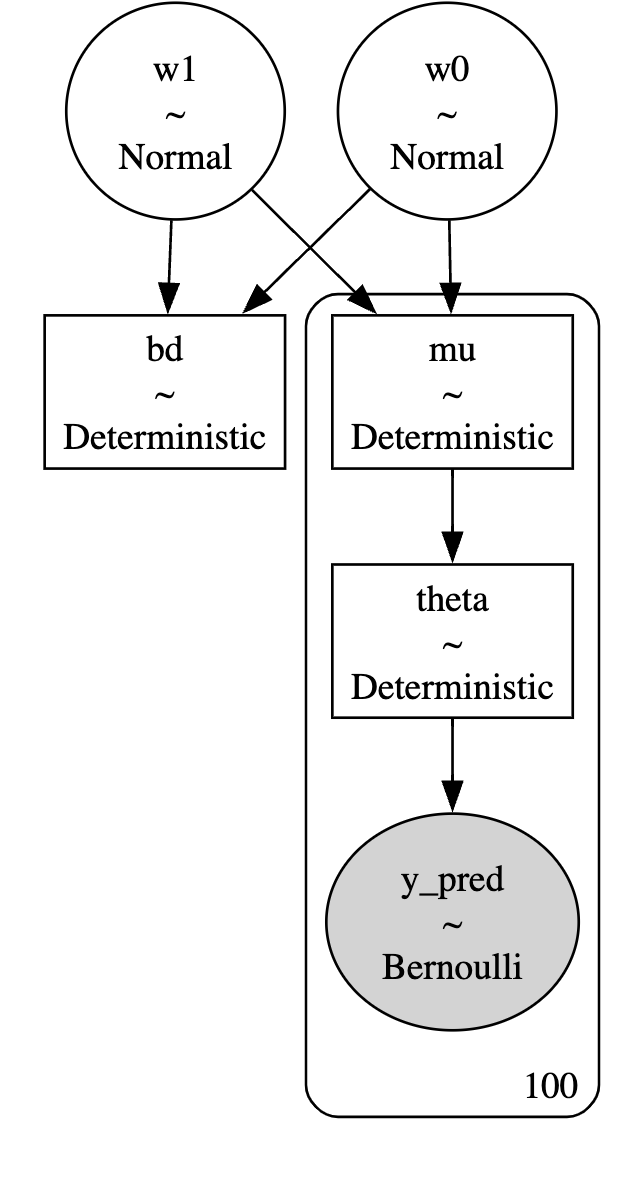
with pm.Model() as model:
w0 = pm.Normal("w0", mu=0, sigma=10)
w1 = pm.Normal("w1", mu=0, sigma=10)
mu = pm.Deterministic("mu", w0 + pm.math.dot(x, w1))
theta = pm.Deterministic("theta", 1/(1 + pm.math.exp(-mu)))
bd = pm.Deterministic("bd", -w0/w1)
y_pred = pm.Bernoulli("y_pred", p=theta, observed=y)
trace = pm.sample(1000, chains=2, cores=1)
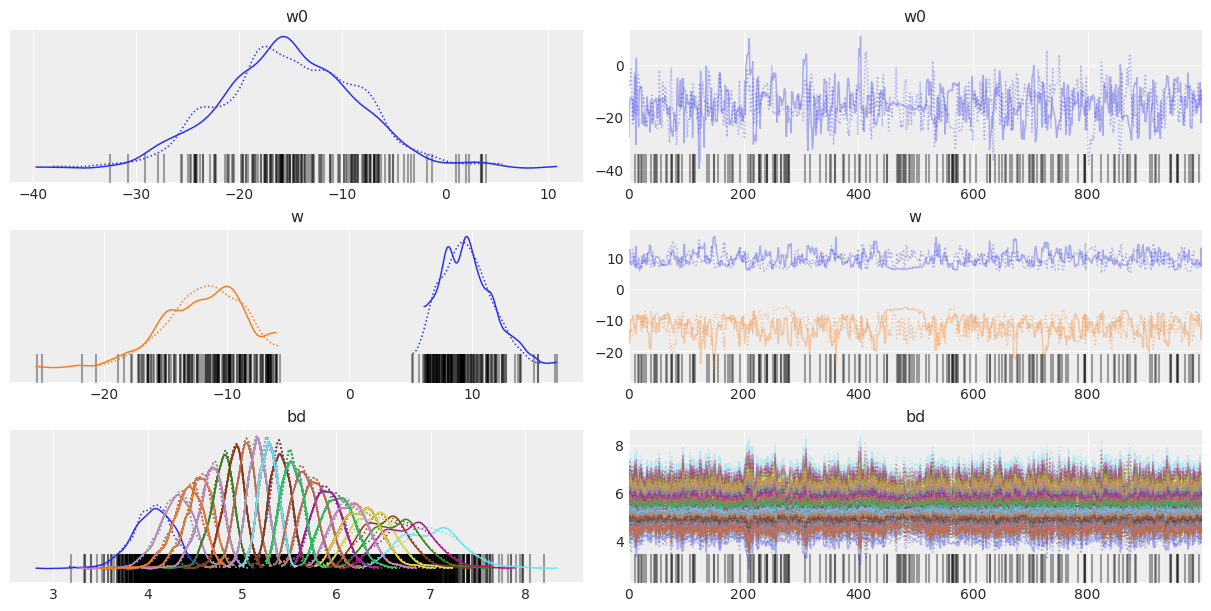
az.plot_trace(trace, var_names=["w0", "w1", "bd"])
az.plot_autocorr(trace, var_names=["w0", "w1", "bd"], combined=True)
az.summary(trace, var_names=["w0", "w1", "bd"])
pm.model_to_graphviz(model=model)
mean sd hdi_3% hdi_97% mcse_mean mcse_sd ess_bulk ess_tail r_hat
w0 -23.360 3.763 -30.394 -16.738 0.217 0.154 294.0 355.0 1.0
w1 4.314 0.698 3.073 5.614 0.040 0.028 295.0 314.0 1.0
bd 5.416 0.069 5.283 5.548 0.002 0.001 1751.0 1296.0 1.0


print(trace.posterior)
idx = np.argsort(x)
post_theta = trace.posterior["theta"].values
post_theta_hdi = az.hdi(trace.posterior, hdi_prob=0.90)["theta"].values
post_bd = trace.posterior["bd"].values
post_bd_hdi = az.hdi(trace.posterior, hdi_prob=0.90)["bd"].values
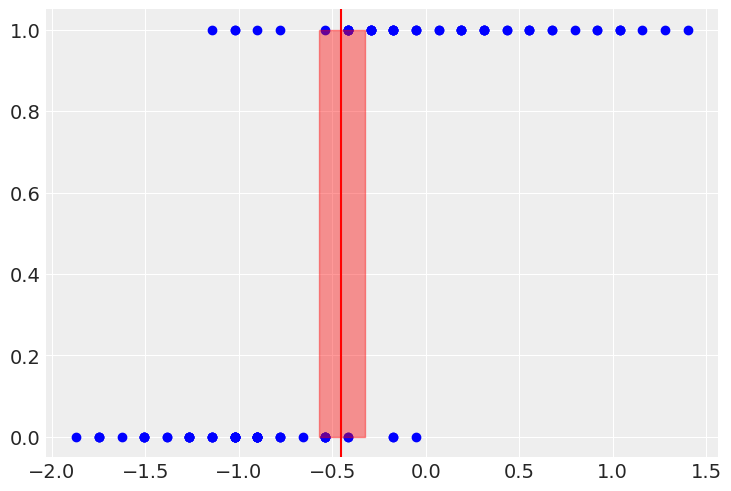
plt.scatter(x, y, color="b")
plt.plot(x[idx], post_theta.mean(axis=(0,1))[idx], color="r")
plt.fill_between(x[idx], post_theta_hdi[idx][:,0], post_theta_hdi[idx][:,1], color="r", alpha=0.4)
plt.axvline(post_bd.mean(), ymax=1, color="r")
plt.fill_betweenx(y=[0,1], x1=post_bd_hdi[0], x2=post_bd_hdi[1], color="r", alpha=0.4)
<xarray.Dataset>
Dimensions: (chain: 2, draw: 1000, mu_dim_0: 100, theta_dim_0: 100)
Coordinates:
* chain (chain) int64 0 1
* draw (draw) int64 0 1 2 3 4 5 6 7 ... 993 994 995 996 997 998 999
* mu_dim_0 (mu_dim_0) int64 0 1 2 3 4 5 6 7 8 ... 92 93 94 95 96 97 98 99
* theta_dim_0 (theta_dim_0) int64 0 1 2 3 4 5 6 7 ... 92 93 94 95 96 97 98 99
Data variables:
w0 (chain, draw) float64 -21.82 -23.99 -23.83 ... -29.68 -29.26
w1 (chain, draw) float64 4.047 4.409 4.373 ... 5.465 5.446 5.431
mu (chain, draw, mu_dim_0) float64 6.511 4.083 ... -0.477 -2.106
theta (chain, draw, theta_dim_0) float64 0.9985 0.9834 ... 0.1085
bd (chain, draw) float64 5.391 5.441 5.449 ... 5.45 5.45 5.388
Attributes:
created_at: 2024-01-06T19:34:46.653198
arviz_version: 0.13.0
inference_library: pymc
inference_library_version: 5.9.0
sampling_time: 3.5929160118103027
tuning_steps: 1000

モデルの定義より
2. 重ロジスティック回帰
独立変数の数を増やす。決定境界の議論については1変数の時と簡単に拡張することができる。
n変数の決定境界は超平面の方程式で表現でき、計算で求める事ができる。
dataset = pd.concat([iris[iris["species"] == "versicolor"], iris[iris["species"] == "setosa"]])
y = pd.Categorical(dataset["species"]).codes
x = dataset[["sepal_length", "sepal_width"]].values
with pm.Model() as model:
w0 = pm.Normal("w0", mu=0, sigma=10)
w = pm.Normal("w", mu=0, sigma=10, shape=2)
mu = pm.Deterministic("mu", w0 + pm.math.dot(x, w))
theta = pm.Deterministic("theta", 1/(1 + pm.math.exp(-mu)))
bd = pm.Deterministic("bd", - w0/w[0] - w[1]/w[0] * x[:,1])
y_pred = pm.Bernoulli("y_pred", p=theta, observed=y)
trace = pm.sample(1000, chains=2, cores=1)
az.plot_trace(trace, var_names=["w0", "w", "bd"])
az.plot_autocorr(trace, var_names=["w0", "w"], combined=True)
az.summary(trace, var_names=["w0", "w"])
mean sd hdi_3% hdi_97% mcse_mean mcse_sd ess_bulk ess_tail r_hat
w0 -15.157 6.737 -27.691 -2.919 0.311 0.220 471.0 551.0 1.00
w[0] 9.629 2.115 5.932 13.485 0.139 0.098 214.0 244.0 1.00
w[1] -11.906 3.218 -17.557 -6.210 0.219 0.155 161.0 56.0 1.01
idx = np.argsort(x[:,1])
post_bd = trace.posterior["bd"].values
post_bd_hdi = az.hdi(trace.posterior, hdi_prob=0.90)["bd"].values
print(post_bd_hdi[:,0].shape)
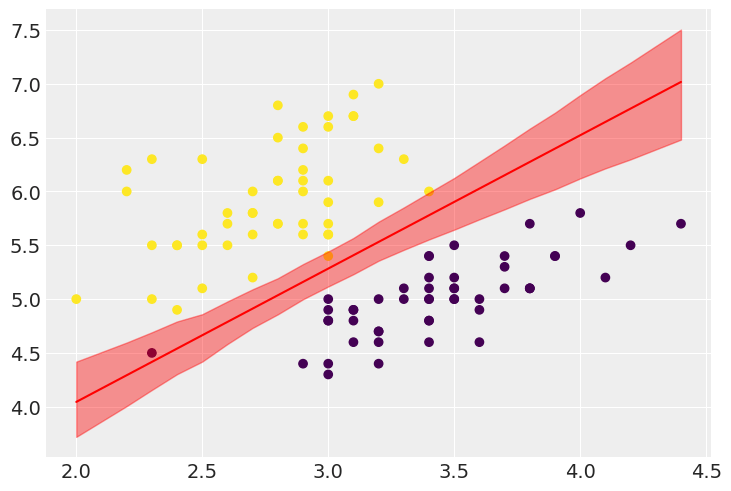
plt.scatter(x[:,1], x[:,0], c=y, cmap="viridis")
plt.plot(x[:,1][idx], post_bd.mean(axis=(0,1))[idx], color="r")
plt.fill_between(x[:,1][idx], post_bd_hdi[:,0][idx], post_bd_hdi[:,1][idx], color="r", alpha=0.4)
相関のある変数
相関が高い変数を入れる場合、変数にモデルへの成約を与えるパワーが少なくなる。これをなんとかするため、事前分布に強い情報を与えることである。Andrew GelmanとStanチームは
# irisデータの共分散行列を可視化し、各変数の相関を見る
corr = iris[iris["species"] != "virginica"].corr()
mask = np.tri(*corr.shape).T
sns.heatmap(corr.abs(), mask=mask, annot=True, cmap="viridis")

ロジスティック回帰係数の解釈
シグモイド活性化関数は非線形なので、回帰係数を解釈する場合は注意する必要がある。
この逆関数であるロジット関数
対数オッズ: 特にこの真数部分はオッズ として知られ、係数
ソフトマックス回帰
複数クラスの分類に拡張する。
irisデータセットを全て使って、複数独立変数から複数クラス分類を行う。
y = pd.Categorical(iris["species"]).codes
x = iris[["sepal_length", "sepal_width", "petal_length", "petal_width"]].values
x = (x - x.mean(axis=0))/x.std(axis=0)
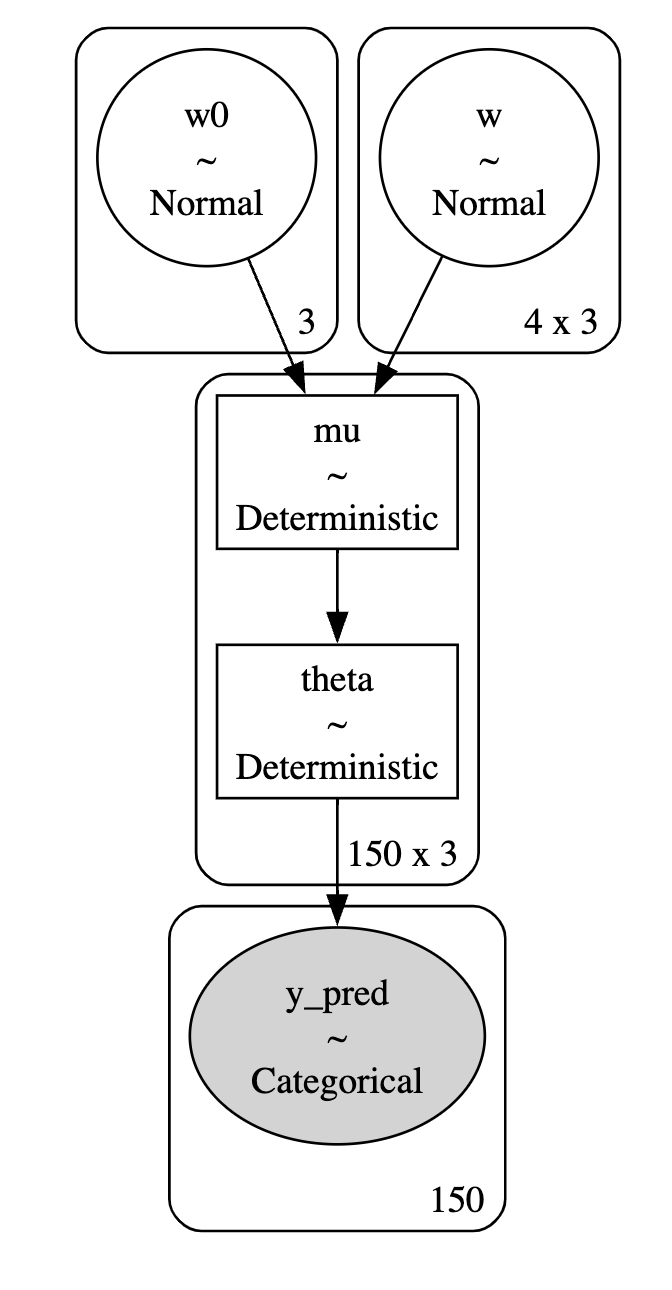
with pm.Model() as model:
w0 = pm.Normal("w0", mu=0, sigma=10, shape=3)
w = pm.Normal("w", mu=0, sigma=10, shape=(4,3))
mu = pm.Deterministic("mu", w0 + pm.math.dot(x, w))
theta = pm.Deterministic("theta", pm.math.softmax(mu, axis=1)) # axis設定しないとダメになるので注意
y_pred = pm.Categorical("y_pred", p=theta, observed=y)
trace = pm.sample(1000, chains=2, cores=1)
az.plot_trace(trace, var_names=["w0", "w"])
az.summary(trace, var_names=["w0", "w"])
pm.model_to_graphviz(model=model)

post_w0 = trace.posterior["w0"].values
post_w = trace.posterior["w"].values
pred_list = post_w0.mean(axis=(0,1)) + np.dot(x, post_w.mean(axis=(0,1)))
pred = []
for p in pred_list:
pred.append(np.exp(p)/np.sum(np.exp(p), axis=0))
acc = np.sum(y == np.argmax(pred, axis=1))/len(y)
print(acc)
0.98
98%の精度で分類できている。
3. 識別モデルと生成モデル
PRML 4.2 4.3
識別モデル(discriminative model): 今までは当該クラス
生成モデル(generative model): 最初にクラスの事前分布
線形判別分析(linear discriminant analysis; LDA)は正規分布の各
以下ではLDAによりirisが生成される
y = pd.Categorical(iris["species"]).codes
x = iris[["sepal_length", "sepal_width", "petal_length", "petal_width"]].values
x = (x - x.mean(axis=0))/x.std(axis=0)
with pm.Model() as model:
mu = pm.Normal("mu", mu=0, sigma=10, shape=(3, 4))
sigma = pm.HalfCauchy("sigma", 5, shape=(3, 4))
c00 = pm.Normal("c00", mu=mu[0,0], sigma=sigma[0,0], observed=x[0:50, 0])
c01 = pm.Normal("c01", mu=mu[0,1], sigma=sigma[0,1], observed=x[0:50, 1])
c02 = pm.Normal("c02", mu=mu[0,2], sigma=sigma[0,2], observed=x[0:50, 2])
c03 = pm.Normal("c03", mu=mu[0,3], sigma=sigma[0,3], observed=x[0:50, 3])
c10 = pm.Normal("c10", mu=mu[1,0], sigma=sigma[1,0], observed=x[50:100, 0])
c11 = pm.Normal("c11", mu=mu[1,1], sigma=sigma[1,1], observed=x[50:100, 1])
c12 = pm.Normal("c12", mu=mu[1,2], sigma=sigma[1,2], observed=x[50:100, 2])
c13 = pm.Normal("c13", mu=mu[1,3], sigma=sigma[1,3], observed=x[50:100, 3])
c20 = pm.Normal("c20", mu=mu[2,0], sigma=sigma[2,0], observed=x[100:150, 0])
c21 = pm.Normal("c21", mu=mu[2,1], sigma=sigma[2,1], observed=x[100:150, 1])
c22 = pm.Normal("c22", mu=mu[2,2], sigma=sigma[2,2], observed=x[100:150, 2])
c23 = pm.Normal("c23", mu=mu[2,3], sigma=sigma[2,3], observed=x[100:150, 3])
trace = pm.sample(1000, chains=2, cores=1)
az.plot_trace(trace, var_names=["mu", "sigma"])
az.summary(trace)
pm.model_to_graphviz(model=model)
# データ生成が実データと合っているか確認
myposterior = pm.sample_posterior_predictive(trace, model=model)
az.plot_ppc(myposterior, num_pp_samples=20, kind="scatter")
plt.show()
# c00(C="setosa", x="sepal_length")とc10(C="versicolor", x="sepal_length")の境界を見てみる
post_mu00 = trace.posterior["mu"].values[:,:,0,0] # shape [chain, sample, class, feat]
post_mu10 = trace.posterior["mu"].values[:,:,1,0]
post_mu00_hdi = az.hdi(trace.posterior, hdi_prob=0.90)["mu"].values[0,0] # shape [class, feat, 2(=top+bottom)]
post_mu10_hdi = az.hdi(trace.posterior, hdi_prob=0.90)["mu"].values[1,0]
plt.scatter(x[0:100, 0], y[0:100], color="b")
plt.axvline((post_mu00.mean()+post_mu10.mean())/2, ymax=1, color="r")
plt.fill_betweenx(y=[0,1], x1=(post_mu00_hdi[0]+post_mu10_hdi[0])/2, x2=(post_mu00_hdi[1]+post_mu10_hdi[1])/2, color="r", alpha=0.4)
plt.show()

自作のモデルは共通の分散を仮定するLDAというよりは、2次線形識別器(quadratic linear discriminant analysis; QDA)になっている気がする。この場合は決定境界は線形ではなく2次曲線になる。
一般的には分析に使うデータが正規分布に従う場合はLDAやQDAが良いが、そもそもデータが正規分布に従ってない場合はロジスティック回帰のほうが良い予測を与える。識別モデルはモデルに組み込む母数の情報がある場合に、簡単に自然に事前分布にそれを与えられることである(?)。
LDAやQDAの決定境界は閉じていて、分布の期待値を求めるだけで決定境界を得られる。ベイズでは代数計算など難しい制約なしに、例えばツールとしてT分布を使って決定境界を求めるなど、閉形式の解を求められなくても計算ができてしまう良さがある。
感想
今回はPRMLのガウス分布を用いたときの代数計算による結果を振り返りながらPyMCによるサンプリング計算ベースのプログラムで実演するという形で勉強した。
今まで確認不足や不慣れもあって、勉強ノート1~4では微妙に間違った言葉などを書いていたので時間があったら直しておきたい...
ただ、線形回帰から振り返ってある程度手に馴染んできた感じはあるので、何らかのデータで実際に遊んでみようと思う。
Discussion