RとPythonの決定木、同じにできる?
📌はじめに
もともとRで購買デモグラフィック(購買層の属性)を決定木で分析していましたが、
Pythonでも同じ結果が得られるか気になり、今回比較してみることにしました。
📌サンプルデータ(sample.csv)
以下のようなデータを使って分析を行います。

上記のデータを使って、顧客属性から購入した車のメーカーを予測する決定木モデルを作成します。
| カラム名 | 説明 | 種類 |
|---|---|---|
| ID | 顧客ごとのユニークな識別子 | 除外 |
| family | 一緒に住んでいる家族の人数 | 説明変数 |
| age | 年齢(整数) | 説明変数 |
| gender | 1 → 男性、2 → 女性 | 説明変数 |
| income | 世帯年収 | 説明変数 |
| car | 購入した車のメーカー名(目的変数) | 目的変数 |
| cluster | クラスタリングのグループ番号 | 除外 |
📌フォルダ構成
├─ 1_flow/
│ │─ run_decision_tree.py # Python 実行スクリプト
│ └─ run_decision_tree.R # R 実行スクリプト
├─ 2_data/
│ └─ sample.csv # サンプルデータ
├─ 3_output/ # 決定木出力画像用(自動作成)
📌環境
- Python 3.x
- R (RGui)
📌Python コード解説(run_decision_tree.py)
リーケージ注意報! AIモデルは思わぬ情報を使っちゃうことがあるよ
#============================================
# 0. ライブラリと変数設定
#============================================
import os
import sys
import subprocess
import pandas as pd
from sklearn.model_selection import train_test_split
from sklearn.tree import DecisionTreeClassifier, plot_tree
import matplotlib.pyplot as plt
import japanize_matplotlib # matplotlibで日本語ラベルを扱うためのライブラリ
# ディレクトリ・ファイル設定
INPUT_FOLDER = '2_data'
INPUT_FILE = 'sample.csv'
OUTPUT_FOLDER = '3_output'
save_name = 'decision_tree.png' # 比率あり
non_save_name = 'decision_tree_N.png' # 比率なし
# 除外カラム設定
ID = 'ID'
cluster = 'cluster'
#目的変数
target_col = 'car'
#============================================
# 1. パス設定とCSV読み込み
#============================================
current_path = os.getcwd()
parent_path = os.path.dirname(current_path)
input_path = os.path.join(parent_path, INPUT_FOLDER, INPUT_FILE)
output_path = os.path.join(parent_path, OUTPUT_FOLDER)
os.makedirs(output_path, exist_ok=True) # フォルダがなければ作成
save_path = os.path.join(output_path, save_name)
Non_save_path = os.path.join(output_path, non_save_name)
# CSV読み込み(文字コード自動判定)
try:
df = pd.read_csv(input_path, encoding="utf-8")
except UnicodeDecodeError:
df = pd.read_csv(input_path, encoding="cp932")
0. ライブラリと変数設定
-
pandas: データ操作用 -
scikit-learn: 決定木モデル作成と学習用 -
matplotlib+japanize_matplotlib: 可視化用、日本語ラベル対応 - 分析で使わないカラム(
IDやcluster)は除外しています - 目的変数指定:
car
1. パス設定とCSV読み込み
- 出力用フォルダが存在しない場合は自動作成
-
try:UTF-8で読み込みを試み、失敗した場合はCP932(Shift-JIS互換)で再読み込み。
環境やファイルごとの文字コードの違いにで応できます。
#============================================
# 3. 説明変数と目的変数に分ける
#============================================
X_df = df.drop([ID, cluster, target_col], axis=1) # cluster列は事前に作成されていると仮定
y_df = df[target_col] # 目的変数(car)
#============================================
# 4. 訓練・テストデータに分割
#============================================
X_train, X_test, y_train, y_test = train_test_split(X_df, y_df, random_state=0)
print(f"学習データ数: {len(X_train)} / テストデータ数: {len(X_test)}")
#============================================
# 5. 決定木モデル構築
#============================================
clf = DecisionTreeClassifier(criterion='gini', max_depth=3, random_state=0)
model = clf.fit(X_train, y_train)
print(
f"正解率(train): {model.score(X_train, y_train):.3f} / "
f"正解率(test): {model.score(X_test, y_test):.3f}"
)

正解率について
「うっ、精度低い!」と思うかもしれませんが、今回は簡易的なサンプルデータを使っています。
目的変数(car)と説明変数(age・genderなど)の間に明確なパターンがないため、予測が難しいデータです。
本番の分析では、より情報量が多く、目的変数との関連が強い特徴量を使うことで、
精度の高いモデル構築が可能になります 👍
3. 説明変数と目的変数に分ける
-
X_df: 説明変数。予測に使う入力データ。
使用しない列(IDやクラスタ列など)や目的変数を除き、残りの列を説明変数として使用します。 -
y_df: 目的変数。予測したい結果(car)
4. 訓練・テストデータに分割
-
X_train, X_test, y_train, y_test = train_test_split(X_df, y_df, random_state=0):
モデルがどのくらい「予測できるか」を確認するため、データを 訓練用(train) と テスト用(test) に分割。
❓訓練用データとテスト用データ❓
訓練用データとテスト用データとは❓
-
訓練用データ(X_train, y_train)
モデルを作るために使うデータ。モデルが「学習」する材料です。 -
テスト用データ(X_test, y_test)
学習には使わず、モデルの予測性能を検証するために使います。未知データに近い役割です。
💡 全データ数が100件の場合
- 訓練データ数 → 100 − 25 = 75
- テストデータ数 → 100 × 0.25 = 25
正解率とは❓
-
訓練データ(train)の正解率
モデルを学習させたデータに対して、どれだけ正しく予測できたかを示す指標です。
高すぎる場合は 過学習(覚えすぎてしまう状態) の可能性があります。 -
テストデータ(test)の正解率
学習に使っていないデータに対して、どれだけ正しく予測できるかを示す指標です。
こちらが高ければ、未知のデータに対しても性能が良いモデルといえます。
💡 例えると
- 訓練データは「教科書」
- テストデータは「試験」
教科書を丸暗記しても、試験で応用できなければ意味がありません。
モデルも同様で、テストデータでの正解率を確認することで、本当に役立つモデルかを判断できます。
※ 評価の際はデータを訓練用・テスト用に分割しますが、モデル評価後は全データで再学習して最終的な決定木を作ります。
5. 決定木モデル構築
-
criterion='gini': 不純度の計算方法(ジニ係数) -
max_depth=3: 木の深さの上限 -
random_state=0: データ分割やモデル内部のランダム処理を固定する設定(再現性の確保)
❓ジニ係数(Gini Impurity)とは❓
クラス(分類対象)がどれだけ混ざっているかを示す指標です。
- 0 に近いほど → ほぼ1つのクラスにまとまっている状態
- 1 に近いほど → 複数クラスが均等に混ざっている状態
-
clf.fit(X_train, y_train): 訓練データをもとに、どの特徴量をどの順番で分割すれば目的変数を分類できるかを学習します。
この時点で木の構造が決まります -
fit(): 訓練済みのモデル(self)を返すため、modelには学習済みの決定木が入ります。
Python(scikit-learn)でのモデル構築の流れ
- まず「モデルの設計図(クラス)」を用意
- その後に「学習させる(fit)」
つまり今回の決定木は、「こういうルールで分割してね」と学習させた状態です。
#============================================
# 6. 関数:決定木のルールを作成
#============================================
def add_class_legend(legend_text, x=0.02, y=0.98):
plt.gcf().text(
x, y,
legend_text,
fontsize=13,
va="top",
ha="left",
bbox=dict(
boxstyle="round,pad=0.5",
fc="lightyellow",
ec="gray",
lw=1
)
)
#===========================================
# 7. クラスインデックス作成
#===========================================
legend_text = "Class index mapping:\n" + "\n".join(
[f"{idx}: {label}" for idx, label in enumerate(model.classes_)]
)
#===========================================
# 8. 決定木の描画
#===========================================
# 比率なし
plt.figure(figsize=(20,10))
plot_tree(model, feature_names=X_df.columns,
class_names=model.classes_.astype(str),
filled=True, max_depth=6)
add_class_legend(legend_text)
plt.savefig(Non_save_path, dpi=300)
plt.show()
# 比率あり
plt.figure(figsize=(20,10))
plot_tree(model, feature_names=X_df.columns,
class_names=model.classes_.astype(str),
filled=True, max_depth=6, proportion=True)
add_class_legend(legend_text)
plt.savefig(save_path, dpi=300)
plt.show()
# OS別で出力フォルダを開く
if sys.platform.startswith('win'):
os.startfile(output_path)
elif sys.platform.startswith('darwin'):
subprocess.run(['open', output_path])
else:
subprocess.run(['xdg-open', output_path])
print('処理完了')
6. 決定木のルールを作成
木の分岐ルールが完成
-
def add_class_legend(): テキストで凡例(目的変数)を追加 -
model.fit(X_df, y_df): 訓練データだけでなく 全データを使って決定木のルールが構築 される
7. クラスインデックス作成
-
model.classes_: 目的変数(car)のユニークなクラス名を配列で返す
例 →["foreign car", "honda", "mitsubishi", "nissan", "toyota"] -
enumerate(): インデックスとクラス名を組み合わせ、凡例に「0: foreign car」などと表示
出力例

8. 決定木の描画
学習済みのモデルをもとに、決定木を可視化します。
クラスの対応表をグラフ内に追加し、ノードごとに分岐条件・サンプル数・比率を表示しています。
-
model: 学習済みの決定木 -
feature_names=X_df.columns: 特徴量の列名をノードに表示 -
class_names=model.classes_.astype(str): ノードにクラス名(文字列)を表示 -
filled=True: ノードに色を付ける -
max_depth=6: ツリーの深さを指定(木が浅ければ実際の深さ分しか描画されない)
補足
max_depth=6 を指定しても、木の実際の深さが 4 なら4階層までしか描画されません。
これは「最大で 6 階層まで表示できる」という上限設定です。
-
proportion=True: サンプル数を割合で表示
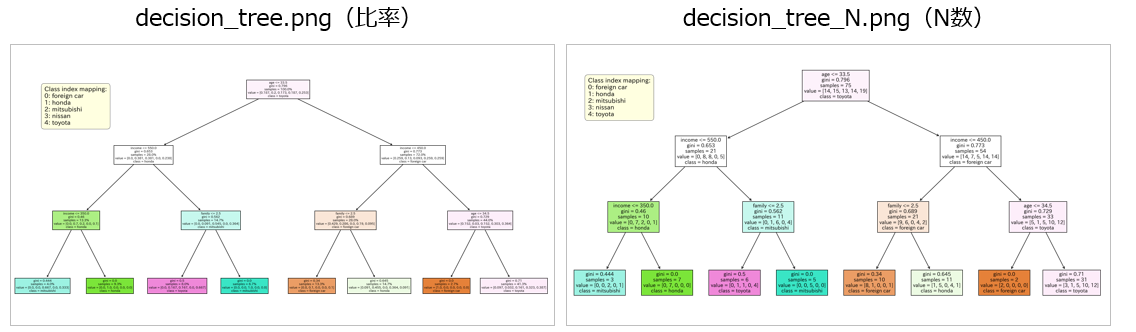
ノードの読み取り方
-
age<= 33.5: 条件が真なら左へ、偽なら右へ分岐 -
gini=0.796: 不純度を示す指標。0に近いほど純粋(1つのクラスに偏っている) -
value=[14,15,13,14,19]: 各クラス([foreign car, honda, mitsubishi, nissan, toyota])のサンプル数 -
class: 最も多いクラス(図の場合は toyota) -
sample =100人: このノードに属するデータ数(全体で100件)

age <= 33.5の意味
age が 33.5 以下なら左へ、33.5 より大きければ右へ
- 33歳 → 左に進む
- 34歳 → 右に進む
👉 scikit-learn の決定木は境界値を小数で表します。
整数データでも、境界値に従って分岐するので問題ありません。
⚠️ ただし「性別」などのカテゴリ変数を数値化すると、大小関係で分割されてしまう点に注意。
これは R の決定木(カテゴリ型をそのまま扱う)との違いです。
📌比較
R版コード例
#----------------------------------------------
# ライブラリ
#----------------------------------------------
library(rpart)
library(rpart.plot)
#----------------------------------------------
# ディレクトリ・ファイル設定(親ディレクトリ基準)
#----------------------------------------------
current_path <- getwd()
parent_path <- dirname(current_path)
INPUT_FOLDER <- "2_data"
OUTPUT_FOLDER <- "3_output"
INPUT_FILE <- "sample.csv"
input_path <- file.path(parent_path, INPUT_FOLDER, INPUT_FILE)
output_path <- file.path(parent_path, OUTPUT_FOLDER)
if (!dir.exists(output_path)) dir.create(output_path)
# 出力ファイル名
save_name <- file.path(output_path, "R_decision_tree.png") # 比率あり
non_save_name <- file.path(output_path, "R_decision_tree_N.png") # 人数のみ
#----------------------------------------------
# CSV読み込み(文字コード自動判定)
#----------------------------------------------
# まずUTF-8で読み込み、失敗したらCP932(Shift-JIS)で
df <- tryCatch({
read.csv(input_path, fileEncoding = "UTF-8")
}, error = function(e) {
read.csv(input_path, fileEncoding = "CP932")
})
#----------------------------------------------
# 除外カラム設定・目的変数
#----------------------------------------------
ID <- "ID"
cluster <- "cluster"
target_col <- "car"
x_df <- df[, setdiff(names(df), c(ID, cluster, target_col))]
y_df <- df[[target_col]]
# rpart用に結合
data_all <- data.frame(y_df, x_df)
#----------------------------------------------
# 訓練・テストデータ分割(75% / 25%)
#----------------------------------------------
set.seed(0) # 再現性確保
train_idx <- sample(seq_len(nrow(data_all)), size = 0.75 * nrow(data_all))
train_data <- data_all[train_idx, ]
test_data <- data_all[-train_idx, ]
cat(sprintf("学習データ数: %d / テストデータ数: %d\n", nrow(train_data), nrow(test_data)))
#----------------------------------------------
# 決定木モデル構築(max_depth=3)
#----------------------------------------------
model <- rpart(y_df ~ .,
data = train_data,
method = "class",
parms = list(split = "gini"),
control = rpart.control(maxdepth = 3))
# 正解率計算
train_pred <- predict(model, train_data, type = "class")
train_acc <- mean(train_pred == train_data$y_df)
test_pred <- predict(model, test_data, type = "class")
test_acc <- mean(test_pred == test_data$y_df)
cat(sprintf("正解率(train): %.3f / 正解率(test): %.3f\n", train_acc, test_acc))
#----------------------------------------------
# 決定木作成(全データ, maxdepth=6)
#----------------------------------------------
model_full <- rpart(y_df ~ .,
data = data_all,
method = "class",
parms = list(split = "gini"),
control = rpart.control(maxdepth = 6))
#----------------------------------------------
# 決定木プロット(比率あり) & PNG保存
#----------------------------------------------
png(save_name, width = 1000, height = 800)
rpart.plot(model_full, type = 2, extra = 104, fallen.leaves = TRUE, main = "決定木(比率)")
dev.off()
#----------------------------------------------
# 決定木プロット(人数のみ) & PNG保存
#----------------------------------------------
png(non_save_name, width = 1000, height = 800)
rpart.plot(model_full, type = 2, extra = 1, fallen.leaves = TRUE, main = "決定木(人数のみ)")
dev.off()
cat("処理完了\n")
1. 正解率比較
| train正解率 | test正解率 | |
|---|---|---|
| Python | 0.600 | 0.320 |
| R | 0.467 | 0.360 |
2. 決定木の比較
(1) 見た目

python
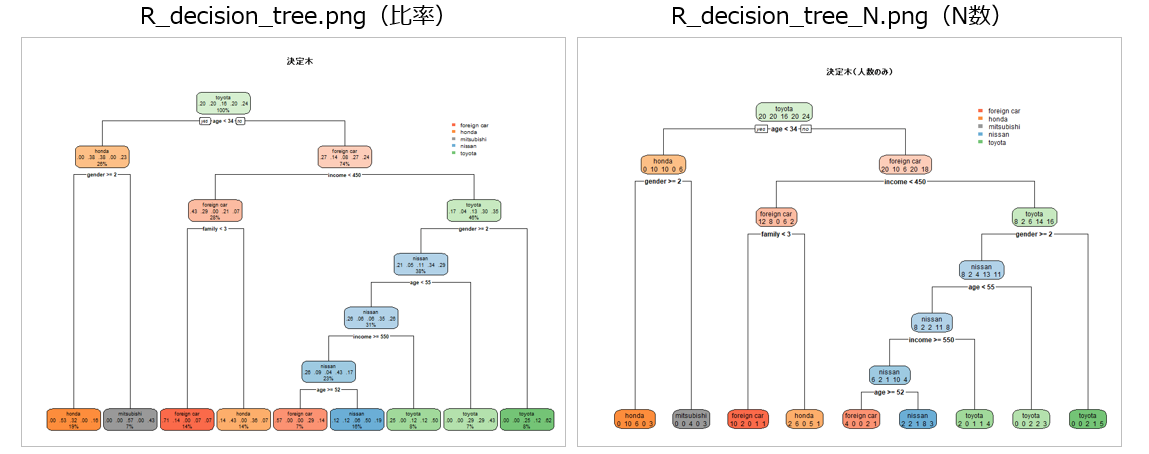
R
並べて比較

(2) モデルの特徴差
| 要因 | Python (DecisionTreeClassifier) |
R (rpart) |
|---|---|---|
| 分割基準 | 既定でジニ不純度 | 既定でジニ不純度 or 情報利得(制御可能) |
| 枝刈り(pruning) | デフォルトでは枝刈りなし(max_depth等で調整) |
自動的に cp(複雑度パラメータ)に基づいて枝刈り |
| カテゴリ変数の扱い | 数値化され、大小比較による二分割 | カテゴリ型をそのまま扱い、最適なグルーピングで分割 |
| 乱数シード | 同様に乱数依存だが、計算順序も影響 | 分割候補選択に乱数が絡む場合あり |
| 共通点 |
income と age は重要な特徴量として認識 |
同左 |
| 補足 |
feature_importances_ のスケールはRと異なる絶対値ではなく相対比較が重要 |
variable.importance のスケールはPythonと異なる絶対値ではなく相対比較が重要 |
(3) 特徴量重要度の比較
| 特徴量 | Python (feature_importances_) | R (variable.importance) |
|---|---|---|
| family | 0.313 | —(小さく表示されず) |
| age | 0.276 | 4.776 |
| gender | 0.000 | 5.364 |
| income | 0.411 | 2.593 |
📌それでどうする
1. 実務や簡単な分析なら
-
Python (scikit-learn)
- データ前処理や他の機械学習と統合しやすい
- Jupyter/Notebookや
Streamlitでそのまま可視化・自動化できる - 特徴量の重要度や結果の再利用が簡単
2. R (rpart) を使う場面
-
R (rpart)
- R環境に慣れている場合や、既存のRコードやレポートと連携したい場合
- 自動枝刈りや見た目の調整がしやすく、統計的に簡単に扱える
3. 両方を理解しておくメリット
- 「決定木の理屈」は両方共通
- 実装の違い(枝刈り、分岐候補、カテゴリ扱いなど)を理解すれば、ライブラリを問わず同じ分析方針で進められる
- データや環境に応じて、柔軟に使い分けるのが現実的
結論
| 影響の大きさ | 条件・状況 | 具体例 |
|---|---|---|
| ほとんど影響しない | - 目的が傾向把握や特徴量の重要度可視化 - 前処理やパラメータがRとPythonでほぼ一致 |
- 木の形は少し違っても主要分岐は同じ - 木の深さや枝刈り基準が違っても大勢は変わらない |
| 影響が出やすい | - カテゴリ変数が多く、RとPythonで扱いが異なる - 枝刈りや分岐アルゴリズムが違う - モデル結果をそのまま予測や意思決定に使う |
- scikit-learnではカテゴリを数値比較して分岐 - Rではカテゴリ型をそのまま分岐 - 分岐ルールが大きく変わり予測精度も変動 |
📌まとめ
分析は好きな方法でOK!
状況に応じて柔軟に使い分けましょう。
参考リンクについて
GitHubリポジトリ
本記事で紹介したコードやサンプルデータはこちらで公開しています。
Discussion