(3) 「Dev Containers」で構築したCWLの作成環境で、実践的なワークフローを作成、実行する
今回の記事で使用したCWLのファイルをおいているリポジトリは以下からアクセスすることができます。
前回の記事はこちらです↓
はじめに
これまで、環境構築およびgrepとwcの処理に関するワークフローを作成する過程を紹介しました。
この記事では、具体的にバイオインフォマティクス分野で使用されているツールの例をCWLとして記述し、 ワークフロー として実行する部分までをご紹介します。
このプロセスにおいては、これまでと同様にzatsu-cwl-generatorを軸としてCWLファイルを作成し、その後に細かい修正を加えていきながら作成していきます。
実行するプロセスの概要
今回は以下に示す5つのステップをワークフローとして記述します。
(1)blastpコマンドによる配列類似性検索
(2)awkによるヒットしたIDの抽出
(3)blastdbcmdによるヒットしたIDのfastaファイルの抽出
(4)ClustalOmegaによるマルチプルアラインメント
(5)fasttreeによるnewick形式ファイルの出力
これにより、BLASTpでヒットした配列から、系統樹を作成する前段階の処理(newick形式のファイルを出力)をワークフローとして記述します。
CWLとして記述するコマンドは以下の通りです。
このように、5つのステップをワークフローとして記述します。
# Step1
blastp -query MSTN.fasta -db uniprot_sprot.fasta -evalue 1e-5 -num_threads 4 -outfmt 6 -out blastp_result.txt -max_target_seqs 20
# Step2
awk '{ print $2 }' blastp_result.txt > blastp_result_id.txt
#Step3
blastdbcmd -db uniprot_sprot.fasta -entry_batch blastp_result_id.txt -out blastdbcmd_result.fasta
#Step4
clustalo -i blastdbcmd_result.fasta --outfmt=fasta -o clustalo_result.fasta
#Step5
FastTree -boot 100 -out MSTN_tree.newick clustalo_result.fasta
zatsu-cwl-generatorを使ってblastpを実行するCWLファイルを書く
次に、上記の処理について、CWLファイルを記述していきましょう。 zatsu-cwl-generatorを使ってコードを生成し、修正しながら作成していきます。
今回は、zatsu_cwl_bioinformaticsディレクトリで作業を行っています。修正のプロセスはトグル内に示してあるので、ぜひご覧ください。
以下では、最初のプロセス、blastpのコマンドを例にして(修正のプロセスも含めながら)説明していきます。
(1) zatsu-cwl-generatorを使ってblastpのCWLファイルを生成する
これまでと同様に、zatsu-cwl-generatorを使ってCWLファイルを生成します。
zatsu-cwl-generator 'blastp -query MSTN.fasta -db uniprot_sprot.fasta -evalue 1e-5 -num_threads 4 -outfmt 6 -out blastp_result.txt -max_target_seqs 20' > 1_blastp.cwl
cwltool --validateでチェックします。
cwltool --validate 1_blastp.cwl
INFO /usr/local/bin/cwltool 3.1.20240508115724
INFO Resolved '1_blastp.cwl' to 'file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/1_blastp.cwl'
1_blastp.cwl:45:7: Warning: Field 'location' contains undefined reference to
'file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/blastp_result.txt'
WARNING 1_blastp.cwl:45:7: Warning: Field 'location' contains undefined reference to
'file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/blastp_result.txt'
1_blastp.cwl is valid CWL.
1_blastp.cwl is valid CWL.という出力がされているものの、WARNING 1_blastp.cwl:45:7: Warning: Field 'location' contains undefined reference to 'file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/blastp_result.txt'として実行例として使った入力ファイルが存在しないことを表す警告が出ていますが、修正方法は後ほど説明します。
(2) コンテナオプション(-c)を使って出力する
CWLでは、docker imageを使って実行する場合、これまでのCWLファイルでは書いていなかったhintsフィールド(あるいはrequirementsフィールド)を使って記述します。
zatsu-cwl-generatorでは、-c, --container オプションを使うことで、コンテナを使用するコマンドを出力することができます。
以下のようにコマンドを実行します。
zatsu-cwl-generator 'blastp -query MSTN.fasta -db uniprot_sprot.fasta -evalue 1e-5 -num_threads 4 -outfmt 6 -out blastp_result.txt -max_target_seqs 20' --container biocontainers/blast:v2.2.31_cv2 > 1_blastp_docker.cwl
これまでと同じようにコマンドを''でくくったあとに、-c もしくは--container オプションをつけて、image(tagの情報も含めて)を記載します。 すると、以下のようにCWLファイルが出力されます。
これまでと同じように出力されますが、コンテナオプションをつけると、下にhintsフィールドが出力されています。
このように、hintsフィールドを使ってdocker imageを指定することができます。
cwltool --validateを使ってチェックします。
cwltool --validate 1_blastp_docker.cwl
INFO /usr/local/bin/cwltool 3.1.20240508115724
INFO Resolved '1_blastp_docker.cwl' to 'file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/1_blastp_docker.cwl'
1_blastp_docker.cwl:45:7: Warning: Field 'location' contains undefined reference to
'file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/blastp_result.txt'
WARNING 1_blastp_docker.cwl:45:7: Warning: Field 'location' contains undefined reference to
'file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/blastp_result.txt'
1_blastp_docker.cwl is valid CWL.
こちらのバージョンについても、1_blastp_docker.cwl:45:7: Warning: Field 'location' contains undefined reference to 'file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/blastp_result.txt'として実行例として使った入力ファイルが存在しないことを表す警告が出ています。 (修正方法は後ほど説明します。)
一旦このファイルを使って実行してみましょう。
cwltool --debug 1_blastp_docker.cwl
これまでと異なり、エラーが発生しました。
エラー内容
INFO /usr/local/bin/cwltool 3.1.20240508115724
INFO Resolved '1_blastp_docker.cwl' to 'file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/1_blastp_docker.cwl'
1_blastp_docker.cwl:45:7: Warning: Field 'location' contains undefined reference to
'file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/blastp_result.txt'
WARNING 1_blastp_docker.cwl:45:7: Warning: Field 'location' contains undefined reference to
'file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/blastp_result.txt'
DEBUG Can't make command line argument from Any
DEBUG Parsed job order from command line: {
"__id": "1_blastp_docker.cwl",
"query": {
"class": "File",
"location": "file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/MSTN.fasta"
},
"db": {
"class": "File",
"location": "file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta"
},
"num_threads": 8,
"outfmt": 7,
"out": {
"class": "File",
"location": "file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/blastp_result.txt"
},
"max_target_seqs": 20
}
DEBUG [job 1_blastp_docker.cwl] initializing from file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/1_blastp_docker.cwl
DEBUG [job 1_blastp_docker.cwl] {
"query": {
"class": "File",
"location": "file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/MSTN.fasta",
"size": 476,
"basename": "MSTN.fasta",
"nameroot": "MSTN",
"nameext": ".fasta"
},
"db": {
"class": "File",
"location": "file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta",
"size": 284812502,
"basename": "uniprot_sprot.fasta",
"nameroot": "uniprot_sprot",
"nameext": ".fasta"
},
"num_threads": 8,
"outfmt": 7,
"out": {
"class": "File",
"location": "file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/blastp_result.txt",
"basename": "blastp_result.txt",
"nameroot": "blastp_result",
"nameext": ".txt"
},
"max_target_seqs": 20,
"evalue": 1e-05
}
ERROR Input object failed validation:
[Errno 2] No such file or directory: '/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/blastp_result.txt'
Traceback (most recent call last):
File "/usr/local/lib/python3.10/site-packages/cwltool/pathmapper.py", line 178, in visit
st = os.lstat(deref)
FileNotFoundError: [Errno 2] No such file or directory: '/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/blastp_result.txt'
During handling of the above exception, another exception occurred:
Traceback (most recent call last):
File "/usr/local/lib/python3.10/site-packages/cwltool/main.py", line 1314, in main
(out, status) = real_executor(
File "/usr/local/lib/python3.10/site-packages/cwltool/executors.py", line 63, in __call__
return self.execute(process, job_order_object, runtime_context, logger)
File "/usr/local/lib/python3.10/site-packages/cwltool/executors.py", line 146, in execute
self.run_jobs(process, job_order_object, logger, runtime_context)
File "/usr/local/lib/python3.10/site-packages/cwltool/executors.py", line 221, in run_jobs
for job in jobiter:
File "/usr/local/lib/python3.10/site-packages/cwltool/command_line_tool.py", line 992, in job
builder.pathmapper = self.make_path_mapper(reffiles, builder.stagedir, runtimeContext, True)
File "/usr/local/lib/python3.10/site-packages/cwltool/command_line_tool.py", line 485, in make_path_mapper
return PathMapper(reffiles, runtimeContext.basedir, stagedir, separateDirs)
File "/usr/local/lib/python3.10/site-packages/cwltool/pathmapper.py", line 104, in __init__
self.setup(dedup(referenced_files), basedir)
File "/usr/local/lib/python3.10/site-packages/cwltool/pathmapper.py", line 207, in setup
self.visit(
File "/usr/local/lib/python3.10/site-packages/cwltool/pathmapper.py", line 167, in visit
with SourceLine(
File "schema_salad/sourceline.py", line 249, in __exit__
schema_salad.exceptions.ValidationException: [Errno 2] No such file or directory: '/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinforma
tics/blastp_result.txt'
出力ファイルが存在しないというエラー(FileNotFoundError: [Errno 2] No such file or directory: '/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/blastp_result.txt')が出ています。
これはcwltool --validateを実行したときの警告と関係があるようです。
それではこれについて修正してみましょう。
(3) 修正プロセス 1
inputsフィールドに書いてある部分が修正が必要のようです。
ここのblastp_result.txtはファイルそのものではなく、ファイル名 、つまり文字列になるはずなので、以下のように修正してみました。
それでは再度実行してみましょう。
修正後のCWLファイル
cwltool --debug 1_blastp_docker_v2.cwl
INFO /usr/local/bin/cwltool 3.1.20240508115724
INFO Resolved '1_blastp_docker_v2.cwl' to 'file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/1_blastp_docker_v2.cwl'
DEBUG Can't make command line argument from Any
DEBUG Parsed job order from command line: {
"__id": "1_blastp_docker_v2.cwl",
"query": {
"class": "File",
"location": "file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/MSTN.fasta"
},
"db": {
"class": "File",
"location": "file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta"
},
"num_threads": 8,
"outfmt": 7,
"out": "blastp_result.txt",
"max_target_seqs": 20
}
DEBUG [job 1_blastp_docker_v2.cwl] initializing from file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/1_blastp_docker_v2
.cwl DEBUG [job 1_blastp_docker_v2.cwl] {
"query": {
"class": "File",
"location": "file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/MSTN.fasta",
"size": 476,
"basename": "MSTN.fasta",
"nameroot": "MSTN",
"nameext": ".fasta"
},
"db": {
"class": "File",
"location": "file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta",
"size": 284812502,
"basename": "uniprot_sprot.fasta",
"nameroot": "uniprot_sprot",
"nameext": ".fasta"
},
"num_threads": 8,
"outfmt": 7,
"out": "blastp_result.txt",
"max_target_seqs": 20,
"evalue": 1e-05
}
DEBUG [job 1_blastp_docker_v2.cwl] path mappings is {
"file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/MSTN.fasta": [
"/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/MSTN.fasta",
"/var/lib/cwl/stga7998393-9a81-4cc7-9591-8fe9236fcf5d/MSTN.fasta",
"File",
true
],
"file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta": [
"/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta",
"/var/lib/cwl/stgb5786c95-6694-4f59-9707-d79eced66e64/uniprot_sprot.fasta",
"File",
true
]
}
DEBUG [job 1_blastp_docker_v2.cwl] command line bindings is [
{
"position": [
-1000000,
0
],
"datum": "blastp"
},
{
"position": [
0,
0
],
"datum": "-query"
},
{
"position": [
0,
1
],
"valueFrom": "$(inputs.query)"
},
{
"position": [
0,
2
],
"datum": "-db"
},
{
"position": [
0,
3
],
"valueFrom": "$(inputs.db)"
},
{
"position": [
0,
4
],
"datum": "-evalue"
},
{
"position": [
0,
5
],
"valueFrom": "$(inputs.evalue)"
},
{
"position": [
0,
6
],
"datum": "-num_threads"
},
{
"position": [
0,
7
],
"valueFrom": "$(inputs.num_threads)"
},
{
"position": [
0,
8
],
"datum": "-outfmt"
},
{
"position": [
0,
9
],
"valueFrom": "$(inputs.outfmt)"
},
{
"position": [
0,
10
],
"datum": "-out"
},
{
"position": [
0,
11
],
"valueFrom": "$(inputs.out)"
},
{
"position": [
0,
12
],
"datum": "-max_target_seqs"
},
{
"position": [
0,
13
],
"valueFrom": "$(inputs.max_target_seqs)"
}
]
DEBUG [job 1_blastp_docker_v2.cwl] initial work dir {}
INFO [job 1_blastp_docker_v2.cwl] /tmp/17begc9b$ docker \
run \
-i \
--mount=type=bind,source=/tmp/17begc9b,target=/jFvgAu \
--mount=type=bind,source=/tmp/pvwugmew,target=/tmp \
--mount=type=bind,source=/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/MSTN.fasta,target=/var/lib/cwl/stga7998393-9a81-4
cc7-9591-8fe9236fcf5d/MSTN.fasta,readonly \ --mount=type=bind,source=/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta,target=/var/lib/cwl/stgb5786c
95-6694-4f59-9707-d79eced66e64/uniprot_sprot.fasta,readonly \ --workdir=/jFvgAu \
--read-only=true \
--user=1000:1000 \
--rm \
--cidfile=/tmp/nquyx9tk/20240527074301-528880.cid \
--env=TMPDIR=/tmp \
--env=HOME=/jFvgAu \
biocontainers/blast:v2.2.31_cv2 \
blastp \
-query \
/var/lib/cwl/stga7998393-9a81-4cc7-9591-8fe9236fcf5d/MSTN.fasta \
-db \
/var/lib/cwl/stgb5786c95-6694-4f59-9707-d79eced66e64/uniprot_sprot.fasta \
-evalue \
0.00001 \
-num_threads \
8 \
-outfmt \
7 \
-out \
blastp_result.txt \
-max_target_seqs \
20
WARNING: The requested image's platform (linux/amd64) does not match the detected host platform (linux/arm64/v8) and no specific platform was reque
sted BLAST Database error: No alias or index file found for protein database [/var/lib/cwl/stgb5786c95-6694-4f59-9707-d79eced66e64/uniprot_sprot.fasta]
in search path [/jFvgAu::] WARNING [job 1_blastp_docker_v2.cwl] exited with status: 2
WARNING [job 1_blastp_docker_v2.cwl] completed permanentFail
DEBUG [job 1_blastp_docker_v2.cwl] outputs {
"all-for-debugging": [
{
"location": "file:///tmp/17begc9b/blastp_result.txt",
"basename": "blastp_result.txt",
"nameroot": "blastp_result",
"nameext": ".txt",
"class": "File",
"checksum": "sha1$da39a3ee5e6b4b0d3255bfef95601890afd80709",
"size": 0,
"http://commonwl.org/cwltool#generation": 0
}
]
}
DEBUG [job 1_blastp_docker_v2.cwl] Removing input staging directory /tmp/e_qr0ldv
DEBUG [job 1_blastp_docker_v2.cwl] Removing temporary directory /tmp/pvwugmew
DEBUG Moving /tmp/17begc9b/blastp_result.txt to /workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/blastp_result.txt
DEBUG Removing intermediate output directory /tmp/17begc9b
{
"all-for-debugging": [
{
"location": "file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/blastp_result.txt",
"basename": "blastp_result.txt",
"class": "File",
"checksum": "sha1$da39a3ee5e6b4b0d3255bfef95601890afd80709",
"size": 0,
"path": "/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/blastp_result.txt"
}
]
}WARNING Final process status is permanentFail
今回はファイルの出力はされたものの、空のファイルが出力されてしまいました。
エラーを見てみると、BLAST Database error: No alias or index file found for protein database [/var/lib/cwl/stgb5786c95-6694-4f59-9707-d79eced66e64/uniprot_sprot.fasta]などが見られ、データベースが見つからないというエラーが出ています。
これはBLASTなどに見られるエラーで、 インデックス作成時に複数のインデックスファイルも含めて記述する必要があります。
それでは、これを修正してみましょう。
(4) 修正プロセス 2 複数のインデックスファイルの記載
inputsフィールドの部分を修正してみます。
BLASTでは、inputsのインデックス(引数は -db )の指定の部分の記述方法について下記のような方法があるようです。
この記事では別のアプローチとして、 secondaryFiles フィールドを活用し、以下のように記述してみます。
このように記述することで、複数のインデックスファイルを指定することができます。
それでは再度実行してみましょう。
修正後のcwlファイルでの実行結果
cwltool --debug 1_blastp_docker_v3.cwl
INFO /usr/local/bin/cwltool 3.1.20240508115724
INFO Resolved '1_blastp_docker_v3.cwl' to 'file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/1_blastp_docker_v3.cwl'
DEBUG Can't make command line argument from Any
DEBUG Parsed job order from command line: {
"__id": "1_blastp_docker_v3.cwl",
"query": {
"class": "File",
"location": "file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/MSTN.fasta"
},
"db": {
"class": "File",
"location": "file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta"
},
"num_threads": 8,
"outfmt": 7,
"out": "blastp_result.txt",
"max_target_seqs": 20
}
DEBUG [job 1_blastp_docker_v3.cwl] initializing from file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/1_blastp_docker_v3
.cwl DEBUG [job 1_blastp_docker_v3.cwl] {
"query": {
"class": "File",
"location": "file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/MSTN.fasta",
"size": 476,
"basename": "MSTN.fasta",
"nameroot": "MSTN",
"nameext": ".fasta"
},
"db": {
"class": "File",
"location": "file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta",
"size": 284812502,
"basename": "uniprot_sprot.fasta",
"nameroot": "uniprot_sprot",
"nameext": ".fasta",
"secondaryFiles": [
{
"location": "file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta.phd",
"basename": "uniprot_sprot.fasta.phd",
"class": "File",
"nameroot": "uniprot_sprot.fasta",
"nameext": ".phd"
},
{
"location": "file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta.phi",
"basename": "uniprot_sprot.fasta.phi",
"class": "File",
"nameroot": "uniprot_sprot.fasta",
"nameext": ".phi"
},
{
"location": "file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta.phr",
"basename": "uniprot_sprot.fasta.phr",
"class": "File",
"nameroot": "uniprot_sprot.fasta",
"nameext": ".phr"
},
{
"location": "file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta.pin",
"basename": "uniprot_sprot.fasta.pin",
"class": "File",
"nameroot": "uniprot_sprot.fasta",
"nameext": ".pin"
},
{
"location": "file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta.pog",
"basename": "uniprot_sprot.fasta.pog",
"class": "File",
"nameroot": "uniprot_sprot.fasta",
"nameext": ".pog"
},
{
"location": "file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta.psd",
"basename": "uniprot_sprot.fasta.psd",
"class": "File",
"nameroot": "uniprot_sprot.fasta",
"nameext": ".psd"
},
{
"location": "file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta.psi",
"basename": "uniprot_sprot.fasta.psi",
"class": "File",
"nameroot": "uniprot_sprot.fasta",
"nameext": ".psi"
},
{
"location": "file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta.psq",
"basename": "uniprot_sprot.fasta.psq",
"class": "File",
"nameroot": "uniprot_sprot.fasta",
"nameext": ".psq"
}
]
},
"num_threads": 8,
"outfmt": 7,
"out": "blastp_result.txt",
"max_target_seqs": 20,
"evalue": 1e-05
}
DEBUG [job 1_blastp_docker_v3.cwl] path mappings is {
"file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/MSTN.fasta": [
"/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/MSTN.fasta",
"/var/lib/cwl/stgcb307528-6f4f-4251-9517-0121cb068d7e/MSTN.fasta",
"File",
true
],
"file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta": [
"/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta",
"/var/lib/cwl/stg90d0f4dc-8137-4082-8c5d-4eaf670f8786/uniprot_sprot.fasta",
"File",
true
],
"file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta.phd": [
"/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta.phd",
"/var/lib/cwl/stg90d0f4dc-8137-4082-8c5d-4eaf670f8786/uniprot_sprot.fasta.phd",
"File",
true
],
"file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta.phi": [
"/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta.phi",
"/var/lib/cwl/stg90d0f4dc-8137-4082-8c5d-4eaf670f8786/uniprot_sprot.fasta.phi",
"File",
true
],
"file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta.phr": [
"/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta.phr",
"/var/lib/cwl/stg90d0f4dc-8137-4082-8c5d-4eaf670f8786/uniprot_sprot.fasta.phr",
"File",
true
],
"file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta.pin": [
"/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta.pin",
"/var/lib/cwl/stg90d0f4dc-8137-4082-8c5d-4eaf670f8786/uniprot_sprot.fasta.pin",
"File",
true
],
"file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta.pog": [
"/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta.pog",
"/var/lib/cwl/stg90d0f4dc-8137-4082-8c5d-4eaf670f8786/uniprot_sprot.fasta.pog",
"File",
true
],
"file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta.psd": [
"/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta.psd",
"/var/lib/cwl/stg90d0f4dc-8137-4082-8c5d-4eaf670f8786/uniprot_sprot.fasta.psd",
"File",
true
],
"file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta.psi": [
"/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta.psi",
"/var/lib/cwl/stg90d0f4dc-8137-4082-8c5d-4eaf670f8786/uniprot_sprot.fasta.psi",
"File",
true
],
"file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta.psq": [
"/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta.psq",
"/var/lib/cwl/stg90d0f4dc-8137-4082-8c5d-4eaf670f8786/uniprot_sprot.fasta.psq",
"File",
true
]
}
DEBUG [job 1_blastp_docker_v3.cwl] command line bindings is [
{
"position": [
-1000000,
0
],
"datum": "blastp"
},
{
"position": [
0,
0
],
"datum": "-query"
},
{
"position": [
0,
1
],
"valueFrom": "$(inputs.query)"
},
{
"position": [
0,
2
],
"datum": "-db"
},
{
"position": [
0,
3
],
"valueFrom": "$(inputs.db)"
},
{
"position": [
0,
4
],
"datum": "-evalue"
},
{
"position": [
0,
5
],
"valueFrom": "$(inputs.evalue)"
},
{
"position": [
0,
6
],
"datum": "-num_threads"
},
{
"position": [
0,
7
],
"valueFrom": "$(inputs.num_threads)"
},
{
"position": [
0,
8
],
"datum": "-outfmt"
},
{
"position": [
0,
9
],
"valueFrom": "$(inputs.outfmt)"
},
{
"position": [
0,
10
],
"datum": "-out"
},
{
"position": [
0,
11
],
"valueFrom": "$(inputs.out)"
},
{
"position": [
0,
12
],
"datum": "-max_target_seqs"
},
{
"position": [
0,
13
],
"valueFrom": "$(inputs.max_target_seqs)"
}
]
DEBUG [job 1_blastp_docker_v3.cwl] initial work dir {}
INFO [job 1_blastp_docker_v3.cwl] /tmp/8hge4mo3$ docker \
run \
-i \
--mount=type=bind,source=/tmp/8hge4mo3,target=/RrTBZF \
--mount=type=bind,source=/tmp/7sot2rlv,target=/tmp \
--mount=type=bind,source=/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/MSTN.fasta,target=/var/lib/cwl/stgcb307528-6f4f-4
251-9517-0121cb068d7e/MSTN.fasta,readonly \ --mount=type=bind,source=/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta,target=/var/lib/cwl/stg90d0f4
dc-8137-4082-8c5d-4eaf670f8786/uniprot_sprot.fasta,readonly \ --mount=type=bind,source=/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta.phd,target=/var/lib/cwl/stg90
d0f4dc-8137-4082-8c5d-4eaf670f8786/uniprot_sprot.fasta.phd,readonly \ --mount=type=bind,source=/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta.phi,target=/var/lib/cwl/stg90
d0f4dc-8137-4082-8c5d-4eaf670f8786/uniprot_sprot.fasta.phi,readonly \ --mount=type=bind,source=/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta.phr,target=/var/lib/cwl/stg90
d0f4dc-8137-4082-8c5d-4eaf670f8786/uniprot_sprot.fasta.phr,readonly \ --mount=type=bind,source=/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta.pin,target=/var/lib/cwl/stg90
d0f4dc-8137-4082-8c5d-4eaf670f8786/uniprot_sprot.fasta.pin,readonly \ --mount=type=bind,source=/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta.pog,target=/var/lib/cwl/stg90
d0f4dc-8137-4082-8c5d-4eaf670f8786/uniprot_sprot.fasta.pog,readonly \ --mount=type=bind,source=/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta.psd,target=/var/lib/cwl/stg90
d0f4dc-8137-4082-8c5d-4eaf670f8786/uniprot_sprot.fasta.psd,readonly \ --mount=type=bind,source=/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta.psi,target=/var/lib/cwl/stg90
d0f4dc-8137-4082-8c5d-4eaf670f8786/uniprot_sprot.fasta.psi,readonly \ --mount=type=bind,source=/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta.psq,target=/var/lib/cwl/stg90
d0f4dc-8137-4082-8c5d-4eaf670f8786/uniprot_sprot.fasta.psq,readonly \ --workdir=/RrTBZF \
--read-only=true \
--user=1000:1000 \
--rm \
--cidfile=/tmp/jj7sjx1j/20240527075910-603277.cid \
--env=TMPDIR=/tmp \
--env=HOME=/RrTBZF \
biocontainers/blast:v2.2.31_cv2 \
blastp \
-query \
/var/lib/cwl/stgcb307528-6f4f-4251-9517-0121cb068d7e/MSTN.fasta \
-db \
/var/lib/cwl/stg90d0f4dc-8137-4082-8c5d-4eaf670f8786/uniprot_sprot.fasta \
-evalue \
0.00001 \
-num_threads \
8 \
-outfmt \
7 \
-out \
blastp_result.txt \
-max_target_seqs \
20
WARNING: The requested image's platform (linux/amd64) does not match the detected host platform (linux/arm64/v8) and no specific platform was reque
sted INFO [job 1_blastp_docker_v3.cwl] completed success
DEBUG [job 1_blastp_docker_v3.cwl] outputs {
"all-for-debugging": [
{
"location": "file:///tmp/8hge4mo3/blastp_result.txt",
"basename": "blastp_result.txt",
"nameroot": "blastp_result",
"nameext": ".txt",
"class": "File",
"checksum": "sha1$48540e3f3f63e4dd5a26ca6899cea47e8447849e",
"size": 1923,
"http://commonwl.org/cwltool#generation": 0
}
]
}
DEBUG [job 1_blastp_docker_v3.cwl] Removing input staging directory /tmp/4qgo5l0s
DEBUG [job 1_blastp_docker_v3.cwl] Removing temporary directory /tmp/7sot2rlv
DEBUG Moving /tmp/8hge4mo3/blastp_result.txt to /workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/blastp_result.txt
DEBUG Removing intermediate output directory /tmp/8hge4mo3
{
"all-for-debugging": [
{
"location": "file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/blastp_result.txt",
"basename": "blastp_result.txt",
"class": "File",
"checksum": "sha1$48540e3f3f63e4dd5a26ca6899cea47e8447849e",
"size": 1923,
"path": "/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/blastp_result.txt"
}
]
}INFO Final process status is success
今回は成功しています! 結果を見てみましょう。
しっかり出力結果も得られています! ウシのミオスタチンと配列類似性があるタンパク質配列が得られています。
これでCWLの修正は完了ですが、以下のように、更に改善することもできます。
(5) 型の指定
上記のCWLファイルの例では、E-valueの指定をAnyにしていましたが、具体的な型を指定することで、より正確な入力を受け付けることができます。
E-valueは1e-05のように入力するため、floatとして記載しました。
これにより、Expecting one of: ['Directory', 'File', 'boolean', 'double', 'float', 'int', 'long', 'null', 'stderr', 'stdout', 'string'] とハイライトされていた部分が解消されました。
(この部分を修正したファイルはv4として保存しています)
他のファイルもCWLファイルとして記述する
blastpのプロセスのように、他のプロセスもCWLファイルとして書いてみます。
zatsu-cwl-generatorを使って生成し、修正したバージョンはv2などのようにファイル名を変更しています。
また、awk以外のプロセスはdocker imageとしてBioContainersにあるものを使用し、hintsフィールドを追加しています。
2_awk_v2.cwl
(1) zatsu-cwl-generatorを使って生成
zatsu-cwl-generator "awk '{ print $2 }' blastp_result.txt > blastp_result_id.txt" > 2_awk.cwl
awkのプロセスでは、argumentsフィールドを追加し、以下のように修正しました。
(2) CWLファイルの評価
cwltool --validate 2_awk_v2.cwl
INFO /usr/local/bin/cwltool 3.1.20240508115724
INFO Resolved '2_awk_v2.cwl' to 'file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/2_awk_v2.cwl'
2_awk_v2.cwl is valid CWL.
(3) 実行
cwltool --debug 2_awk_v2.cwl
実行したファイルは以下の通りです。
3_blastdbcmd_v2.cwl
(1) zatsu-cwl-generatorを使って生成
zatsu-cwl-generator "blastdbcmd -db uniprot_sprot.fasta -entry_batch blastp_result_id.txt -out blastdbcmd_result.fasta" --container biocontainers/blast:v2.2.31_cv2 > 3_blastdbcmd_docker.cwl
1_blastp.cwlのように、secondaryFilesフィールドを使って複数のインデックスファイルを指定している他、ファイル名をstringに変更し、修正を加えました。
(2) CWLファイルの評価
cwltool --validate 3_blastdbcmd_docker_v2.cwl
INFO /usr/local/bin/cwltool 3.1.20240508115724
INFO Resolved '3_blastdbcmd_docker_v2.cwl' to 'file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/3_blastdbcmd_docker_v2.cwl'
3_blastdbcmd_docker_v2.cwl is valid CWL.
(3) 実行
cwltool --debug 3_blastdbcmd_docker_v2.cwl
実行した結果は以下の通りです。 無事、ファイルが出力されています。
4_clustalo.cwl
(1) zatsu-cwl-generatorを使って生成
zatsu-cwl-generator "clustalo -i blastdbcmd_result.fasta --outfmt=fasta -o clustalo_result.fasta" --container biocontainers/clustalo:v1.2.4-2-deb_cv1 > 4_clustalo_docker.cwl
(2) CWLファイルの評価
cwltool --validate 4_clustalo_docker.cwl
INFO /usr/local/bin/cwltool 3.1.20240508115724
INFO Resolved '4_clustalo_docker.cwl' to 'file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/4_clustalo_docker.cwl'
4_clustalo_docker.cwl is valid CWL.
clustaloのプロセスでは修正が必要なく、生成することができているようです... と思いましたが、ワークフローの記述の際に修正が必要になりましたので後ほど修正しています
(3) 実行
cwltool --debug 4_clustalo_docker.cwl
実行した結果は以下の通りです。 無事、ファイルが出力されています。
5_fasttree.cwl
(1) zatsu-cwl-generatorを使って生成
zatsu-cwl-generator "fasttree -nt clustalo_result.fasta > fasttree_result.nwk" --container biocontainers/fasttree:v2.1.10-2-deb_cv1 > 5_fasttree_docker.cwl
(2) CWLファイルの評価
cwltool --validate 5_fasttree_docker.cwl
INFO /usr/local/bin/cwltool 3.1.20240508115724
INFO Resolved '5_fasttree_docker.cwl' to 'file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/5_fasttree_docker.cwl'
5_fasttree_docker.cwl is valid CWL.
fasttreeのプロセスでは、修正が必要なく、生成することができているようです。
(3) 実行
cwltool --debug 5_fasttree_docker.cwl
実行した結果は以下の通りです。 無事、ファイルが出力されています。
全てのプロセスをCWLファイルとして書くことができました。少し修正する部分が必要でしたが、おそらくこれでワークフローも書けるはずです。
このように、zatsu-cwl-generatorを使うことで、簡単にCWLファイルを生成することができます。
ワークフローを記述する
これまで、CommandlinetoolのCWLファイル(一つ一つの処理を記述)を書きました。
次にこれらの5つのステップを実行するワークフロー、blast2tree.cwlを記述していきます。
この例では、ワークフロー全体に関するパラメータを「1_protein_query」のように数字(1_、2_)をつけています。
前の処理のアウトプットを受け取る部分は「blastp_result: step1_blastp/blastp_output_file」のように記述し、それ以外の全ての処理に関するパラメータをinputsで記述しています。
作成したCWLファイルは以下のようになっています。inputsフィールド、stepsフィールド、outputsフィールドをそれぞれ書いてみました。
また、後に実行する際のために、docフィールドも追加しています。
正しい記述かどうかを確認する
一旦 cwltool --validateで書き方を確認してみましょう。
cwltool --validate blast2tree.cwl
INFO /usr/local/bin/cwltool 3.1.20240508115724
INFO Resolved 'blast2tree.cwl' to 'file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/blast2tree.cwl'
ERROR Tool definition failed validation:
blast2tree.cwl:99:11: Source 'all-for-debugging' of type {"type": "array", "items": ["File",
"Directory"]} is incompatible
blast2tree.cwl:104:7: with sink 'nt' of type "File"
blast2tree.cwl:79:11: Source 'all-for-debugging' of type {"type": "array", "items": ["File",
"Directory"]} is incompatible
blast2tree.cwl:84:7: with sink 'blastp_result' of type "File"
blast2tree.cwl:92:11: Source 'all-for-debugging' of type {"type": "array", "items": ["File",
"Directory"]} is incompatible
blast2tree.cwl:97:7: with sink 'i' of type "File"
blast2tree.cwl:92:11: Source 'all-for-debugging' of type {"type": "array", "items": ["File",
"Directory"]} is incompatible
blast2tree.cwl:117:5: with sink 'blastdbcmd_output' of type "File"
blast2tree.cwl:79:11: Source 'all-for-debugging' of type {"type": "array", "items": ["File",
"Directory"]} is incompatible
blast2tree.cwl:109:5: with sink 'blastp_output' of type "File"
blast2tree.cwl:99:11: Source 'all-for-debugging' of type {"type": "array", "items": ["File",
"Directory"]} is incompatible
blast2tree.cwl:121:5: with sink 'clustalo_output' of type "File"
なにやらエラーが出ています。 blast2tree.cwl:79:11: Source 'all-for-debugging' of type {"type": "array", "items": ["File","Directory"]} is incompatible というように、型の不一致があるようです。
このエラーを解消するために、CommandLineTool定義のそれぞれ5つのファイルを少し修正してみます。
例えば、1_blastp_docker_v4.cwlのoutputsフィールドを以下のように修正してみます。
他のファイルも同様に少し修正します。(バージョンをあげました)
修正したファイル
再度作成したCWLファイルは以下のようになっています。
これで再度cwltool --validateを実行してみます。
cwltool --validate blast2tree_v2.cwl
INFO /usr/local/bin/cwltool 3.1.20240508115724
INFO Resolved 'blast2tree_v2.cwl' to 'file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/blast2tree_v2.cwl'
blast2tree_v2.cwl is valid CWL.
これでエラーが解消されました!
CWLviewerでワークフローを確認する
このワークフローの全体像をCWLviewerで確認しましょう。
blast2tree_v2.cwlをCWLviewerにアップロードしてみます。
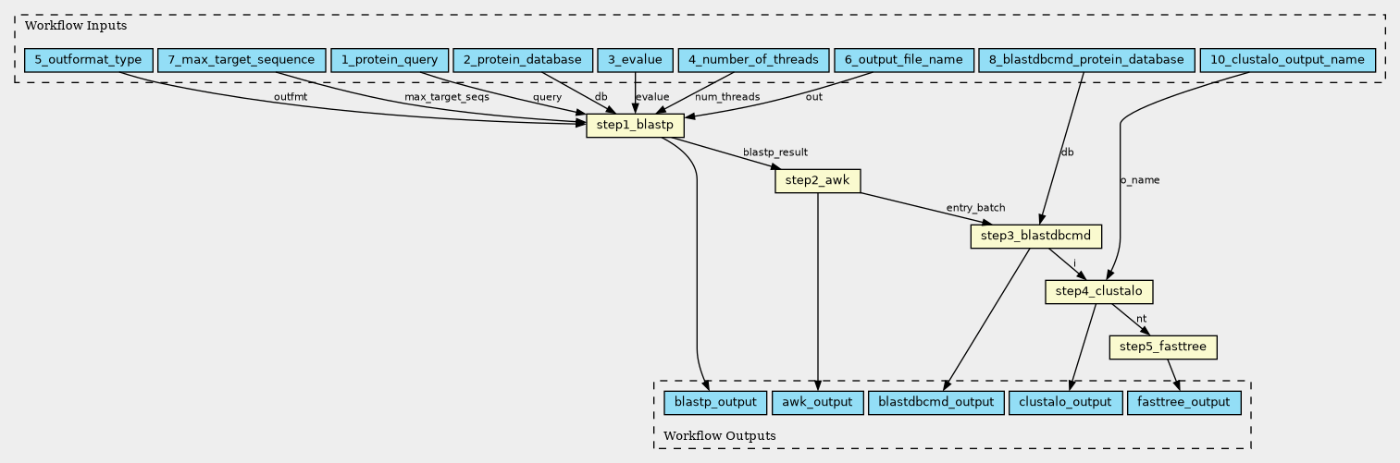
全体としてはこのようになっています。5つものステップがワークフローとして記載されていることが改めてわかります。
(毎回自分はこれに感動しています)
※ 9_clustalo_output_nameのところが10_clustalo_output_nameとなっていますが、修正する前にCWLviewerにアップロードしたのでこのようになっています(プロセスには問題ありません)。
実際に実行する
それでは実際に実行してみましょう。
ワークフローの結果を分けるために、--outdirオプションを使って実行します。
cwl-runner --outdir ./workflow_result/ blast2tree_v2.cwl
ワークフロー実行プロセス
cwl-runner --outdir ./workflow_result/ blast2tree_v2.cwl
INFO /usr/local/bin/cwl-runner 3.1.20240508115724
INFO Resolved 'blast2tree_v2.cwl' to 'file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/blast2tree_v2.cwl'
INFO [workflow ] start
INFO [workflow ] starting step step1_blastp
INFO [step step1_blastp] start
INFO [job step1_blastp] /tmp/2votsws8$ docker \
run \
-i \
--mount=type=bind,source=/tmp/2votsws8,target=/flvvYX \
--mount=type=bind,source=/tmp/ghp_ggow,target=/tmp \
--mount=type=bind,source=/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/MSTN.fasta,target=/var/lib/cwl/stga9b49df8-ee19-46f9-9177-2642075c15fa/
MSTN.fasta,readonly \ --mount=type=bind,source=/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta,target=/var/lib/cwl/stg14f53454-48a1-45aa-9761-7c39
5721a806/uniprot_sprot.fasta,readonly \ --mount=type=bind,source=/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta.phd,target=/var/lib/cwl/stg14f53454-48a1-45aa-9761-
7c395721a806/uniprot_sprot.fasta.phd,readonly \ --mount=type=bind,source=/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta.phi,target=/var/lib/cwl/stg14f53454-48a1-45aa-9761-
7c395721a806/uniprot_sprot.fasta.phi,readonly \ --mount=type=bind,source=/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta.phr,target=/var/lib/cwl/stg14f53454-48a1-45aa-9761-
7c395721a806/uniprot_sprot.fasta.phr,readonly \ --mount=type=bind,source=/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta.pin,target=/var/lib/cwl/stg14f53454-48a1-45aa-9761-
7c395721a806/uniprot_sprot.fasta.pin,readonly \ --mount=type=bind,source=/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta.pog,target=/var/lib/cwl/stg14f53454-48a1-45aa-9761-
7c395721a806/uniprot_sprot.fasta.pog,readonly \ --mount=type=bind,source=/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta.psd,target=/var/lib/cwl/stg14f53454-48a1-45aa-9761-
7c395721a806/uniprot_sprot.fasta.psd,readonly \ --mount=type=bind,source=/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta.psi,target=/var/lib/cwl/stg14f53454-48a1-45aa-9761-
7c395721a806/uniprot_sprot.fasta.psi,readonly \ --mount=type=bind,source=/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta.psq,target=/var/lib/cwl/stg14f53454-48a1-45aa-9761-
7c395721a806/uniprot_sprot.fasta.psq,readonly \ --workdir=/flvvYX \
--read-only=true \
--user=1000:1000 \
--rm \
--cidfile=/tmp/inoumsot/20240612110119-923716.cid \
--env=TMPDIR=/tmp \
--env=HOME=/flvvYX \
biocontainers/blast:v2.2.31_cv2 \
blastp \
-query \
/var/lib/cwl/stga9b49df8-ee19-46f9-9177-2642075c15fa/MSTN.fasta \
-db \
/var/lib/cwl/stg14f53454-48a1-45aa-9761-7c395721a806/uniprot_sprot.fasta \
-evalue \
0.01 \
-num_threads \
8 \
-outfmt \
6 \
-out \
blastp_output.txt \
-max_target_seqs \
20
WARNING: The requested image's platform (linux/amd64) does not match the detected host platform (linux/arm64/v8) and no specific platform was requested
INFO [job step1_blastp] completed success
INFO [step step1_blastp] completed success
INFO [workflow ] starting step step2_awk
INFO [step step2_awk] start
INFO [job step2_awk] /tmp/vmxrp488$ awk \
'{ print $2 }' \
/tmp/7gv05wyk/stga7a18797-23db-4926-a36d-f046b5c40d13/blastp_output.txt > /tmp/vmxrp488/blastp_result_id.txt
INFO [job step2_awk] completed success
INFO [step step2_awk] completed success
INFO [workflow ] starting step step3_blastdbcmd
INFO [step step3_blastdbcmd] start
INFO [job step3_blastdbcmd] /tmp/z0yvl8hc$ docker \
run \
-i \
--mount=type=bind,source=/tmp/z0yvl8hc,target=/flvvYX \
--mount=type=bind,source=/tmp/glzm29j7,target=/tmp \
--mount=type=bind,source=/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta,target=/var/lib/cwl/stg7b0b3776-1de2-4a32-bb1e-475d
84a39cf3/uniprot_sprot.fasta,readonly \ --mount=type=bind,source=/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta.phd,target=/var/lib/cwl/stg7b0b3776-1de2-4a32-bb1e-
475d84a39cf3/uniprot_sprot.fasta.phd,readonly \ --mount=type=bind,source=/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta.phi,target=/var/lib/cwl/stg7b0b3776-1de2-4a32-bb1e-
475d84a39cf3/uniprot_sprot.fasta.phi,readonly \ --mount=type=bind,source=/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta.phr,target=/var/lib/cwl/stg7b0b3776-1de2-4a32-bb1e-
475d84a39cf3/uniprot_sprot.fasta.phr,readonly \ --mount=type=bind,source=/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta.pin,target=/var/lib/cwl/stg7b0b3776-1de2-4a32-bb1e-
475d84a39cf3/uniprot_sprot.fasta.pin,readonly \ --mount=type=bind,source=/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta.pog,target=/var/lib/cwl/stg7b0b3776-1de2-4a32-bb1e-
475d84a39cf3/uniprot_sprot.fasta.pog,readonly \ --mount=type=bind,source=/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta.psd,target=/var/lib/cwl/stg7b0b3776-1de2-4a32-bb1e-
475d84a39cf3/uniprot_sprot.fasta.psd,readonly \ --mount=type=bind,source=/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta.psi,target=/var/lib/cwl/stg7b0b3776-1de2-4a32-bb1e-
475d84a39cf3/uniprot_sprot.fasta.psi,readonly \ --mount=type=bind,source=/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/uniprot_sprot.fasta.psq,target=/var/lib/cwl/stg7b0b3776-1de2-4a32-bb1e-
475d84a39cf3/uniprot_sprot.fasta.psq,readonly \ --mount=type=bind,source=/tmp/vmxrp488/blastp_result_id.txt,target=/var/lib/cwl/stg60f08503-8659-437f-a7cf-02b807cbd09e/blastp_result_id.txt,readonly \
--workdir=/flvvYX \
--read-only=true \
--user=1000:1000 \
--rm \
--cidfile=/tmp/6oya1qwb/20240612110120-940805.cid \
--env=TMPDIR=/tmp \
--env=HOME=/flvvYX \
biocontainers/blast:v2.2.31_cv2 \
blastdbcmd \
-db \
/var/lib/cwl/stg7b0b3776-1de2-4a32-bb1e-475d84a39cf3/uniprot_sprot.fasta \
-entry_batch \
/var/lib/cwl/stg60f08503-8659-437f-a7cf-02b807cbd09e/blastp_result_id.txt \
-out \
blastdbcmd_result.fasta
WARNING: The requested image's platform (linux/amd64) does not match the detected host platform (linux/arm64/v8) and no specific platform was requested
INFO [job step3_blastdbcmd] completed success
INFO [step step3_blastdbcmd] completed success
INFO [workflow ] starting step step4_clustalo
INFO [step step4_clustalo] start
INFO [job step4_clustalo] /tmp/wdeje8er$ docker \
run \
-i \
--mount=type=bind,source=/tmp/wdeje8er,target=/flvvYX \
--mount=type=bind,source=/tmp/5g3q4oh4,target=/tmp \
--mount=type=bind,source=/tmp/z0yvl8hc/blastdbcmd_result.fasta,target=/var/lib/cwl/stgc42faa48-b345-4c6c-9c0c-6d0f6dd22db9/blastdbcmd_result.fasta,readonly \
--workdir=/flvvYX \
--read-only=true \
--user=1000:1000 \
--rm \
--cidfile=/tmp/2_9lnoqd/20240612110121-963427.cid \
--env=TMPDIR=/tmp \
--env=HOME=/flvvYX \
biocontainers/clustalo:v1.2.4-2-deb_cv1 \
clustalo \
-i \
/var/lib/cwl/stgc42faa48-b345-4c6c-9c0c-6d0f6dd22db9/blastdbcmd_result.fasta \
--outfmt=fasta \
-o \
clustalo_output.fasta
WARNING: The requested image's platform (linux/amd64) does not match the detected host platform (linux/arm64/v8) and no specific platform was requested
INFO [job step4_clustalo] completed success
INFO [step step4_clustalo] completed success
INFO [workflow ] starting step step5_fasttree
INFO [step step5_fasttree] start
INFO [job step5_fasttree] /tmp/0s8ydzu6$ docker \
run \
-i \
--mount=type=bind,source=/tmp/0s8ydzu6,target=/flvvYX \
--mount=type=bind,source=/tmp/mlmjr61d,target=/tmp \
--mount=type=bind,source=/tmp/wdeje8er/clustalo_output.fasta,target=/var/lib/cwl/stgbe7e0678-8e28-4bfd-a1dc-758e986f9742/clustalo_output.fasta,readonly \
--workdir=/flvvYX \
--read-only=true \
--log-driver=none \
--user=1000:1000 \
--rm \
--cidfile=/tmp/aj65vozh/20240612110123-014278.cid \
--env=TMPDIR=/tmp \
--env=HOME=/flvvYX \
biocontainers/fasttree:v2.1.10-2-deb_cv1 \
fasttree \
-nt \
/var/lib/cwl/stgbe7e0678-8e28-4bfd-a1dc-758e986f9742/clustalo_output.fasta > /tmp/0s8ydzu6/fasttree_result.nwk
WARNING: The requested image's platform (linux/amd64) does not match the detected host platform (linux/arm64/v8) and no specific platform was requested
FastTree Version 2.1.10 Double precision (No SSE3)
Alignment: /var/lib/cwl/stgbe7e0678-8e28-4bfd-a1dc-758e986f9742/clustalo_output.fasta
Nucleotide distances: Jukes-Cantor Joins: balanced Support: SH-like 1000
Search: Normal +NNI +SPR (2 rounds range 10) +ML-NNI opt-each=1
TopHits: 1.00*sqrtN close=default refresh=0.80
ML Model: Jukes-Cantor, CAT approximation with 20 rate categories
Ignored unknown character D (seen 311 times)
Ignored unknown character E (seen 310 times)
Ignored unknown character F (seen 176 times)
Ignored unknown character H (seen 77 times)
Ignored unknown character I (seen 332 times)
Ignored unknown character K (seen 364 times)
Ignored unknown character L (seen 451 times)
Ignored unknown character M (seen 110 times)
Ignored unknown character P (seen 312 times)
Ignored unknown character Q (seen 232 times)
Ignored unknown character R (seen 237 times)
Ignored unknown character S (seen 278 times)
Ignored unknown character V (seen 279 times)
Ignored unknown character W (seen 78 times)
Ignored unknown character X (seen 205 times)
Ignored unknown character Y (seen 169 times)
WARNING! ONLY 19.6% NUCLEOTIDE CHARACTERS -- IS THIS REALLY A NUCLEOTIDE ALIGNMENT?
Initial topology in 0.01 seconds
Refining topology: 15 rounds ME-NNIs, 2 rounds ME-SPRs, 7 rounds ML-NNIs
Total branch-length 0.107 after 0.01 sec
ML-NNI round 1: LogLk = -167.434 NNIs 5 max delta 0.00 Time 0.02
Switched to using 20 rate categories (CAT approximation)
Rate categories were divided by 0.628 so that average rate = 1.0
CAT-based log-likelihoods may not be comparable across runs
Use -gamma for approximate but comparable Gamma(20) log-likelihoods
ML-NNI round 2: LogLk = -165.205 NNIs 3 max delta 0.00 Time 0.02
Turning off heuristics for final round of ML NNIs (converged)
ML-NNI round 3: LogLk = -165.204 NNIs 2 max delta 0.00 Time 0.02 (final)
Optimize all lengths: LogLk = -165.204 Time 0.03
Total time: 0.06 seconds Unique: 13/20 Bad splits: 0/10
INFO [job step5_fasttree] completed success
INFO [step step5_fasttree] completed success
INFO [workflow ] completed success
{
"awk_output": {
"location": "file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/workflow_result/blastp_result_id.txt",
"basename": "blastp_result_id.txt",
"class": "File",
"checksum": "sha1$354af893fc9afa558d0a01d6d68bf9d3a935cb53",
"size": 418,
"path": "/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/workflow_result/blastp_result_id.txt"
},
"blastdbcmd_output": {
"location": "file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/workflow_result/blastdbcmd_result.fasta",
"basename": "blastdbcmd_result.fasta",
"class": "File",
"checksum": "sha1$e9167fd8855ce938590f64e179b3aedb0edd9e15",
"size": 9581,
"path": "/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/workflow_result/blastdbcmd_result.fasta"
},
"blastp_output": {
"location": "file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/workflow_result/blastp_output.txt",
"basename": "blastp_output.txt",
"class": "File",
"checksum": "sha1$31464e2d0bdfd65f89e0542688844d3686bf278c",
"size": 1536,
"path": "/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/workflow_result/blastp_output.txt"
},
"clustalo_output": {
"location": "file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/workflow_result/clustalo_output.fasta",
"basename": "clustalo_output.fasta",
"class": "File",
"checksum": "sha1$57ec8d531af2e6a5a0add1f1fc8ff413a12d62f6",
"size": 9621,
"path": "/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/workflow_result/clustalo_output.fasta"
},
"fasttree_output": {
"location": "file:///workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/workflow_result/fasttree_result.nwk",
"basename": "fasttree_result.nwk",
"class": "File",
"checksum": "sha1$90a12b35e23309a1e73fac58d05794beeab49a38",
"size": 807,
"path": "/workspaces/togotv_cwl_for_remote_container/zatsu_cwl_bioinformatics/workflow_result/fasttree_result.nwk"
}
}INFO Final process status is success
無事、全ての処理が終了しているようです! それでは、実際に出力されたファイルを見てみましょう。
無事、出力されています。このように、ワークフローをCWLで記述することで、一つのコマンドで複数の処理を一度に実行することができます。
終わりに
今回は、バイオインフォマティクス分野で使用されるツールをCWLファイルとして記述し、ワークフローまで実行するプロセスを記述しました。
zatsu-cwl-generatorを使うことで、簡単にCWLファイルを生成することができるため、CWLを記述するハードルを下げることができます。
これまでの記事で説明しているプロセスとしては、まとめると以下のようになっています。
- zatsu-cwl-generatorを使って、コマンドラインツールの処理をCWLファイルとして生成する
- cwltoolの
--validateオプションを使って、CWLファイルをチェックする - 問題がある場合、修正を行う(ドキュメントを読む、など)
- cwltoolを使って、CWLファイルを実行する(
--debugオプションをつけておく) - エラーが出た場合、修正を行う
このように、 zatsu-cwl-generator をCWLファイルを書くプロセスの起点として利用することが可能です。
ぜひ、皆さんも活用して素晴らしいCWLライフを満喫してください!
参考
これまでの3つの記事では詳しい部分についてはあまり解説していません。
CWLの詳細な記述方法については、以下のようなドキュメントが充実しているので、ぜひそちらを参考にしてください。
参考:Dockerでコマンド実行する例
# Step1
docker run --rm -it -v `pwd`:`pwd` -w `pwd` biocontainers/blast:v2.2.31_cv2 blastp -query MSTN.fasta -db uniprot_sprot.fasta -evalue 1e-5 -num_threads 4 -outfmt 6 -out blastp_result_MSTN.txt -max_target_seqs 20
# Step2
docker run --rm -it -v `pwd`:`pwd` -w `pwd` ubuntu:23.10 awk '{ print $2 }' `pwd`/data/blastp_result.txt > `pwd`/data/blastp_result_id.txt
#Step3
docker run --rm -it -v `pwd`:`pwd` -w `pwd` biocontainers/blast:v2.2.31_cv2 blastdbcmd -db uniprot_sprot.fasta -entry_batch blastp_result_id.txt -out blastdbcmd_result.fasta
#Step4
docker run --rm -it -v `pwd`:`pwd` -w `pwd` biocontainers/clustalo:v1.2.4-2-deb_cv1 clustalo -i `pwd`/blastp_results_MSTN.fasta --outfmt=fasta -o clustalo_result.fasta
#Step5
docker run --rm -it -v `pwd`:`pwd` -w `pwd` biocontainers/fasttree:v2.1.10-2-deb_cv1 FastTree -boot 100 -out MSTN_tree.newick `pwd`/clustalo_result.fasta
今回の記事で使用したCWLのファイルをおいているリポジトリは以下からアクセスすることができます。
Discussion