GenePatternをlocalに導入(docker使用)
経緯
VarScan2でCNV検出して、その結果をGISTIC2でplotしようとWSL2にインストール(anacondaサイトから取得できた mamba install -c hcc gistic2)するも実行時にPython関連のエラーがでるので、色々調べているとGISTIC2の使い方を紹介しているyoutubeに辿り着いた。
その中で GenePattern というWebツールが使われており、フリーでコマンド不要でできる解析の多さに驚かされた。

ゲノムへのマッピングツール、発現変動解析、シングルセル解析、生存時間解析、フローサイトメトリー、作図などなど多岐にわたったModuleが200以上用意されている。
Web serverで解析
Web serverで解析を行う場合は、Run > Public Serverを選択する。

Webページの左上
ユーザー登録が必要なので、初回はClick to Registerから登録しておく。

ログイン画面
(解析例)GISTIC2
(解析例)GISTIC2
解析の一例としてyoutubeで紹介されていた流れをやってみる。
inputの用意
デモデータとして cBioportalからTCGAの肝臓がんデータを使用する。
https://www.cbioportal.org/study/summary?id=lihc_tcga_pan_can_atlas_2018
開いたページの上部の小さなダウンロードボタンからデータ一式を取得する。

ダウンロードして解凍すると「data_cna_hg19.seg」というファイルが確認できるはず。こちらを使用する。
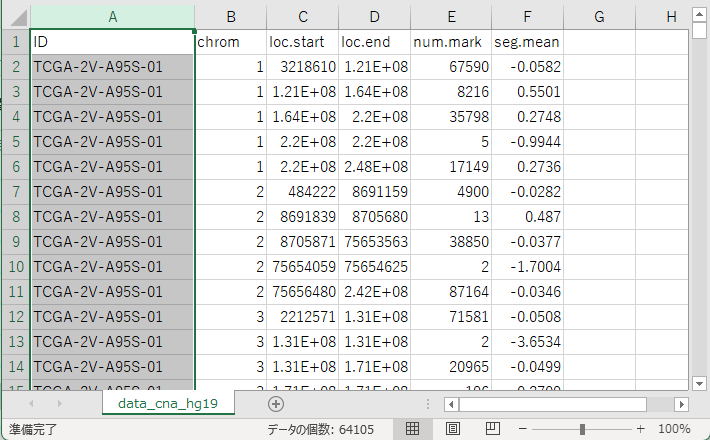
Excelで確認すると、64105行、6列のデータとなっていた。ID列はサンプル名で、複数のサンプルのCNVデータが1つのファイルにまとまっている。(GISTIC2では1サンプルのデータではなく、複数サンプルのデータを使う。)
Moduleの選択
まずは必要なModuleを選択する。左側パネルのModulesダブでModuleを検索するか、Browse Modulesからカテゴリーごとに分けられたModuleを探す。Module名がわかっているときは検索した方が早い。

Browse Modulesの画面
実行
GISTIC_2.0を選択すると、次の画面に遷移する。

GISTIC_2.0の設定画面
アスタリスクがついている箇所が最低限の設定項目である。
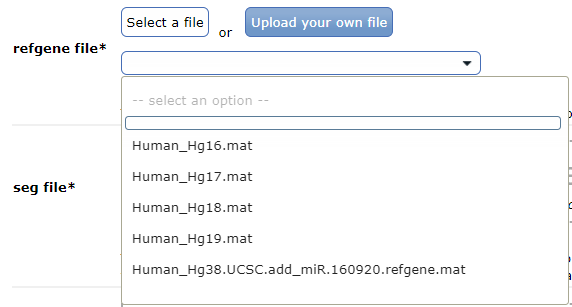
最低限必要なファイルとして、参照ゲノムデータがあるが、こちらはWeb serverに用意されている。
seg fileのファイル名からhg19にマッピングしていることがわかるので、refgene fileからは「Human_Hg19.mat」を選択した。
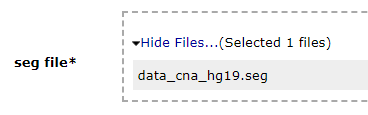
seg fileの箇所には、cBioportalから取得したファイルをドラッグ&ドロップでUploadする。
ページ右上か右下にあるRunを押すとプロセスが進む。

プロセス画面に遷移するので、解析が終了するまで放置しておく。Email Reminderにチェックを入れると登録していたメールアドレスにJob完了の通知をくれる。

Job開始画面
終了すると、出力の一覧が表示される。
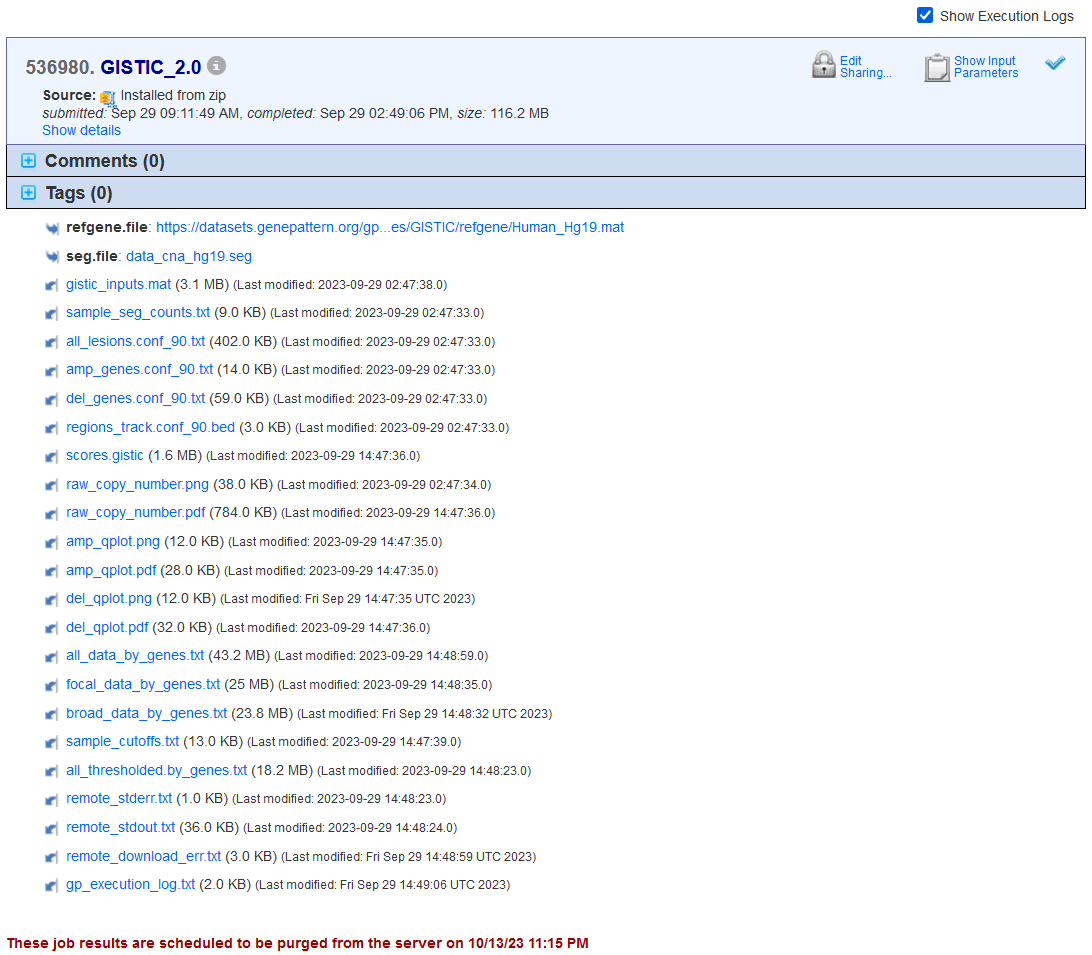
終了画面
出力ファイル1つ1つがリンクになっており、クリックすると個別にダウンロードできる。一括でダウンロードする場合は、「Job番号. Module名」(ここでは「536980. GISTIC_2.0」)のModule名のリンクをクリックすると、Jobに対する操作メニューが開くので、Download Jobを選択する。

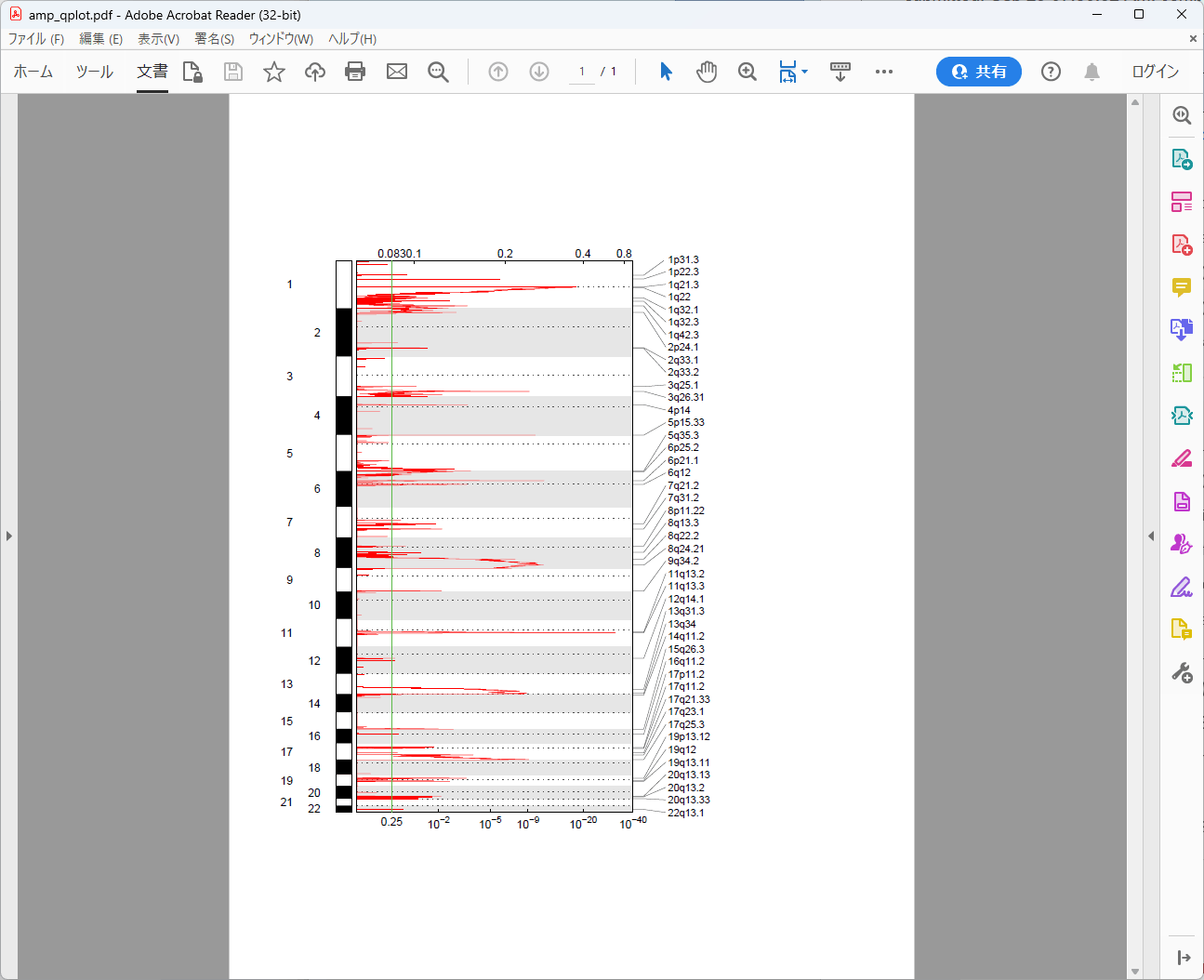
無事、CNVのplotが得られた。
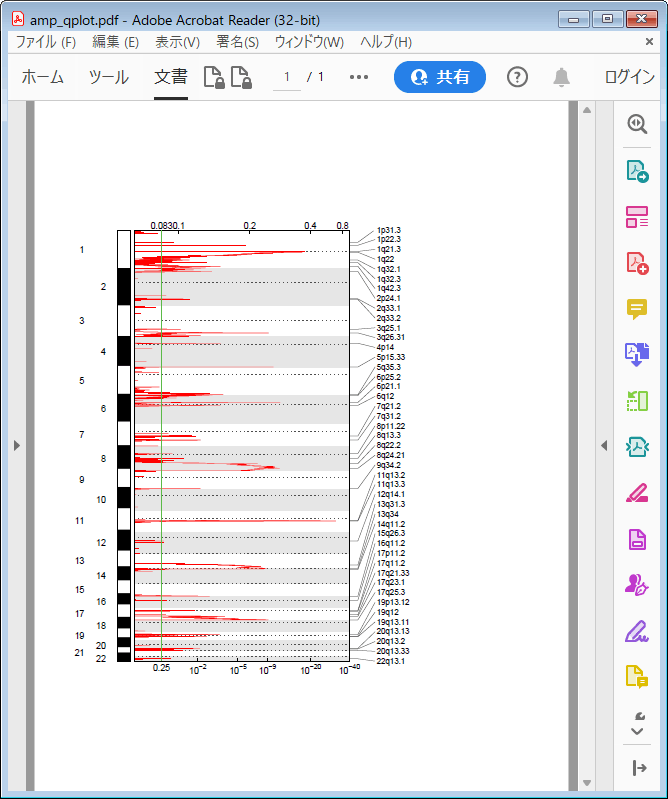
dockerでGenePattern serverを立ち上げる
ここからが本題。
バイオインフォマティクスでは巨大なデータを扱うことも多く、アップロードに時間がかかったり、向こうのserver状態に解析が左右されたりしたくないので、本書ではGenePatternをローカル環境に導入する方法を紹介する。(実際、Public serverで解析するよりも早かった。)
こちらが参考ページ。OSごとに異なるようだ。
本記事ではWSL2のdocker環境を使用した。環境は以下の記事で作成したもの。
初期設定
1. Githubページから「start-genepattern.sh」をダウンロードする。
https://github.com/genepattern/genepattern-server/blob/develop/docker/start-genepattern.sh
こちらに初期のディレクトリ構築やdockerコマンドなどが書かれており、シェルスクリプトを実行することでdocker imageを取得する。(シェルスクリプト内でdocker runなどが行われる。)
2. docker imageのTag情報
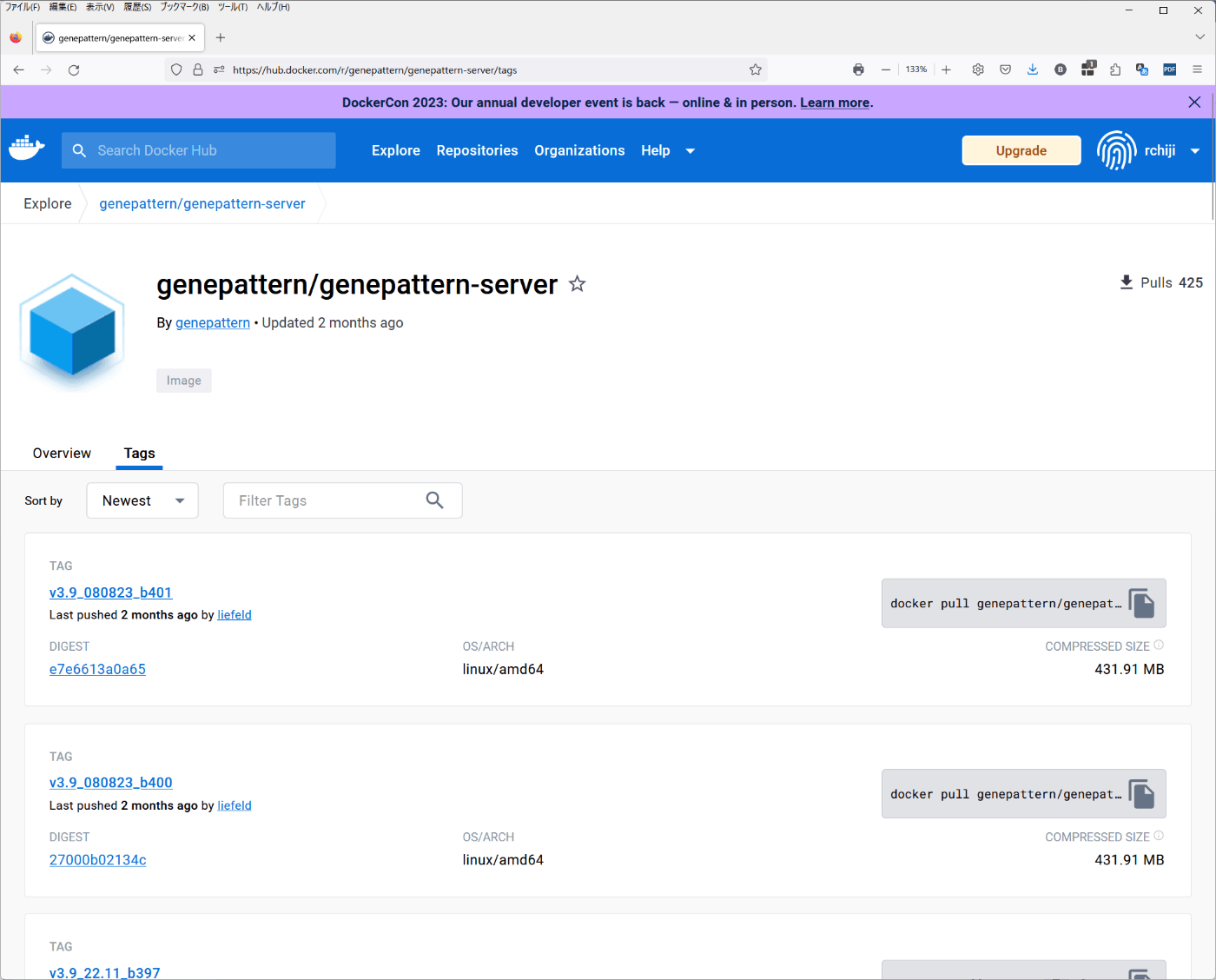
シェルスクリプト実行の際にdocker imageのTag情報を使用する。次のリンクから欲しいversionのTAG名を控えておく。本記事では最新の「v3.9_080823_b401」を使用する。
https://hub.docker.com/r/genepattern/genepattern-server/tags
3. シェルスクリプト実行
シェルスクリプト実行時に幾つかの初期ディレクトリが作られる。
ホームディレクトリに作られないように、「start-genepattern.sh」を任意のフォルダに移動させておく。本記事では ~/tools/GenePattern ディレクトリを作成し、そこに「start-genepattern.sh」を配置した。
さらにカレントディレクトリを ~/tools/GenePattern に移動しておいた。
cd ~/tools/GenePattern
次のようにシェルスクリプトに実行権限を与えて実行する。-vオプションに上記のTag情報を指定する。
chmod +x start-genepattern.sh
./start-genepattern.sh -v v3.9_080823_b401
私の場合は、途中でWindowsセキュリティのウィンドウが立ち上がったが、許可して進めた。

このように終了。

カレントディレクトリに4つのディレクトリが構築されていた。
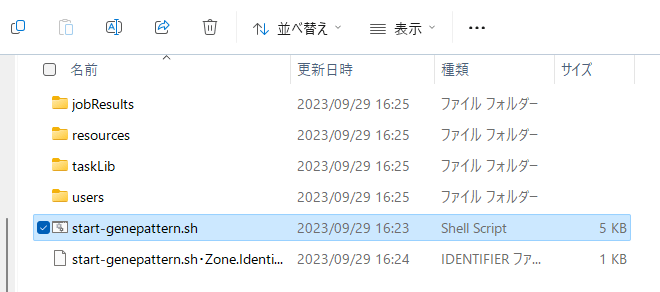
dockerコンテナが起動した状態なのがdocker psから確認できるはず。
4. ブラウザでアクセス
dockerコンテナが起動した状態で、ウェブブラウザから「 http://127.0.0.1:8080/gp 」にアクセスする。
初回はSign in画面が出てくるが、Click to Registerからユーザー名を登録するだけで使えるようになる。パスワードはない。
Public serverと同様の画面が立ち上がる。
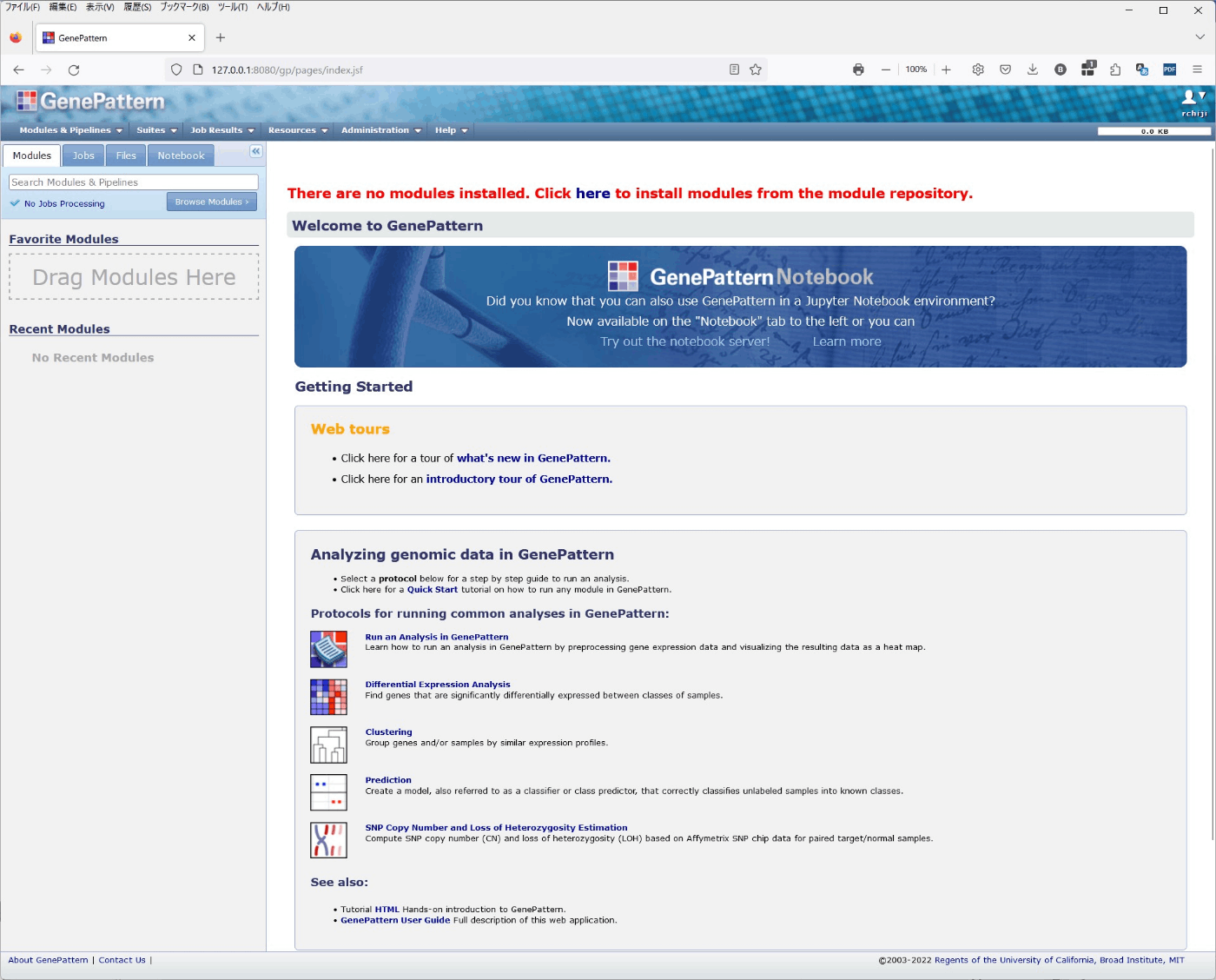
5. Moduleのインストール
初めはModuleが何も入っていない状態のGenePatternになっている。

Modules & PipelinesのメニューからModuleをインストールする。
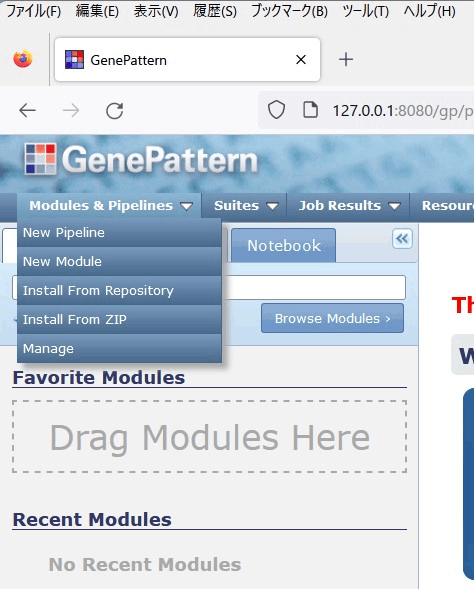
ここではInstall from Repositryを選択した。次のようにインストールできるものはチェックが入った状態になっている。
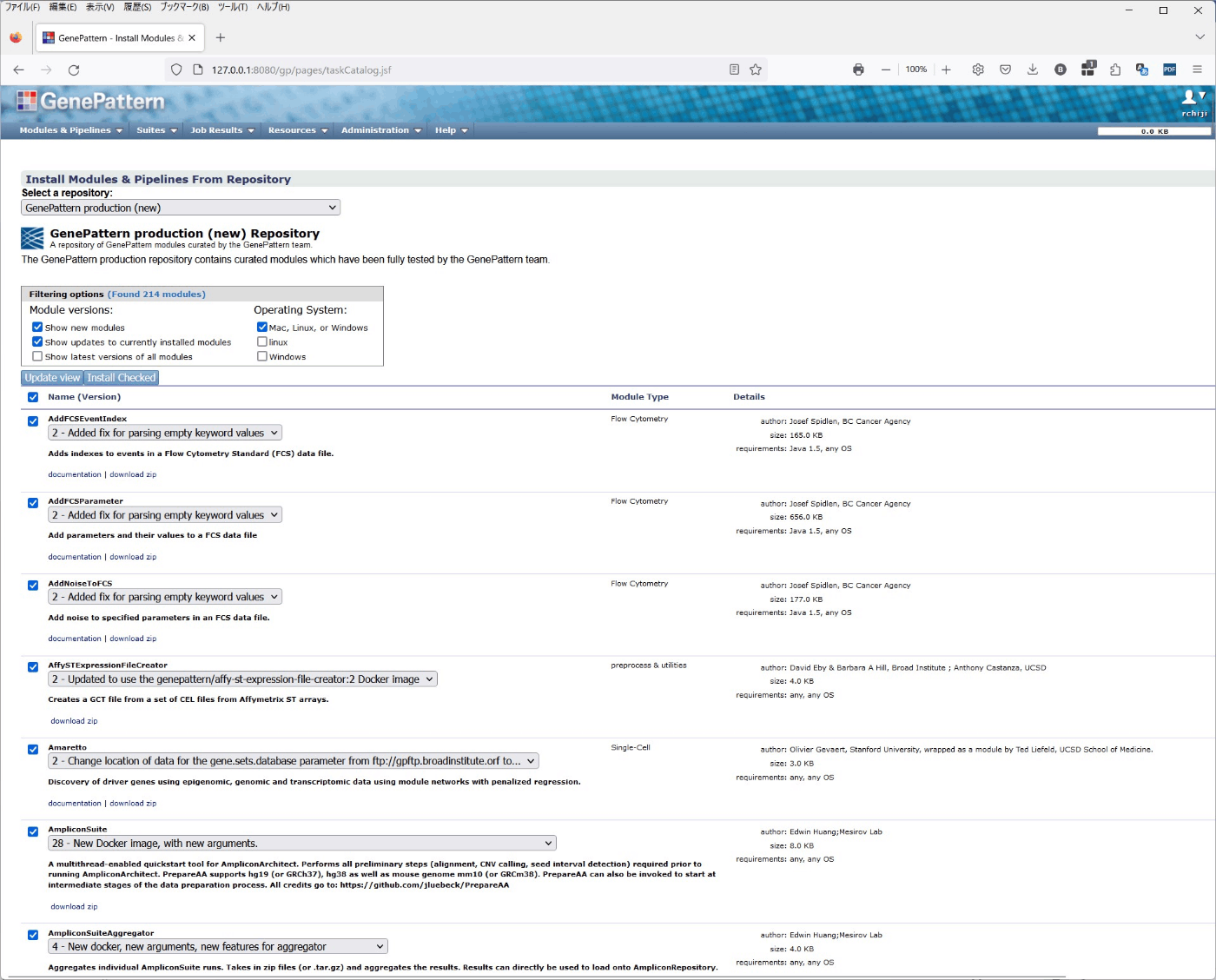
Install Checkedからインストールする。
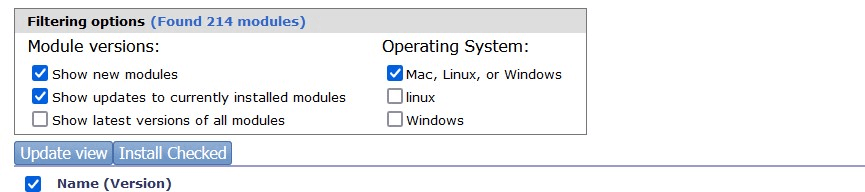
インストールできるものとできないものがある。Rの古いversionに依存しているものなどは基本的にインストールできなかった。
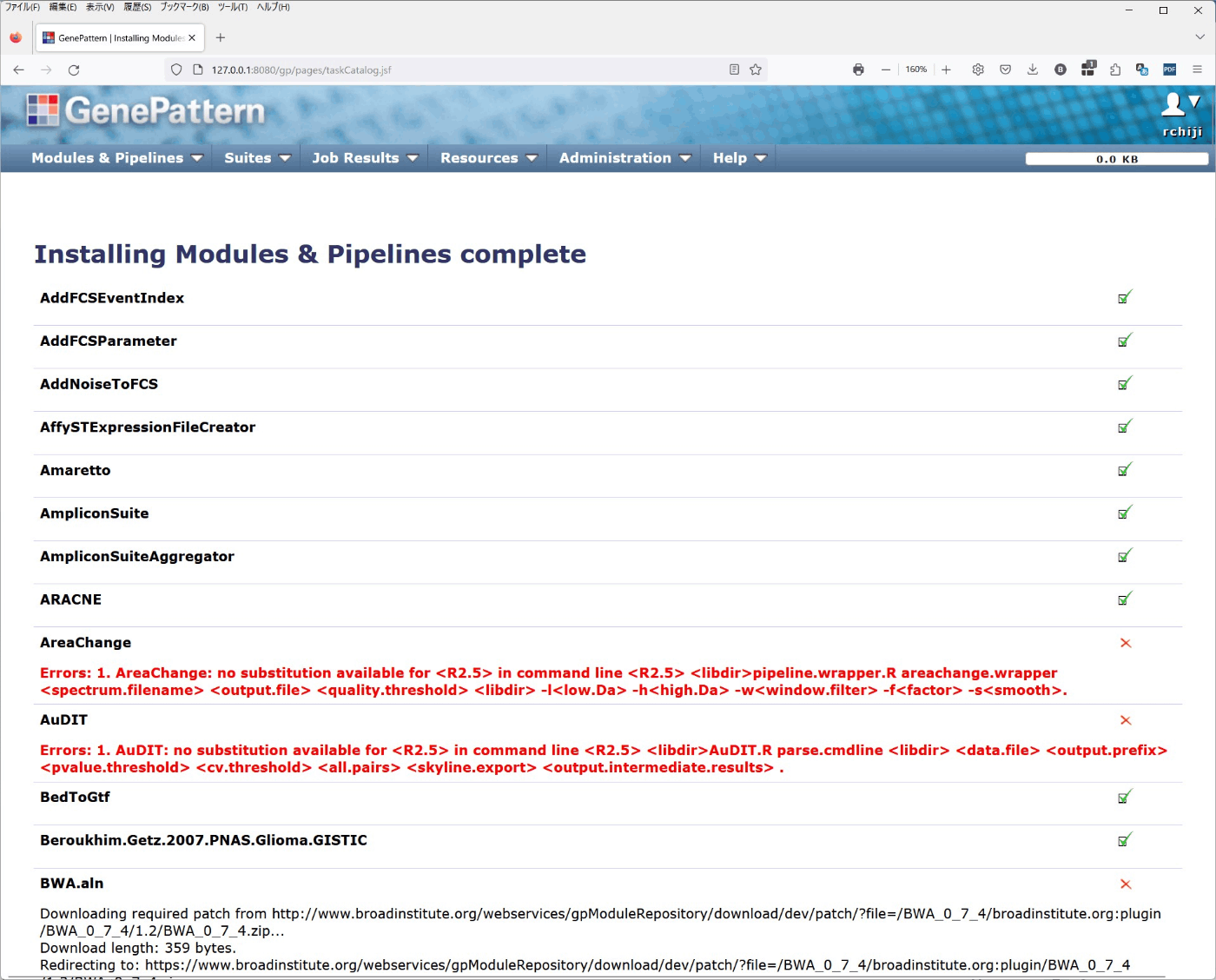
GISTIC_2.0はインストールできたのでとりあえず良しとする。
インストールが終わると、ページ下部のDoneから元のページに戻る。
ModulesからModuleが確認できるようになる。
(解析例)GISTIC2
(解析例)GISTIC2
デモデータで同様の解析を進める。
不安だったが、ローカルのGenePatternでもrefgene fileは同様のものが選択できた。
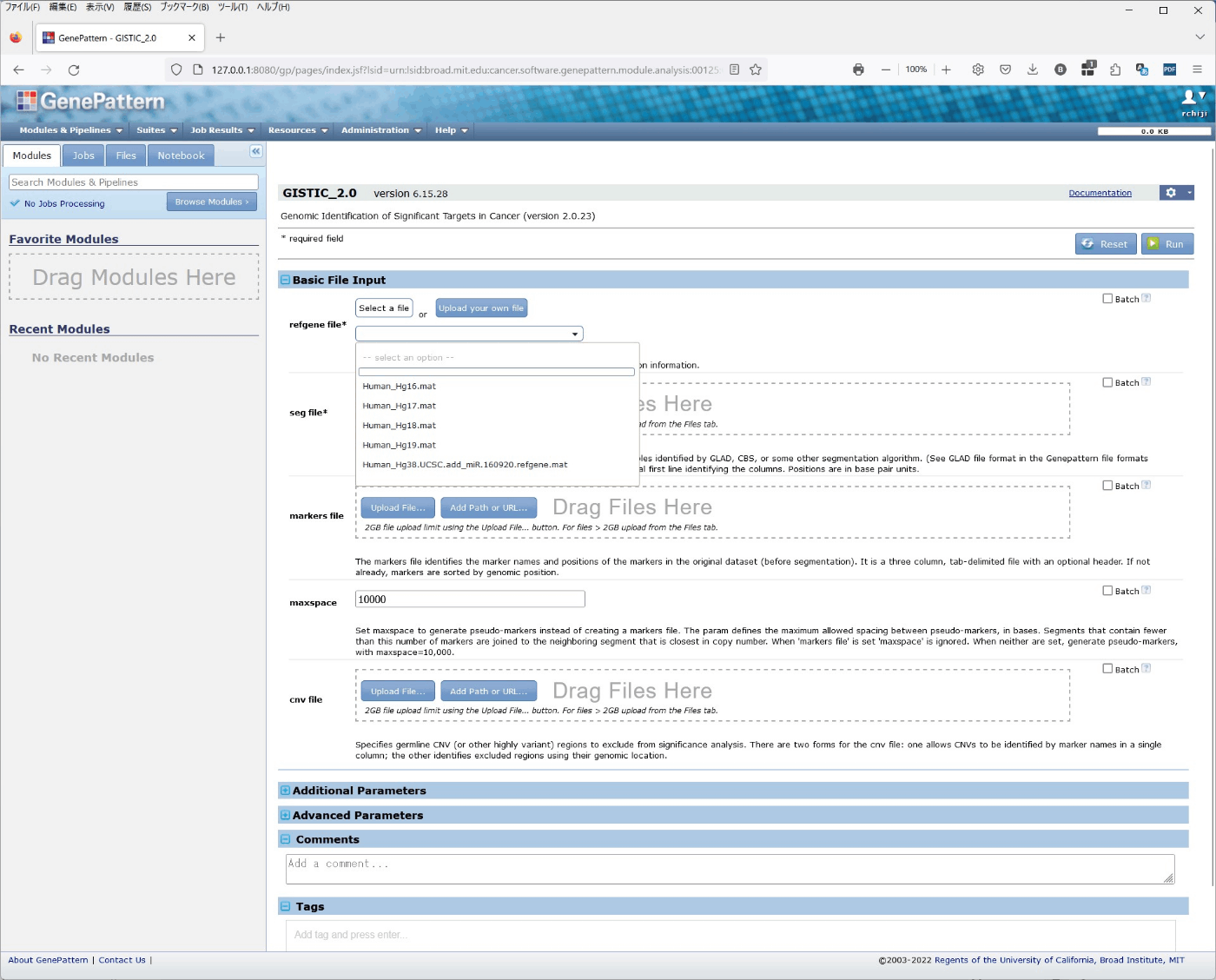
この条件でRun

プロセス画面

ちゃんと終了。

Public Serverで解析したものと同じ出力が得られた。
2回目以降の立ち上げ
GenePatternを再度立ち上げる時も「start-genepattern.sh」を実行する。
cd tools/GenePattern/
./start-genepattern.sh
Ubuntuの画面には次のように出力されるが、ウェブブラウザから「 http://127.0.0.1:8080/gp 」にアクセスすれば使用できる。

Module類もそのまま使用できるし、過去のJobもアクセスできる。

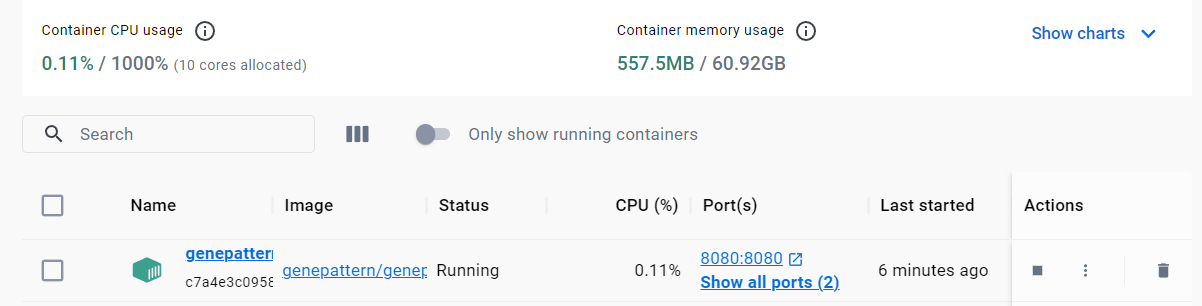
もしくはDocker desktopのActionsからStart/Stopを操作するだけでも使用できる。Start状態で http://127.0.0.1:8080/gp 」にアクセスすればいいだけ。
Docker desktopから立ち上げる場合


Discussion