【WSL】[bioinfomatics] Windows11にWSL環境を整える(2)
前回↓↓
【Anaconda】
Anacondaは (1) データサイエンスのためのツールの管理 (2) Python仮想環境作成 が行えるプラットフォームである。
Anacondaで公開されているツールであればcondaコマンドでツールをインストールして使うことができるようになる。バイオインフォマティクスツールの多くもAnacondaで公開されている。
(バイオインフォでの利用ではひとまずツールをダウンロードするためのツールぐらいの認識で良いと思う。)
インストール
Anacondaの公式サイトのページ下部 Anaconda InstallersからLinux版をダウンロードする。(トップページのDownloadのリンクからはWindows版のインストーラーがダウンロードされてしまうので注意)

64-Bit (x86) Installer (860 MB)をインストールすることにする。
ブラウザのダウンロード機能を使ってもよいが折角なのでUbuntuのwgetコマンドでダウンロードしてみる。
wget https://repo.anaconda.com/archive/Anaconda3-2023.03-Linux-x86_64.sh

wgetの画面
wgetを使えばホームディレクトリにダウンロードされているため以後の処理がやりやすい。

sh拡張子のインストーラーをshコマンドで実行する。
sh Anaconda3-2023.03-Linux-x86_64.sh

Enterを押し続けて誓約書的なものをすべて表示させるとyesかnoか打ち込むように言われるのでyes。その後もインストール先を聞かれたりとあるが、デフォルトの設定のままでEnterやyesをしていく。
途中で以下のメッセージが出るのでyesと打っておくと、.bashrcにanacondaへのパスが通るように追記される。
Do you wish the installer to initialize Anaconda3
by running conda init? [yes|no]
.bashrcを開いてみて追記がされていないようであれば、以下を追記しておくとよい。※ /home/rchijiの箇所は自分の環境に合わせる。
# >>> conda initialize >>>
# !! Contents within this block are managed by 'conda init' !!
__conda_setup="$('/home/rchiji/anaconda3/bin/conda' 'shell.bash' 'hook' 2> /dev/null)"
if [ $? -eq 0 ]; then
eval "$__conda_setup"
else
if [ -f "/home/rchiji/anaconda3/etc/profile.d/conda.sh" ]; then
. "/home/rchiji/anaconda3/etc/profile.d/conda.sh"
else
export PATH="/home/rchiji/anaconda3/bin:$PATH"
fi
fi
unset __conda_setup
# <<< conda initialize <<<
Ubuntuを立ち上げなおしてwhich condaでcondaコマンドへのパスが表示されれば成功である。

echo $PATHで通っているパスが確認できる。以下のようにanaconda3/binが表示されない場合は手動でパスを通さなければならない。
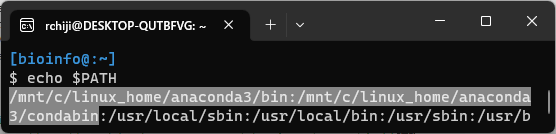
パスを通すと検索すれば沢山情報が出てくるのでここでは解説しないが、上記の.profile (.bash_profile)や.bashrcに以下のように追記しておけばよい。(/home/rchiji/の箇所は自分のものに置き換える。)
export PATH="/home/rchiji/anaconda3/bin:$PATH"
conda installコマンドでツールをインストール
試しにconda installでfastqcをインストールしてみる。Anacondaで管理されているツールはインストールコマンドも紹介してくれているので、それをコピぺで使えばよい。

conda install -c bioconda fastqc
conda installでインストールするとanaconda3/binフォルダの中にツールの実行ファイルが入るため追加でパスを通したりする必要はない。
which fastqc
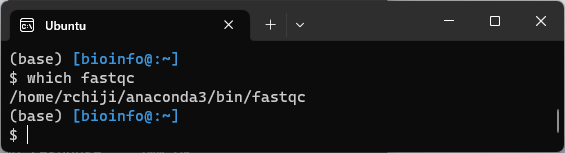
condaを介さずにどこかしらかソースコードを取ってきて使用するのであれば、そのファイルの保存先にパスを通す必要が出てくる。
IGV
WSL2になりGUIアプリが扱いやすくなったため、ゲノムブラウザのIGVもUbuntuから立ち上げるのに苦労しなくなった。
# igv v2.13.2
conda install -c bioconda igv -y
igvと打つだけでIGVのウィンドウが立ち上がる。
igv
【Mamba】
condaよりも高速で安定に動作するmambaを使ったインストールも有用である。
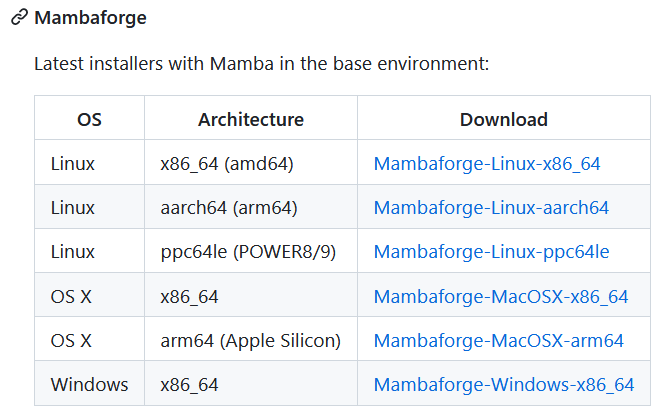
Mambaforge distribution
まずはMambaforge distributionをインストールする。
(conda install -c conda-forge mambaを使ってmambaを導入することもできるようだが、公式ドキュメントでは推奨されていない。)
Mambaforge distributionのリンクからGithubページへ移動し、自身の環境にあったインストーラーをダウンロードする。
私の場合は x86_64 (amd64) のものをダウンロードした。
wget https://github.com/conda-forge/miniforge/releases/latest/download/Mambaforge-Linux-x86_64.sh
shコマンドでインストーラーを実行する。
sh Mambaforge-Linux-x86_64.sh
Enterを押しながら規約を進めて、途中の Do you accept the license terms? [yes|no]が出てきたらyesと打ち込んでEnter。(Enterを押しっぱなしにするとnoになってインストールできないかも)

その後関連するツール類のインストールが進み、Do you wish the installer to initialize Mambaforgeと聞いてくるのでyesと打つ。
.bashrcに書き込まれる内容
yesとすると、以下の内容が .bashrc に書き込まれる。Ubuntuを立ち上げる際に、anacondaではなくmambaforgeが自動で立ち上がるように変わっている。
noとしてしまった場合は、以下を .bashrc に書き込めばよい。
# >>> conda initialize >>>
# !! Contents within this block are managed by 'conda init' !!
__conda_setup="$('/home/rchiji/mambaforge/bin/conda' 'shell.bash' 'hook' 2> /dev/null)"
if [ $? -eq 0 ]; then
eval "$__conda_setup"
else
if [ -f "/home/rchiji/mambaforge/etc/profile.d/conda.sh" ]; then
. "/home/rchiji/mambaforge/etc/profile.d/conda.sh"
else
export PATH="/home/rchiji/mambaforge/bin:$PATH"
fi
fi
unset __conda_setup
if [ -f "/home/rchiji/mambaforge/etc/profile.d/mamba.sh" ]; then
. "/home/rchiji/mambaforge/etc/profile.d/mamba.sh"
fi
# <<< conda initialize <<<
anacondaも併用する場合
Mambaのロゴが表示されて、インストールが終了。

Ubuntuを立ち上げなおすと、mambaコマンドが使えるようになる。
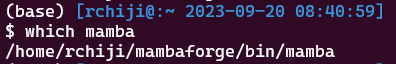
mambaコマンドでインストール
conda install ...の condaをmambaに変えるだけでよい。
試しにRSEMをインストールすると、conda installよりもインストール過程が非常に分かりやすくて良心的だ。
mamba install -c bioconda rsem

versionもv1.3.1がインストールされている。

他のツールもmambaでインストールできることが確認できた。
mamba install -c bioconda fastqc -y
mamba install -c bioconda fastp -y
mamba install -c bioconda star -y
mamba install -c bioconda samtools -y

condaでインストール済みのものをmambaでインストールし直した場合の注意点
condaコマンドでインストールしたツールは anaconda3/bin に実行ファイルが存在し、mambaコマンドでインストールしたツールは mambaforge/bin や mambaforge/condabin にファイルが存在する。
どちらにも実行ファイルがある場合は、パスの優先順位が先のものから使用される。
通っているパスを確認し、anaconda3/bin の方が先に表示されるなら condaコマンドでインストールしたツールが優先的に使用されてしまう。
echo $PATH

パスは後から記載したものが優先される。
Ubuntuの立ち上げ時に .profile (.bash_profile)が読み込まれ、.profile内の最初に.bashrcを読みに行って、.profileの残りの項目を読み込むので、export PATH="/home/rchiji/anaconda3/bin:$PATH"が mambaへのパスを通すコマンドより後に来ていないかを確認し、後に来ている場合は、コマンドの順番を変えておく。
私の場合は、.bashrcを次のように anaconad3/bin へパスを通す -> conda initializeでmambaforge/bin へパスを通すという順番に変更した。

.bashrc
パスを再確認。mambaforge/bin が先に来ている。

【R】
Windows版Rでは並列実行パッケージが使えないものがあるが、WSLでRを使えばそれは解決できる。
sudo apt install r-base

R

Rのversion 4.1.2がインストールされていた。
Rの最新版をインストール
CRANのページからUbuntuを選択すると、Ubuntuに最新のRを入れるコマンドが出てくる。
sudo apt update
sudo apt upgrade
sudo apt install --no-install-recommends software-properties-common dirmngr
wget -qO- https://cloud.r-project.org/bin/linux/ubuntu/marutter_pubkey.asc | sudo tee -a /etc/apt/trusted.gpg.d/cran_ubuntu_key.asc
sudo add-apt-repository "deb https://cloud.r-project.org/bin/linux/ubuntu $(lsb_release -cs)-cran40/"
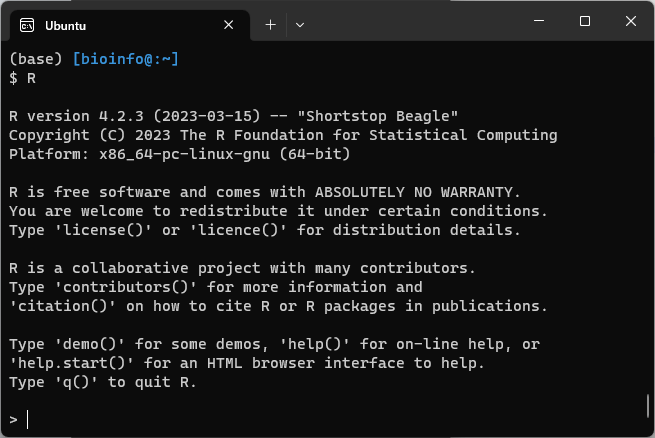
R
version 4.2.3のRがインストールされた。
【RStudio server】
Rを使うならやはりRStudioが使えないと不便である。WSLでもRStudioが使える。
ブラウザを介して使用するRStudio server版のインストールを行う。(GUIアプリ版もあるのだが、日本語を使えるようにするのが難しかったのであきらめた。)
【参考】
https://support.posit.co/hc/en-us/articles/360049776974-Using-RStudio-Server-in-Windows-WSL2
http://www.psy.ritsumei.ac.jp/~hoshino/Wsl/
# インストールに必要なツールを入れておく
sudo apt install gdebi-core
Rstudio-serverのサイトからインストーラーをダウンロードする。
自身のディストリビューションを選択。

下にスクロールするとwgetコマンドが得られる。
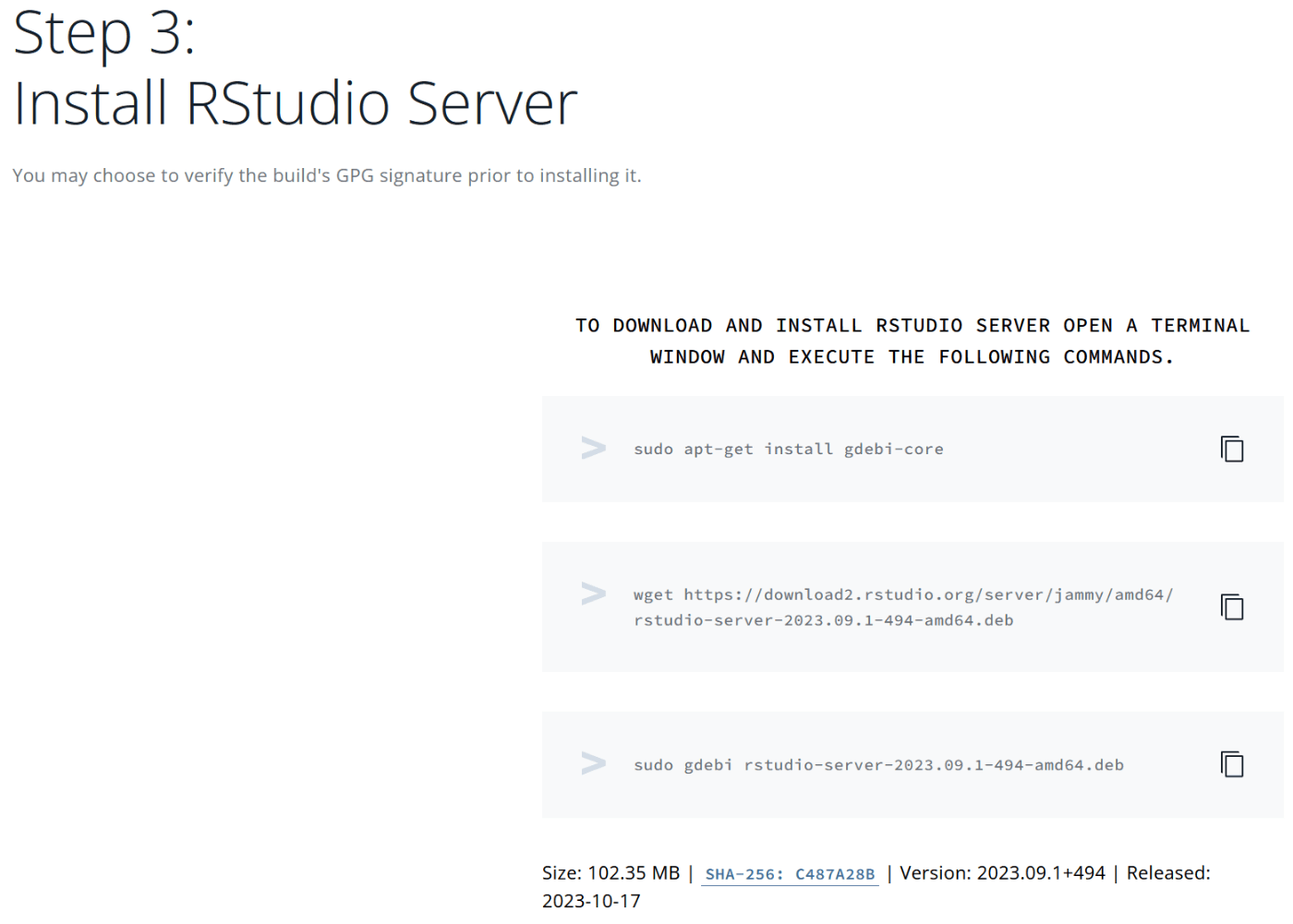
# インストーラーのダウンロード
wget https://download2.rstudio.org/server/jammy/amd64/rstudio-server-2023.09.1-494-amd64.deb
sudo gdebiコマンドでインストール。自身の環境にダウンロードされたファイルを指定。
# RStudio serverのインストール
sudo gdebi rstudio-server-〇〇-amd64.deb
成功

RStudio serverの開始
sudo rstudio-server start
何かウィンドウが開くわけでもないが、実際にはブラウザで http://localhost:8787 にアクセスすると使えるようになっている。

ブラウザでアクセスするとサインイン画面が出てくる。このUsernameとPasswordはUbuntuインストール時に設定したものと同じ。

サインインするとソフトウェア版のRStudioと同様の画面が立ち上がる。操作方法も同じ。

終了する場合はWSL2で次のように打ち込む
sudo rstudio-server stop
サインイン画面に戻る時間を変更
デフォルトでは60分放置すると毎回サインイン画面に戻るので面倒である。rserver.confファイルに設定を書き込めばいいとのこと。
sudo vim /etc/rstudio/rserver.conf
以下を書き込む。vimでrserver.confが開かれたら、Insertキー -> 書き込む -> Escキー -> :wqで編集できる。この例では 72時間 * 60分 = 4320を入れた。
auth-timeout-minutes=4320

catで中身を確認
【Docker】
「参考」
https://zenn.dev/fehde/articles/ea0e8a0a0a1de4
https://qiita.com/ucan-lab/items/62c3ce7ecfc475ccd1a8
- 公式ページの「Docker Desktop Installer.exe」のリンクからインストーラをダウンロード。
-
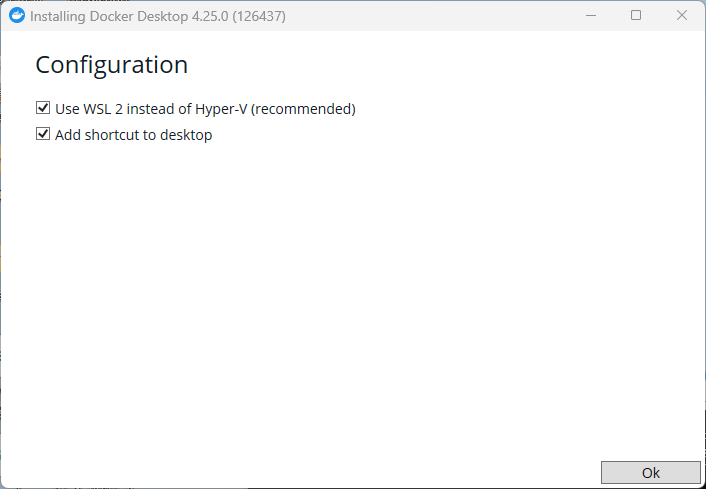
インストーラを実行。デフォルト設定で進める。
-
「Close and restart」を押して再起動

再起動後、勝手にインストールのウィンドウが立ち上がるので、デフォルト設定で進める。
-
Docker Desktopでサインイン
-
「上部タブの設定マーク」 > 「Resources」 > 「WSL Integration」から使用するディストリビューションをオンにして「Apply & restart」
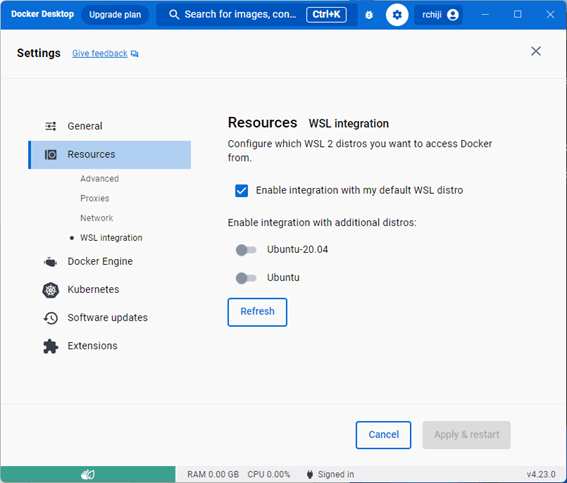
画像は初期画面。どちらもオフになっていた。 -
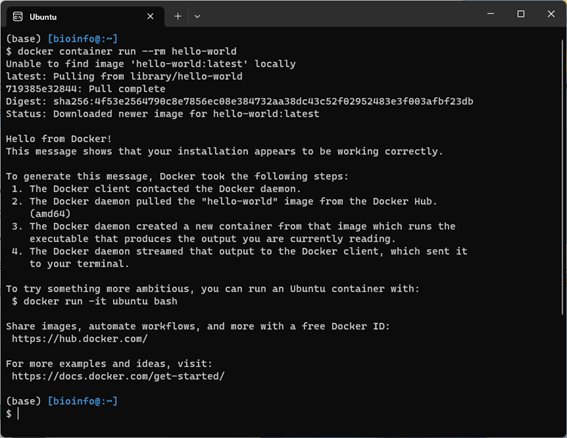
Ubuntuを立ち上げなおして確認すると進んだ
docker container run --rm hello-world
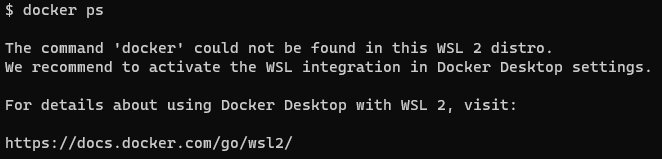
dockerが使えなくなるとき
dockerを使おうとすると次のようなメッセージが出ることがある。
上記の方法は、Docker desktopに依存しているので、docker desktopを立ち上げてdocker engineを作動させるとWSL2からもdockerコマンドが使えるようになる。
Docker desktopは一度立ち上げた後は、閉じても大丈夫。ただ立ち上げていると作業中のdockerのモニタリングもできるので便利。
Discussion