【シングルセル解析】[Seurat] 細胞の抽出 subset()の例
Seuratでのシングルセル解析で得られた細胞データで大まかに解析したあとは、特定の細胞集団を抜き出してより詳細な解析を行うことが多い。Seurat objectからはindex操作かsubset()関数で細胞の抽出ができる。細かなtipsがあるのでここにまとめておく。
目次
デモデータ
データの用意
Seuratのチュートリアルページから取得できるPBMC 2700細胞のデータを使用。
チュートリアルと同様に処理を進める。デモ用にcluster番号の情報と細胞名の情報をmeta.dataスロットに保存している。
library(Seurat)
library(dplyr)
pbmc.data <- Read10X(data.dir = "pbmc3k_filtered_gene_bc_matrices/filtered_gene_bc_matrices/hg19")
pbmc <- CreateSeuratObject(counts = pbmc.data, project = "pbmc3k", min.cells = 3, min.features = 200)
pbmc[["percent.mt"]] <- PercentageFeatureSet(pbmc, pattern = "^MT-")
pbmc <- subset(pbmc, subset = nFeature_RNA > 200 & nFeature_RNA < 2500 & percent.mt < 5)
pbmc <- pbmc %>%
NormalizeData() %>%
FindVariableFeatures() %>%
ScaleData() %>%
RunPCA() %>%
RunUMAP(dims = 1:10) %>%
FindNeighbors(dims = 1:10) %>%
FindClusters(resolution = 0.5)
pbmc$Cluster <- pbmc@active.ident
DimPlot(pbmc, reduction = "umap", label = TRUE, pt.size = 0.5) + NoLegend()

new.cluster.ids <- c("Naive CD4 T", "CD14+ Mono", "Memory CD4 T", "B", "CD8 T", "FCGR3A+ Mono",
"NK", "DC", "Platelet")
names(new.cluster.ids) <- levels(pbmc)
pbmc <- RenameIdents(pbmc, new.cluster.ids)
pbmc$Cell_type <- pbmc@active.ident
DimPlot(pbmc, reduction = "umap", label = TRUE, pt.size = 0.5, repel = T) + NoLegend()

active.identの情報で抜き出す
active.identはseurat_object@active.identに保存されいてる情報で、現在採用しているクラスター情報を一時的に保存している。
plot機能各種やFindMarkers(), AverageExpression()などの機能も明示しなければactive.identの情報を使用する。
active.identは以下のようにmeta.dataスロットの列名を指定するだけで変更できる。
Idents(pbmc) <- "Cluster"
pbmc <- SetIdent(pbmc, value = "Cluster")
pbmc@active.ident <- pbmc@meta.data$Cluster
active.identの情報をもとに細胞を抽出するには、idents=引数を使用する。
subset()の戻り値はSeuratオブジェクトである。
active.identがcluster番号の場合
pbmc2 <- subset(pbmc, idents = 0)
DimPlot(pbmc2)

cluster番号の時はクオーテーションがあってもなくても良い
pbmc2 <- subset(pbmc, idents = "1")
DimPlot(pbmc2)

cluster番号のベクトルを使って複数クラスターを同時に抽出できる。
pbmc2 <- subset(pbmc, idents = c(2,3,4))
DimPlot(pbmc2)

active.identが細胞名などの文字の場合
Idents(pbmc) <- "Cell_type"
文字列の場合はクオーテーションをつけて指定する。こちらも複数一度に抜き出すことができる。
pbmc2 <- subset(pbmc, idents = c("Naive CD4 T","B"))
DimPlot(pbmc2)

指定以外を抽出 invert
invert=引数をTRUEにすると、指定以外の細胞を抜き出すことができる。
pbmc2 <- subset(pbmc, idents = c("Naive CD4 T","B"), invert = TRUE)
DimPlot(pbmc2)

meta.data列で抜き出す
active.identではなくmeta.data列を使って細胞抽出するにはsubset()機能の中でsubset=引数を使う。
subset(seurat_object, subset = meta.data列名 == "欲しい因子")のように書く。
列名はクオーテーション無しで指定する。
pbmc2 <- subset(pbmc, subset = Cluster == "1")
DimPlot(pbmc2)

発現量で抜き出す
特定の遺伝子発現に基づいて細胞を抽出することも可能。この際も遺伝子名にはクオーテーションはつけない。
pbmc2 <- subset(pbmc, subset = CD4 > 1)
DimPlot(pbmc2)

複数条件を組むこともできる。
pbmc2 <- subset(pbmc, subset = CD3E>1 & CD4>1)
DimPlot(pbmc2)
Seuratではassayスロットに①counts, ②data, ③scale.dataの3種類の発現データがある。
slotの指定はslot=で行えた。デフォルトではdataスロットを参照しているようだ。
pbmc2 <- subset(pbmc, subset = CD3E>1 & CD4>1, slot = "counts")
DimPlot(pbmc2)

assayが複数ある場合はDefaultAssayを変更したうえで行うのが無難だろう。
細胞バーコードで抜き出す
特定の条件で細胞バーコードのベクトルを用意したのなら、cells=引数で細胞抽出することも可能。
cells <- WhichCells(object = pbmc, idents = "Naive CD4 T")

pbmc2 <- subset(pbmc, cells = cells)
DimPlot(pbmc2)

UMAP値で抽出
次元圧縮した時に、外れ値のような小さい細胞集団が生まれることがある。
クラスタリングで他の集団と異なるクラスター番号で区別できればクラスター番号で除くこともできるが、都合よくいかないこともある。
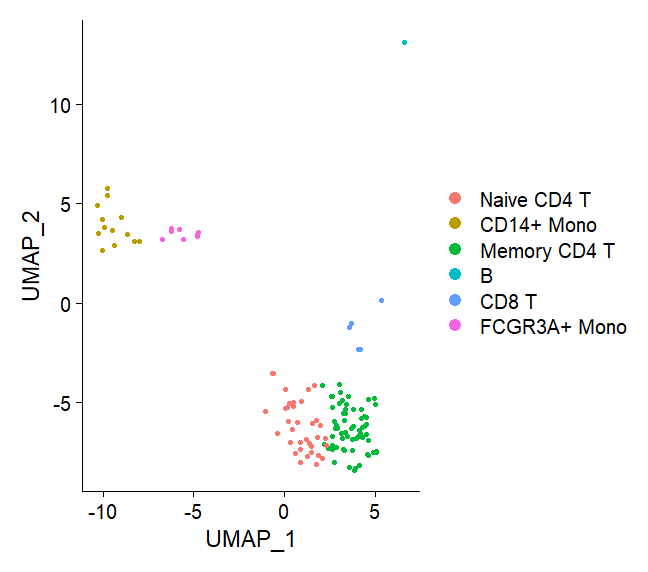
UMAP上で他の集団と十分離れているのなら、UMAPの1次元目、2次元目の値を使って細胞抽出することもできる。
例えば、このUMAPでは右上にB細胞の集団が離れて位置していて、UMAPの2次元目の値のみで分離できそうである。
DimPlot(pbmc)

pbmc2 <- subset(pbmc, subset = UMAP_2 < 10)
DimPlot(pbmc2)

右下の箇所を抽出
pbmc2 <- subset(pbmc, subset = UMAP_1 > -1 & UMAP_2 < 3)
DimPlot(pbmc2)

マウス操作で細胞抽出
DimPlot()の出力をCellSelector()に渡すと、shinyパッケージによりマウス操作で細胞を囲うviewerが立ち上がる。
plot <- DimPlot(pbmc)
select.cells <- CellSelector(plot = plot)

画面上でドラッグして欲しい細胞集団を囲う。Doneを押して終了。

CellSelectorの戻り値は細胞バーコードのベクトルである。

cells=引数に渡して使える。
pbmc2 <- subset(pbmc, cells = select.cells)
DimPlot(pbmc2)



Discussion