ショートリードビニングツール : COMEBin
はじめに
ショットガンメタゲノミクスは、環境水や糞便から回収したゲノムを直接分析することで、微生物の多様性や遺伝子機能に関する情報を得ることのできる分析技術です。
ショットガンメタゲノムのデータ処理の流れはざっくりいうと以下の通りです。
- シーケンサーで取得したfastq.gzのQC & filtering (e.g. fastp)
- ホストゲノム除去 (e.g. kneaddata, hostile)
- Filtering後のFastqをアセンブルしてcontig配列取得 (e.g. metaSPAdes, MEGAHIT)
- contig配列で同じゲノム由来と予測されたグルーピングするビニング (e.g. MetaBat2, MaxBin2)
- bin配列のQC (e.g. CheckM2)
- 系統分類や機能予測 etc...
ショットガンメタゲノムはハイスループットシーケンサーで網羅的に微生物を含むサンプルのゲノム情報を取得するので、ボリュームが大きく複雑なデータになりがちです。
そして、複雑で大ボリュームなデータ処理には計算コストの問題がつきまといます。
その中でもBinningは参照データ無しで実行することが多いので、精度と速度を両立させることが難しい解析ステップです。
今回は複数の異種データを用いた表現学習により、既存のBinnerより結果が向上した、COMEBinを使ってみた記事になります。
ちなみに、Long readについては以下の記事で、SemiBin2とMaxBin2を使ったBinning実施していますので気になったらみてみて下さい。
COMEBin
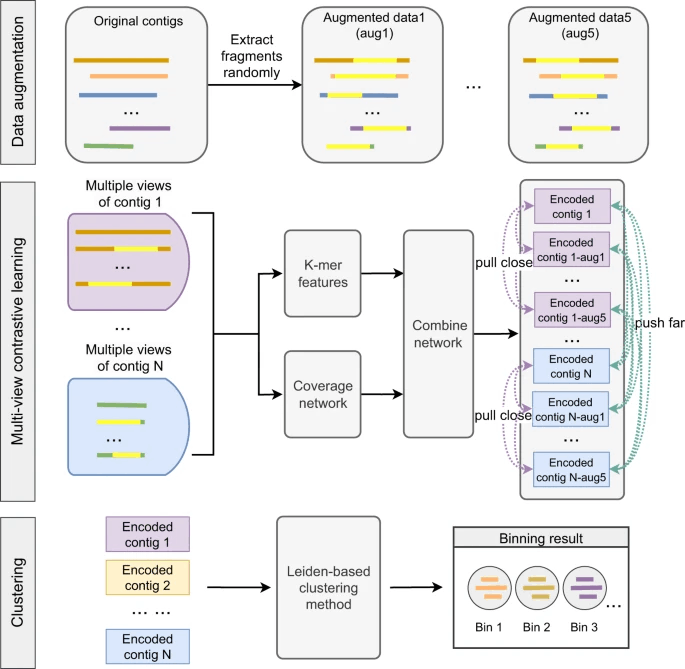
COMEBinは配列のカバレッジとk-merの分布という異なる複数の特徴の対比学習により高品質なbinを生成します。加えて、2019年に開発されたクラスター検出手法であるLeidenクラスタリングを採用し、シングルコピーのマーカーとcontig配列長の情報を与えることで正確なビニング結果を提供します。
比較対象になっていたBinner
| Name | Pub.year | Link | Source |
|---|---|---|---|
| CONCOCT | 2014 | Nat. Methods | https://github.com/RasmussenLab/vamb |
| MetaBat1 | 2015 | PeerJ | https://bitbucket.org/berkeleylab/metabat/src/master/ |
| MetaBat2 | 2019 | PeerJ | https://bitbucket.org/berkeleylab/metabat/src/master/ |
| MaxBin1 | 2017 | BMC Bioinform. | https://sourceforge.net/projects/maxbin/ |
| MaxBin2 | 2015 | Bioinformatics | https://sourceforge.net/projects/maxbin2/ |
| MetaDecoder | 2022 | Microiome | https://github.com/liu-congcong/MetaDecoder |
| VAMB | 2021 | Nat.Biol. | https://doi.org/10.1186/s40168-022-01237-8 |
| CLMB | 2022 | Lect. Notes Comput. Sci. | https://github.com/zpf0117b/CLMB/ |
| SemiBin1 | 2022 | Nat.Comm. | https://github.com/BigDataBiology/SemiBin?tab=readme-ov-file |
| SemiBin2 | 2023 | Bioinformatics | https://github.com/BigDataBiology/SemiBin/ |
COONCOT~MaxBin2まではk-mer頻度ベース(配列の組成情報)かcontigカバレッジ(存在量)のどちらか、またはハイブリッドした方式でBinningを実行します。加えて、MetaDecorderは、k-mer頻度とcontigカバレッジの情報を用いた半教師あり学習によるBinningが実行されます。
VAMB, CLMB, SemiBin1, SemiBin2は深層学習ベースの手法を採用しています。
全体的にアルゴリズムの部分は私が理解しきるのが難しかったので、詳細が知りたい場合は、表に記載した原著論文を参照ください。
CONEBin以外のビニングツールの実行と、論文中の図の生成コマンドは参考になると思いますので置いておきます。
- 論文の結果を得るための各Binnerの実行コマンド
- 視覚化
Insatall COMEBin via bioconda
githubのreadmeに沿って進めていきます。
bioconda経由でCOMEBinをインストール
作業場所の作成
mkdir COMEBin
- CPUのみでCOMEBinを使用する場合、PyTorchなしでインストール
mamba create -n comebin_env
mamba activate comebin_env
mamba install -c conda-forge -c bioconda comebin -y
- NVIDIAのGPUを利用する場合は、PyTorchも同時にインストール (私はNVIDIAのGPUを搭載したPCを使用しているのでこちらで実行)
mamba create -n comebin_env
mamba activate comebin_env
mamba install -c conda-forge -c bioconda comebin
mamba install pytorch pytorch-cuda=11.8 -c pytorch -c nvidia -c conda-forge -y
※ pytorch-cudaはいらないかもしれない。
USAGE
COMEBin version: 1.0.2
Usage: bash run_comebin.sh [options] -a contig_file -o output_dir -p bam_file_path
Options:
-a STR metagenomic assembly file
-o STR output directory
-p STR path to access to the bam files
-n INT number of views for contrastive multiple-view learning (default=6)
-t INT number of threads (default=5)
-l FLOAT temperature in loss function (default=0.07 for assemblies with an N50 > 10000, default=0.15 for others)
-e INT embedding size for comebin network (default=2048)
-c INT embedding size for coverage network (default=2048)
-b INT batch size for training process (default=1024)
デモデータを使ったCOMEBinの実行
著者のgoogle driveからBamファイルとContigファイルをダウンロード
zipファイルを解凍する
unzip comebin_test_data.zip
内包されている以下のファイルを使用します。
- comebin_test_data/BATS_SAMN07137077_METAG.scaffolds.min500.fasta.f1k.fasta
- comebin_test_data/bamfiles/SRR5720343.bam
BATS_SAMN07137077_METAG.scaffolds.min500.fasta.f1k.fastaはアセンブルした後のMulti fastaファイルで、-aに指定します。
comebin_test_data/bamfiles/SRR5720343.bamは、contigにアセンブル前のreadをマッピングしたファイルで、-pに配置しているフォルダを指定します。
では、テストデータセットを使ってCOOMEBinを実行します。 (GPUがあればそれを使用するような実行方法になっています)
CUDA_VISIBLE_DEVICES=0 \
bash run_comebin.sh \
-a comebin_test_data/BATS_SAMN07137077_METAG.scaffolds.min500.fasta.f1k.fasta \
-p comebin_test_data/bamfiles \
-o comebin_test_data/run_comebin_test \
-n 6 \
-t 24 \
2>&1 | tee comebin.log
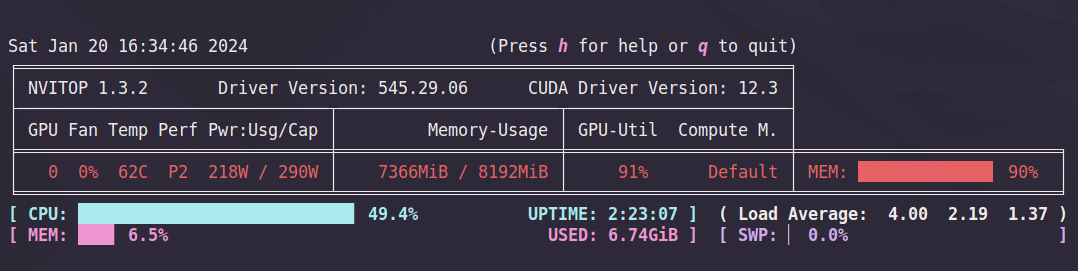
実行中にnvitopでGPUが使われているのか確認したところ、trainingの部分で使われてそうでした。多分、インストールしたpytorchが使われているのかと思います。
最後に、Writing bins: /run_comebin_test/comebin_res/comebin_res_binsと出力されればビニングは終了です。
cpuの環境も作って解析して、実行時間を比較してみました。
| 項目 | Data augmentation | Training | Clustering? | CheckM | total |
|---|---|---|---|---|---|
| CPU | 5分 | 285分 | 15分 | 2分 | 307分 |
| GPU | 5分 | 20分 | 15分 | 4分 | 43分 |
トレーニングのステップでかなりの違いが出ていますね。
※ Logからおおよそで実行時間を目視で抽出したので1分以内のブレがあります。
標準出力に出てきたファイルの内容を確認してみます。
- path : run_comebin_test/comebin_res/cluster_res/unitem_profile/binning_methods/weight_seed_kmeans_k_25_result.tsv/checkm_ar
-
- path : run_comebin_test/comebin_res/cluster_res/unitem_profile/binning_methods/weight_seed_kmeans_k_25_result.tsv/checkm_bac
- file : genome_quality.tsv
BacteriaとArchaeaで分割されたCheckMの結果ファイルでした。tsv形式でbinのContami率や配列長などがまとめられてます。
- path : run_comebin_test/comebin_res/cluster_res
- file : Leiden_bandwidth_0.1_res_maxedges100respara_1_partgraph_ratio_100.tsv.filtersmallbins_200000.tsv
Leidenクラスタリングでまとめられたcontigとそのグループの対応を示したファイルでした。
- path : /home/naoki/Desktop/zenn/run_comebin_test/comebin_res/comebin_res_bins
binningしてまとめられた配列がmulti fasta形式のファイルがまとめられているフォルダでした。
Binを使った後続解析
COMEBinでBinが取得することが出来ました。
Binの使い道は様々ですが、COMEBin_benchmarkを参考に、系統分類であればGTDB-tk、薬剤耐性遺伝子 (ARGs)であればRGI、VFCsであればProdigal + blastpでさっと解析ができるようです。
(またこれは追々...)
以上になります。
備考
GPUを使ってくれない
UserWarning: User provided device_type of 'cuda', but CUDA is not available. Disabling
GPUが使えない状態の場合Warningが出ます。意図的に使わない場合は気にしなくてもいいのですが、GPU搭載のPCを使っているのであれば実行時間短縮のためにも使わない手はないです。
ただ私の場合、試行錯誤しているうちに、CUDA関連のツールのバージョンとPyTorchのバージョンの相性の合うものが用意できたのかGPUでの実行ができるようになりました。
多分大体の人がぶち当たる問題だと思うので、関連しそうなツールのバージョンや参考にしたサイトを残しておきます。参考になれば幸いです。
| Tool | Version | 備考 |
|---|---|---|
| cudNN | 8.9.6 |
https://developer.nvidia.com/rdp/cudnn-archive mamba install cudnn=8.9.2.26でインストールも可 |
| PyTorch | 1.10.2 | https://pytorch.org/ |
| torchvision | 0.11.2 | これは必要 |
| cuda-version | 11.8 | |
| cudatoolkit | 11.8 | https://developer.nvidia.com/cuda-toolkit |
| cuda | 12.3 | https://developer.nvidia.com/cuda-downloads |
| cuda-driver | 545.29.06 | https://developer.nvidia.com/cuda-downloads |
後、cuda関連のツールの利用規約は確認しておいてください。
- cuda関連で参考にしたサイト
Information
追記
2024.1.23 : cuDNN 8.9.2.26をcondaでインストールして実行できること確認
Discussion