NumPyro:打ち切りデータの扱い方
連載している記事の1つです。以前までの記事を読んでいる前提で書いているので、必要であればNumPyroの記事一覧から各記事を参考にしてください。
はじめに
今回は打ち切りデータを扱います。NumPyroには打ち切りデータをいろんな分布に適用できる便利クラスが用意されているので簡単に実装できます。
ライブラリのインポート
import os
import jax.numpy as jnp
from jax import random
import matplotlib.pyplot as plt
import numpy as np
import pandas as pd
import seaborn as sns
import numpyro
from numpyro.diagnostics import hpdi
import numpyro.distributions as dist
import numpyro.distributions.constraints as constraints
from numpyro.infer import MCMC, NUTS
from numpyro.infer import Predictive
from numpyro.infer.util import initialize_model
import arviz as az
az.style.use("arviz-darkgrid")
assert numpyro.__version__.startswith("0.11.0")
numpyro.enable_x64()
numpyro.set_platform("cpu")
numpyro.set_host_device_count(1)
打ち切りデータの扱い方
データの準備
アヒル本のchap07のデータ(chap07/input/data-protein.txt)を使用します。<25が打ち切られたデータで、値の下限が25になっています。今回は打ち切られたデータを使用して打ち切られなかった場合のデータの分布を知りたいと想定します。
df = pd.read_csv("../tutorial/RStanBook/chap07/input/data-protein.txt")
df.head()
# 適当な前処理
df.loc[df["Y"].str.find("<") == 0] = 25
df = df.astype(float)
Y
0 <25
1 32.3
2 <25
3 28.3
4 30.8
打ち切られた分布の定義
NumPyroではdist.TruncatedDistributionを使用していろいろな分布の打ち切られた分布を定義することができます。dist.TransformedDistributionの時と同様にベースとなる分布をbase_distに指定し、打ち切られる値をlowとhighで指定します。
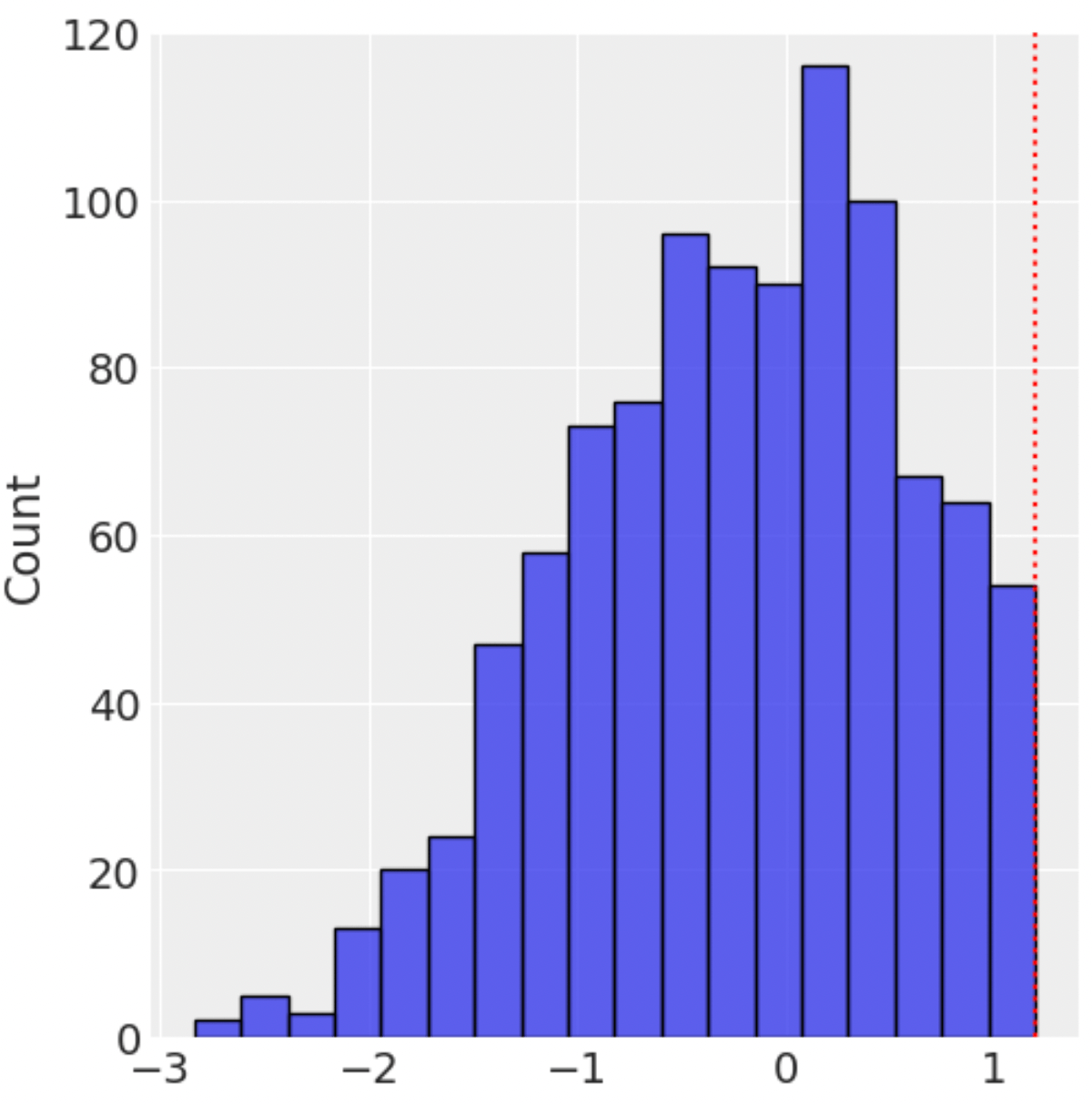
実際の例を以下で見ると、上限値が1.2で打ち切られた分布を定義できていることがわかります。
d = dist.TruncatedDistribution(
base_dist=dist.Normal(0, 1),
low=None,
high=1.2)
samples = d.sample(random.PRNGKey(0), (1000,))
sns.displot(samples)
plt.vlines(1.2, ymin=0, ymax=120, linestyles=":", colors="red")
plt.ylim([0, 120])
また上記と同様の実装になりますが、正規分布のような代表的な分布は既存の打ち切り用のクラスdist.TruncatedNormalが以下のように用意されています。
d = dist.TruncatedNormal(0, 1, low=None, high=1.2)
samples = d.sample(random.PRNGKey(0), (1000,))
sns.displot(samples)
plt.vlines(1.2, ymin=0, ymax=120, linestyles=":", colors="red")
plt.ylim([0, 120])

モデルの定義
上記の例を応用して今回のモデルを定義します。dist.TruncatedDistributionを使用しているところ以外は特別なことはないです。
def model(num_observations, low, y=None):
loc = numpyro.sample("loc", dist.Normal(30, 5))
scale = numpyro.sample("scale", dist.LogNormal())
with numpyro.plate("observations", num_observations):
numpyro.sample("obs", dist.TruncatedNormal(loc, scale, low=low), obs=y)
事前分布からのサンプリング
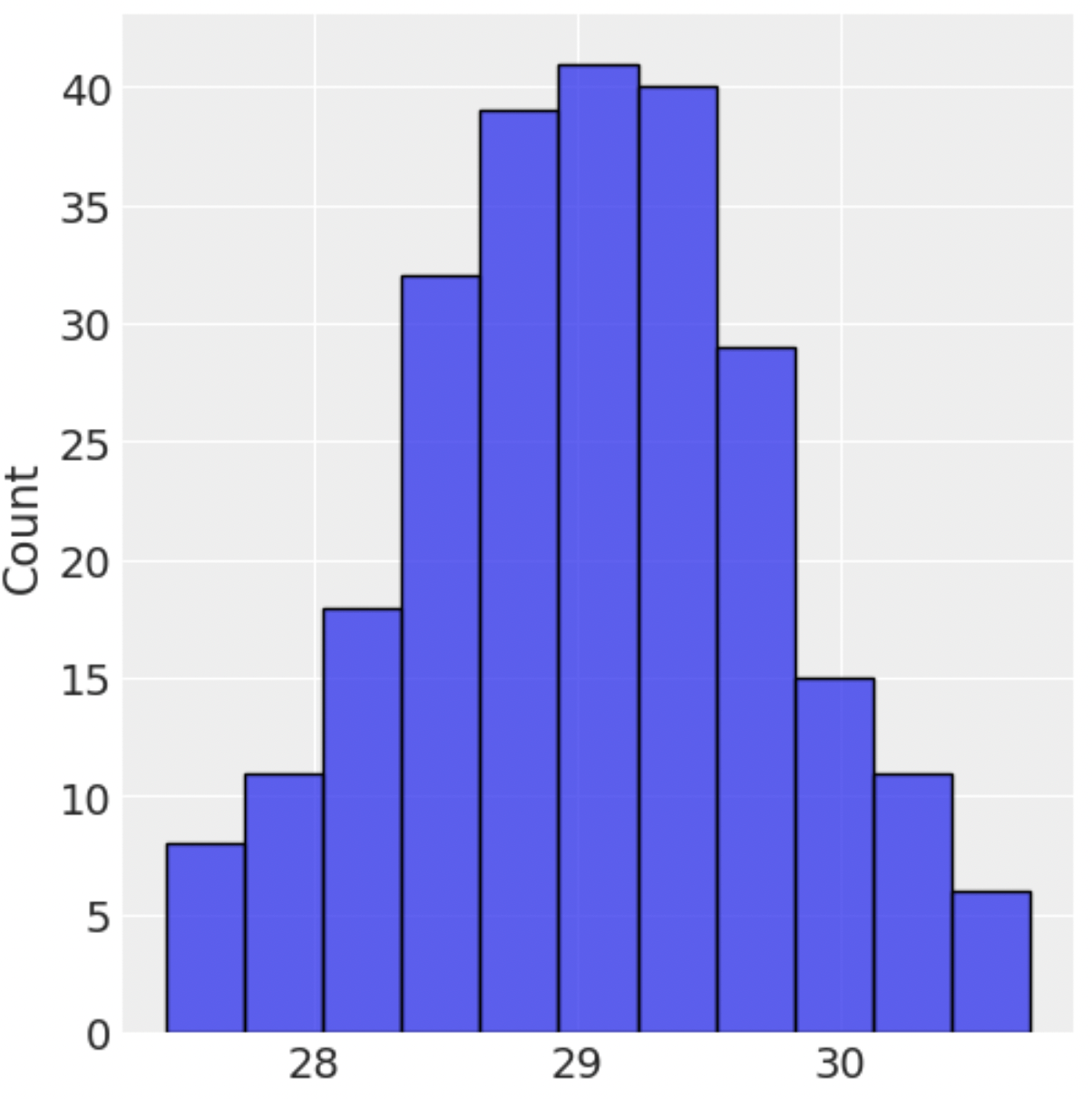
事前分布からのサンプリングを行なって分布の形状を確認してみます。指定した下限値の25で打ち切られた分布になっていることを確認できます。
low = 25
num_observations = 250
num_prior_samples = 100
prior = Predictive(model, num_samples=num_prior_samples)
prior_samples = prior(random.PRNGKey(0), num_observations, low)
sns.displot(prior_samples["obs"][1])
MCMC
# 乱数の固定に必要
rng_key= random.PRNGKey(0)
# NUTSでMCMCを実行する
kernel = NUTS(model)
mcmc = MCMC(kernel, num_warmup=1000, num_samples=2000, num_chains=4, thinning=1)
mcmc.run(
rng_key=rng_key,
y=df["Y"].values,
low=25.,
num_observations=len(df)
)
mcmc.print_summary()
mean std median 5.0% 95.0% n_eff r_hat
loc 26.99 2.89 27.40 22.27 31.29 2756.35 1.00
scale 5.07 1.73 4.77 2.63 7.57 2566.33 1.00
Number of divergences: 0
打ち切りされなかった場合の分布の確認
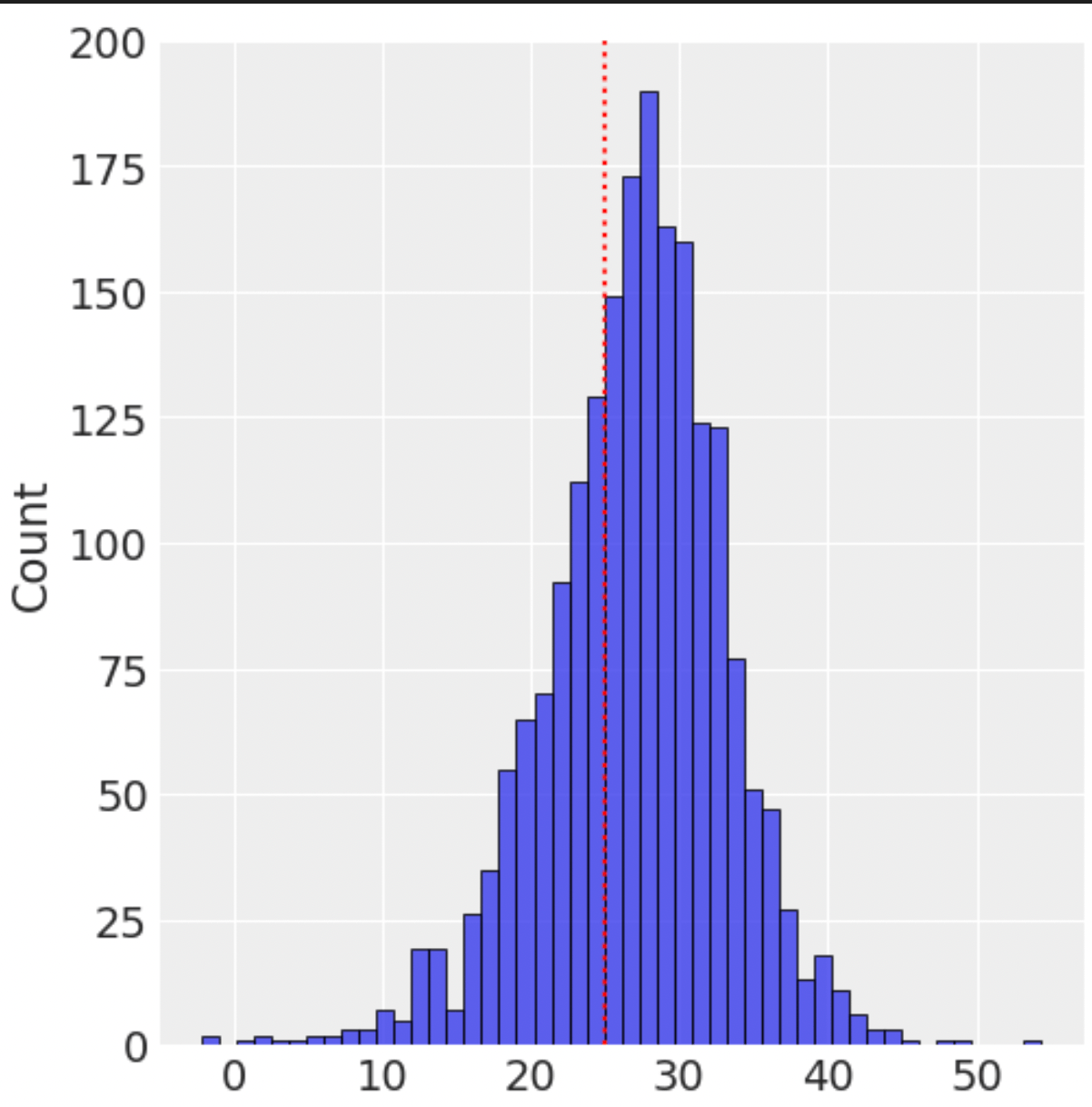
モデルのパラメータが推定できたので、打ち切りがなければどんな分布だったのかを確認してみます。これは事後分布のサンプルを使用して、以下のようにパラメータlowにマイナス無限大を設定するだけで実施できます。それっぽい分布が推定できていることがわかります。
pred = Predictive(model, posterior_samples=mcmc.get_samples())
pred_samples = pred(random.PRNGKey(0), num_observations=num_observations, low=-np.inf)
samples_thinned = pred_samples["obs"].ravel()[::1000]
sns.displot(samples_thinned)
plt.vlines(x=25, ymin=0, ymax=200, linestyles=":", colors="red")
plt.ylim([0, 200])
最後に
以上で「打ち切りデータの扱い方」は終わりです。NumPyroでは簡単に打ち切りデータを扱えますね。次は「欠損値の扱い方」です。
Discussion