Strong bias arising from extremely difficult regression problems 考察
この記事は 仮想通貨botter Advent Calendar 2022 22日目の記事です。
去年に引き続きアドベントカレンダーやっていきます。
まえがき
もともとは、時系列の効率的なメモリであるHiPPO/S4の理論解説と実装を紹介する予定でしたが、3日目の記事で紹介されていたノイズにより回帰がうまく機能しない問題が面白そうだったため、今回はラベルノイズが回帰に与える影響について考えたいと思います。面白い問題を提案してただきありがとうございます!
問題設定
問題設定はrichwomanbtcの記事がわかりやすいです。
端的に書くと、回帰問題で
ここで
コード
import random
import numpy as np
import torch
import torch.nn as nn
import torch.nn.functional as F
import pandas as pd
import skorch
from skorch.callbacks import Callback, Checkpoint, EarlyStopping
from skorch.dataset import CVSplit
from sklearn.model_selection import train_test_split
import holoviews as hv
from holoviews import opts
hv.extension('bokeh')
opts.defaults(opts.Histogram(alpha=0.5, width=600), opts.Overlay(width=600, legend_position='right'))
def torch_fix_seed(seed=42):
# Python random
random.seed(seed)
# Numpy
np.random.seed(seed)
torch.manual_seed(seed)
torch.cuda.manual_seed(seed)
torch.backends.cudnn.deterministic = True
torch.use_deterministic_algorithms = True
torch_fix_seed()
def get_y(d, df):
net = nn.Sequential(
nn.Linear(d, 100, bias=False),
nn.Tanh(),
nn.Linear(100, 1, bias=False)
)
train_data = torch.tensor(np.array(df.astype('f')))
y = pd.Series(net(train_data).detach().numpy().reshape(-1))
y /= dist_factor*y.std() # normalize
return y
net = nn.Sequential(
nn.Linear(d, 100, bias=False),
nn.Tanh(),
nn.Linear(100, 1, bias=False)
)
monitor = lambda Net: all(Net.history[-1, ('train_loss_best', 'valid_loss_best')])
# set param(make trainer)
model = skorch.regressor.NeuralNetRegressor(
net,
max_epochs=500,
lr=0.005,
warm_start=True,
optimizer=torch.optim.Adam,
#optimizer=torch.optim.SGD,
#optimizer__momentum=0.9,
iterator_train__shuffle=True,
callbacks=[Checkpoint(), EarlyStopping()],
train_split=CVSplit(cv=4)
)
def create_model(d):
net = nn.Sequential(
nn.Linear(d, 100, bias=False),
nn.Tanh(),
nn.Linear(100, 1, bias=False)
)
model = skorch.regressor.NeuralNetRegressor(
net,
max_epochs=500,
lr=0.005,
warm_start=True,
optimizer=torch.optim.Adam,
#optimizer=torch.optim.SGD,
#optimizer__momentum=0.9,
iterator_train__shuffle=True,
callbacks=[Checkpoint(), EarlyStopping()],
train_split=CVSplit(cv=4)
)
return model
def get_results(d, N, alpha, dist_factor=1, train_size=0.5):
ret = {}
model = create_model(d)
df = pd.DataFrame(np.random.normal(size=[N, d]).astype('float32'), columns=[f"feat_{i}" for i in range(d)])
y = get_y(d, df)
if alpha == 0:
noisy_y = y.values
else:
noisy_y = y.values + np.random.normal(size=y.shape, scale=alpha).astype('float32')
noisy_y = noisy_y / noisy_y.std()/dist_factor
if train_size == 1.0:
train_df = df
test_df = df
train_noisy_y = noisy_y
test_noisy_y = noisy_y
else:
train_df, test_df = train_test_split(df, train_size=train_size)
train_noisy_y, test_noisy_y = train_test_split(noisy_y, train_size=train_size)
model.fit(train_df.values, train_noisy_y.reshape(-1, 1))
pred = pd.Series(model.predict(test_df.values).flatten())
print(f"---")
print(f"noise level: {alpha}")
print(f"MSE: {np.mean((pred - test_noisy_y)**2):.3f}")
print(f"target mean: {test_noisy_y.mean():.3f}, std: {test_noisy_y.std():.3f}")
print(f"pred mean: {pred.mean():.3f}, std: {pred.std():.3f}, std: {pred.std():.3f}")
print(f"SNR: {((pred - test_noisy_y.mean())**2 ).sum()/ ((test_noisy_y - pred)**2).sum()}")
print(f"---")
ret['noisy_level'] = alpha
ret['mse'] = np.mean((pred - test_noisy_y)**2)
ret['target_mean'] = test_noisy_y.mean()
ret['target_std'] = test_noisy_y.std()
ret['pred_mean'] = pred.mean()
ret['pred_std'] = pred.std()
ret['snr'] = ((pred - test_noisy_y.mean())**2 ).sum()/ ((test_noisy_y - pred)**2).sum()
ret['test_x'] = test_df
ret['test_y'] = test_noisy_y
plot_dist(test_noisy_y, pred)
return ret
_ = get_results(d=100, N=5000, alpha=1, dist_factor=1, train_size=1)

なのに

なぜこんなことが起きるのか?
以下の2つの問題がある
- train(+validation)とtest dataがわけてないこと
- ノイズを乗せたあとに分散を1に正規化していること
TrainとTestを分けた場合と分けなかった場合
元記事では学習で、trainとvalidationを分けていましたが、予測はholdout sampleになっていなかったです。これはoverfittingを引き起こします (ニューラルネットはlabelノイズが強い場合は特にoverfittingすることが知られています)。

trainとtestを分けた場合そもそももとの分布をちゃんとfitできない
元記事の設定に合わせた実験でノイズなしの場合
trainとtestをわけない
get_results(d=10, N=10000, alpha=0, dist_factor=1.0, train_size=1)
MSE: 0.001
target mean: -0.005, std: 1.000
pred mean: -0.005, std: 0.999, std: 0.999
SNR: 846.236328125

trainとtestを50%づつに分ける
get_results(d=10, N=10000, alpha=0, dist_factor=1.0, train_size=0.5)
MSE: 1.024
target mean: 0.007, std: 1.005
pred mean: -0.000, std: 0.125, std: 0.125
SNR: 0.015294821001589298

これは過学習しているため起きている。ノイズが乗っている場合は一見正しく予測できてそうに見えているがそうではない。
データ数やNNのパラメータを頑張ってチューンすると解決するかも? future work
今回は回帰問題だが、分類問題でもノイズにより過学習することが研究で調べられている。
ノイズが多い場合からくる過学習対策の手法は回帰と分類それぞれの場合で多くの論文が提案されているためそういうのを試すといいかも
trainとtestを分けて場合のいろんなパラメータでの実験結果
train dataに対してplotした場合
train N: 5000
d: 100
alpha: 0

MSE: 0.082
target mean: -0.030, std: 2.000
pred mean: -0.029, std: 1.950, std: 1.950
SNR: 46.62129211425781
train N: 5000
d: 100
alpha: 10
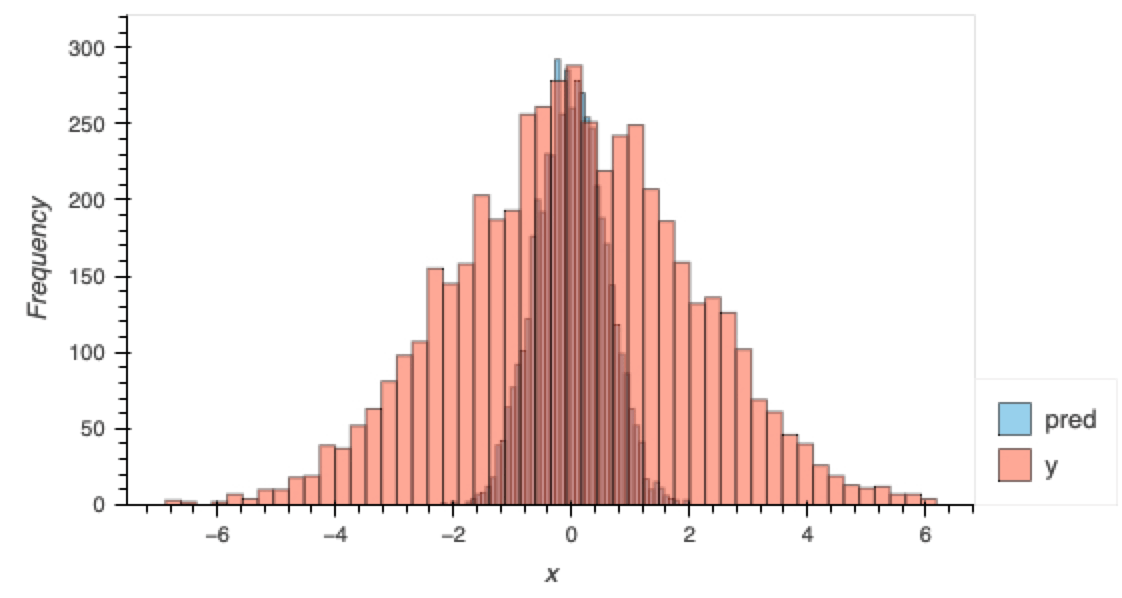
MSE: 3.644
target mean: 0.020, std: 2.000
pred mean: -0.007, std: 0.583, std: 0.583
SNR: 0.09348120540380478
trainとtestに50%ずつでわけて、testに対してpredした場合
train N: 2500
test N: 2500
d: 100
alpha=0

MSE: 4.297
target mean: 0.027, std: 2.006
pred mean: 0.001, std: 0.538, std: 0.538
SNR: 0.06747309863567352
alpha=10

alpha: 10
MSE: 111.611
target mean: 0.054, std: 10.270
pred mean: 0.012, std: 2.741, std: 2.741
SNR: 0.06729775667190552
trainとtestを50%に分けたことでデータ数が半分になっているため、データ数を倍にして学習データが1とおなじになるようにした
train N: 5000
test N: 5000
d: 100
alpha: 0

MSE: 4.107
target mean: -0.059, std: 1.992
pred mean: -0.006, std: 0.360, std: 0.360
SNR: 0.03229061886668205
alpha: 10

MSE: 109.304
target mean: 0.008, std: 10.302
pred mean: 0.055, std: 1.856, std: 1.856
SNR: 0.03153183311223984
ノイズを乗せたあと分散1への正規化の影響
ノイズを乗せて分散が大きくなった分布を正規化すると、もとのノイズが乗る前のデータが0に近づく

コード
N=10000
d=100
alpha = 10
df = pd.DataFrame(np.random.normal(size=[N, d]).astype('float32'), columns=[f"feat_{i}" for i in range(d)])
y = get_y(d, df)
noisy_y = y.values + np.random.normal(size=y.shape, scale=alpha).astype('float32')
y = noisy_y
pred = y/noisy_y.std()
frequencies, edges = np.histogram(pred, 50)
pred_plot = hv.Histogram((edges, frequencies), label='true_y')
frequencies, edges = np.histogram(y, 50)
y_plot = hv.Histogram((edges, frequencies), label='y')
hv.ipython.display(y_plot*pred_plot)
プロットすると

yと比較してノイズが乗っていないyが正規化の影響で0付近に寄っている。
よってそもそも0付近を予測することが正しい問題設定になっている。
ここで得た教訓は、ノイズが多いtargetを予測するときにデータごとにtargetの正規化を行ってしまうとノイズが乗る前のtargetは0付近に集中してしまうということである。(botでの利用の仕方次第では問題はないかもですが)
ノイズが多い場合の精度(MSE)の改善
botterは収益を出してなんぼなので、精度改善を行います。
以下trainとtestを分けて、ノイズにたいして正規化した場合
ノイズが多いデータを点推定しているのが問題だと思うので、確率密度推定を使います。
確率密度推定の手法であるVAEを教師あり学習に適当に改造してみる
普通ののVAEはこちらの記事が参考になります
真の分布を
最後の出てきたのが変分下限で、これを最大化することで、下から2行目のzの事後分布が真の分布と一致します。
最後の行の第一項目は分布を予測する項で、第2項は過学習を抑制する正則化項です。
手前味噌ですが、確率密度推定においては、以前我々が提案した理論的に最適な事前分布をデータから推測する論文があるため今回はこれを使います。
decoderの分布をガウス分布としたとき、testデータ各点に対してp(y|x)は平均と分散を予測します。
test dataに対して予測した平均と分散から作ったガウス分布からサンプリングした場合
確率密度推定
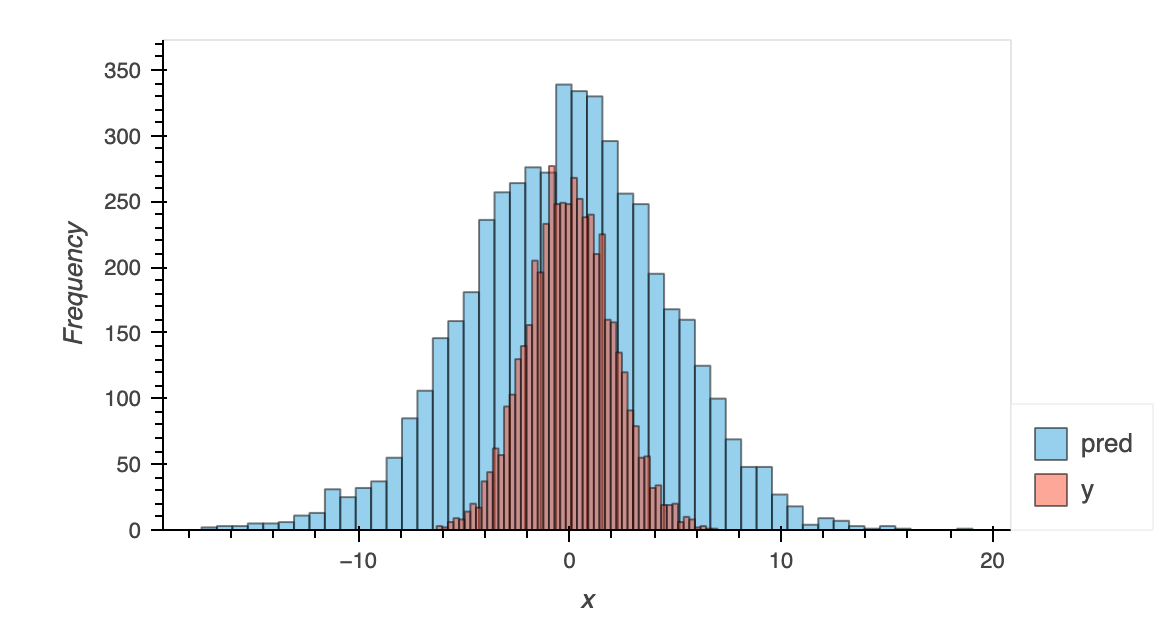
MSE 25.617846
平均値を使った場合

MSE 4.0318956
これは正解データが分散を正規化している問題から、ノイズが乗っていない
ここでデータ
つまり自信がある点だけを使うという方法です。

しきい値以下の分散に対応するデータ点を使ったときのMSE
| しきい値 | MSE |
|---|---|
| 3.5 | 3.52 |
| 4.0 | 3.95 |
| 4.5 | 3.85 |
| 5.0 | 4.02 |
| なし | 4.03 |
データ点ごとの2乗のずれをみているため、結構改善されているのがわかります。
これはbotter的には、取引回数を下げて、自信があるものだけにベットして精度を上げることに対応します。
ポイントは、各点
[追加] regression vs classification
alphaを変えて0より大きいか0以下の2値のaccの変化をプロット
正規化は行っておらず、10 seedでエラーバーをつけた。
結果どっちを使ってもnoiseが大きくなると予測精度が下がり、有意差はなかった。

# Set the noise strengths
alphas_fine_low = np.linspace(0, 10, 10)
# Define dictionaries to store the accuracies for regression and classification
regression_accuracies_no_norm_low = {alpha: [] for alpha in alphas_fine_low}
classification_accuracies_no_norm_low = {alpha: [] for alpha in alphas_fine_low}
for iteration in range(n_iterations):
for alpha in alphas_fine_low:
# Generate the input from a standard normal distribution
x = np.random.randn(n_samples, d)
# Compute y' without normalization
y_prime = f(x) + alpha * np.random.randn(n_samples, d)
# Convert y' to binary labels
y_labels = (y_prime >= 0).astype(int)
# Split the data into a training set and a test set
x_train, x_test, y_train, y_test = train_test_split(x, y_labels, test_size=0.2, random_state=42+iteration)
# Train a logistic regression model
model = LogisticRegression()
model.fit(x_train, y_train.ravel())
# Use the model to predict the output of the test data
y_pred = model.predict(x_test)
# Compute the accuracy of the predictions
classification_accuracies_no_norm_low[alpha].append(accuracy_score(y_test, y_pred))
# Now perform regression
# Split the data into a training set and a test set
x_train, x_test, y_train, y_test = train_test_split(x, y_prime, test_size=0.2, random_state=42+iteration)
# Train a linear regression model
model = LinearRegression()
model.fit(x_train, y_train)
# Use the model to predict the output of the test data
y_pred = model.predict(x_test)
# Convert the predictions and the true outputs to binary labels
y_pred_labels = (y_pred >= 0).astype(int)
y_test_labels = (y_test >= 0).astype(int)
# Compute the accuracy of the predictions
regression_accuracies_no_norm_low[alpha].append(accuracy_score(y_test_labels, y_pred_labels))
# Calculate means and standard deviations
regression_means_no_norm_low = {alpha: np.mean(accs) for alpha, accs in regression_accuracies_no_norm_low.items()}
classification_means_no_norm_low = {alpha: np.mean(accs) for alpha, accs in classification_accuracies_no_norm_low.items()}
regression_stds_no_norm_low = {alpha: np.std(accs) for alpha, accs in regression_accuracies_no_norm_low.items()}
classification_stds_no_norm_low = {alpha: np.std(accs) for alpha, accs in classification_accuracies_no_norm_low.items()}
# Plot the accuracies with error bars
plt.figure(figsize=(10, 6))
plt.errorbar(list(regression_means_no_norm_low.keys()), list(regression_means_no_norm_low.values()), yerr=list(regression_stds_no_norm_low.values()), label='Regression', capsize=5, color='red')
plt.errorbar(list(classification_means_no_norm_low.keys()), list(classification_means_no_norm_low.values()), yerr=list(classification_stds_no_norm_low.values()), label='Classification', capsize=5, color='blue')
plt.xlabel('Alpha (Noise Strength)')
plt.ylabel('Accuracy')
plt.legend()
plt.grid(True)
plt.title('Accuracy of Regression vs Classification with Different Noise Strengths (Alpha from 0 to 10)')
plt.show()

Discussion