「Spike-and-Slab事前分布を理解する」補足(Stan による実装ほか)
はじめに
前回公開した下記の記事では、ベイズ推定において予測への寄与に応じて確率的に変数選択を行うために用いられる Spike-and-Slab 事前分布について解説を行い、そのうえで PyMC を用いた類似実装を紹介した。
本記事では、上記記事に補足する形で下記の3点について記述する。
補足1では、前回の記事で PyMC により実装した(緩やかな)Spike-and-Slab 事前分布を用いた線形回帰について、Stan を用いた書き方を紹介する。
補足2では、CausalImpact で用いられているものと同様な(完全な)Spike-and-Slab 事前分布を用いた線形回帰を、確率的プログラミング言語 (PPL) で実装を試みた結果について紹介する。
補足3では、(完全な)Spike-and-Slab 事前分布による線形回帰を PPL を使わずに Gibbs Sampling を用いて実行した結果を紹介する。
補足に入る前に: この記事でのモデルの呼び分け
Spike-and-Slab 事前分布の最もわかりやすい定式化は、選択変数
一方で前回の PyMC による実装では、実装を容易にするために選択変数が
本記事では便宜的に前者を「完全な Spike-and-Slab 事前分布」、後者を「緩やかな Spike-and-Slab 事前分布」と呼び分けることにする。[1]
補足1: 緩やかな Spike-and-Slab 事前分布を用いた線形回帰の Stan 実装
ここでは、前回の記事で PyMC 実装を行ったのと同様な(緩やかな)Spike-and-Slab 事前分布を用いた線形回帰を Stan を用いて実装する。
Stan も PyMC 同様によく使われる確率的プログラミング言語(PPL)だが、PyMC と異なり離散分布をパラメータとして用いることができない。
したがって、変数選択のために導入した Bernoulli 分布に従う変数
そこで、松浦「StanとRでベイズ統計モデリング」の Chapter 11 で混合正規分布やゼロ過剰ポアソン分布を例に解説されている内容を参考に、周辺化による離散変数の消去を行いモデルの記述を行なった。[2]
モデル概要
前回の記事の繰り返しになるため詳細は省くが、モデルを式で表すと以下のようになる(実行に用いた R のコードはこちら):
記号の定義は前回の記事と同じである。
これのようなモデル設定により、
モデル実装
上記のモデル設定を Stan で書き表すと以下のようになる(なお、説明変数の数 D で表している):
data {
int N;
int D;
matrix[N, D] X;
vector[N] Y;
// for out-of-sample prediction
int N_new;
matrix[N_new, D] X_new;
}
// The parameters accepted by the model.
parameters {
vector[D] beta;
real<lower=0> sigma2;
}
transformed parameters {
vector[N] mu;
mu = X*beta;
real tau = 1/sigma2;
}
// The model to be estimated.
model {
// parameters for beta prior
vector[D] beta0;
array[D] int rho;
vector[D] rho_v;
array[D] real rho_pi;
vector[D] i_D;
vector[to_int(2^D)] lp;
for (i in 1:D) {
beta0[i] = 0;
rho[i] = 0;
rho_v[i] = 0;
rho_pi[i] = 0.5;
i_D[i] = 1;
}
//// variance-covariance matrix
matrix[D, D] des_mat = X' * X;
matrix[D, D] beta_prec_full = 0.5 / N * (des_mat + diag_matrix(diagonal(des_mat)));
// parameters for sigma^2 prior
real nu = 0.01;
real r2 = 0.5;
real sy = variance(Y);
real s = nu * (1-r2) * sy;
// model definition
matrix[D,D] diag_beta_prec = diag_matrix(diagonal(beta_prec_full));
for (k in 1:to_int(2^D)) {
matrix[D,D] beta_prec_mat = quad_form_diag(beta_prec_full, rho_v) + N^2 * diag_pre_multiply(i_D - rho_v, diag_beta_prec);
lp[k] = normal_lpdf(Y | X * beta, sqrt(sigma2)) + multi_normal_lpdf(beta | beta0, sigma2 * inverse(beta_prec_mat)) + inv_gamma_lpdf(sigma2 | nu/2, s/2);
// P(rho) の対数確率を加算する
for (i in 1:D) {
lp[k] += bernoulli_lpmf(rho[i] | rho_pi[i]);
}
// ベクトル rho を更新
int rho_increment = 1;
for (i in 1:D) {
int tmp_added = rho[i] + rho_increment;
if (tmp_added==2) {
rho[i]=0;
} else {
rho[i] = tmp_added;
rho_increment = 0;
}
rho_v[i] = rho[i];
}
}
//print("log_sum_exp(lp): ", log_sum_exp(lp));
target += log_sum_exp(lp);
}
generated quantities {
vector[N_new] Y_new;
vector[N_new] mu_new;
mu_new = X_new * beta;
for (n in 1:N_new) {
Y_new[n] = normal_rng(mu_new[n], sqrt(sigma2));
}
}
着目するべきは、モデル定義を行う下記の箇所である:
for (k in 1:to_int(2^D)) {
matrix[D,D] beta_prec_mat = quad_form_diag(beta_prec_full, rho_v) + N^2 * diag_pre_multiply(i_D - rho_v, diag_beta_prec);
lp[k] = normal_lpdf(Y | X * beta, sqrt(sigma2)) + multi_normal_lpdf(beta | beta0, sigma2 * inverse(beta_prec_mat)) + inv_gamma_lpdf(sigma2 | nu/2, s/2);
// P(rho) の対数確率を加算する
for (i in 1:D) {
lp[k] += bernoulli_lpmf(rho[i] | rho_pi[i]);
}
// ベクトル rho を更新
// (省略)
}
target += log_sum_exp(lp);
ここでは target に加算しているが、その過程で以下のように同時分布
今回の実験の場合でいうと
なお、コード上では配列 lp の各要素に
を代入したうえで、最終的に log_sum_exp(lp) により
を求めている。
可視化結果
前回 PyMC で実行した結果と大局的には大きな違いがないことを確認した。
事後予測値
目的変数
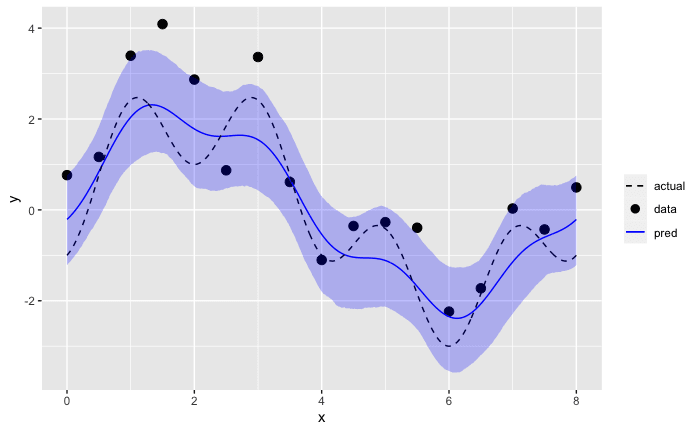
これは、PyMC による予測結果と定性的には大きな違いがない。
回帰係数事後分布
次に、回帰係数
水平の黒線は、細い方は95%、太い方は50% HDI (High Density Interval) である。
赤のマークはサンプルデータ生成時に用いた真の値である。
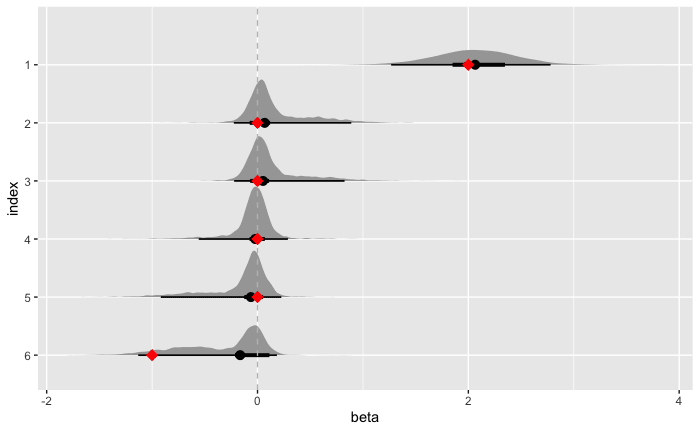
こちらについては PyMC で出力した内容と50%区間の幅など微妙に異なる部分はあるものの、
補足2: 完全な Spike-and-Slab 事前分布の実装を PyMC / Stan で実現できるか?
補足1と前回の PyMC 実装では、冒頭で述べた「緩やかな」方の Spike-and-Slab 事前分布を用いた。
それでは「完全な」方の Spike-and-Slab 事前分布を用いた線形回帰についても、PyMC や Stan を用いて実装できるのだろうか?
これについて筆者は試してみたが、現時点ではうまくいっていない。
困難なポイントとして、以下が挙げられる:
- 確率変数
\boldsymbol{\rho} \boldsymbol{\beta}_\rho - MCMC の収束が遅い
以下、試行内容とその結果について軽く触れる。
PyMC による試行
以下のように、選択変数 rho = pm.Bernoulli('rho', .5, shape=p) の値を用いて回帰係数 beta_rho を定義してみたが、変数 rho には numpy の array のような形で実際の値が入るわけではないためエラーになった。
n = y.shape[0]
p = X.shape[1]
model = pm.Model()
with model:
rho = pm.Bernoulli('rho', .5, shape=p)
p_rho = sum(rho)
X_rho = X[:, rho==1]
Sigma_rho = 0.5 * np.matmul(X_rho.T, X_rho) / n
Sigma_rho += np.diag(np.diag(Sigma_rho))
beta_rho = pm.MvNormal('beta_rho', 0, tau=tau * Sigma_rho, shape=p_rho)
# (以降略)
詳細: https://zenn.dev/link/comments/5a121ffe835838
Stan による試行
MCMC の収束が遅く想定していた結果が得られなかった。[3]
以下のメモのように beta を切り出して beta_rho を作りモデルを記述した。
この場合では Stan コードのコンパイルが成功し実行はできたものの、回帰係数の大きさが
収束の目安となる指標
詳細: https://zenn.dev/link/comments/b68820f6360b9c
補足3: 完全な Spike-and-Slab 事前分布の Gibbs Sampling を用いた実装
そもそもの話ではあるが、今回の設定では完全な Spike-and-Slab 事前分布を用いた線形回帰を確率的プログラミング言語を使わずに実装が可能である。
以下に再掲する前記事の式(1)のように事後分布の関数形が分かるので、これらを利用してサンプリングすればよい:
ただし、
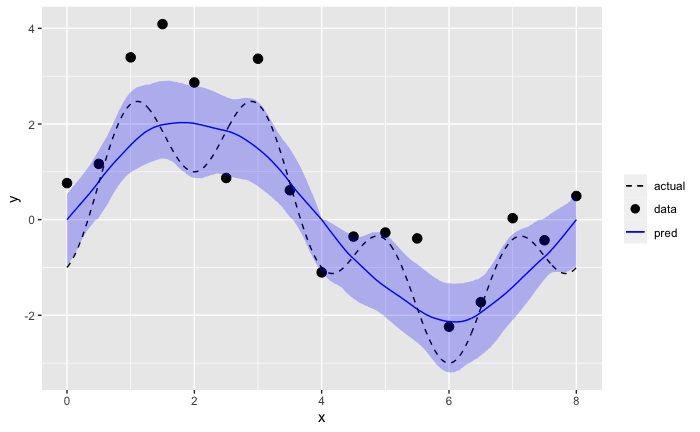
具体的な実行方法についてはこちらのメモもしくは実行コードを参照いただき、以下に結果だけ掲載する。
選択変数
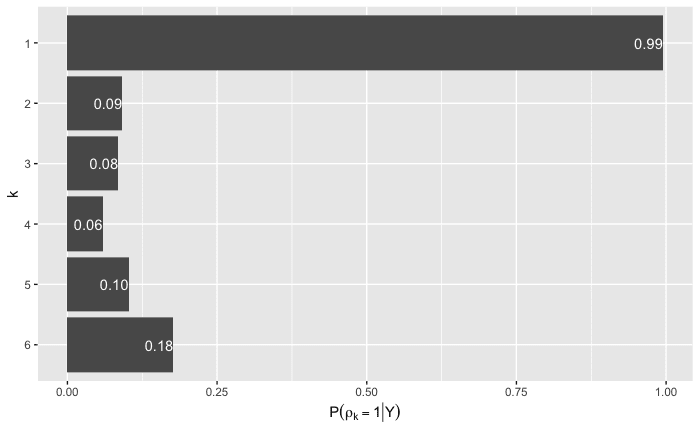
パラメータ
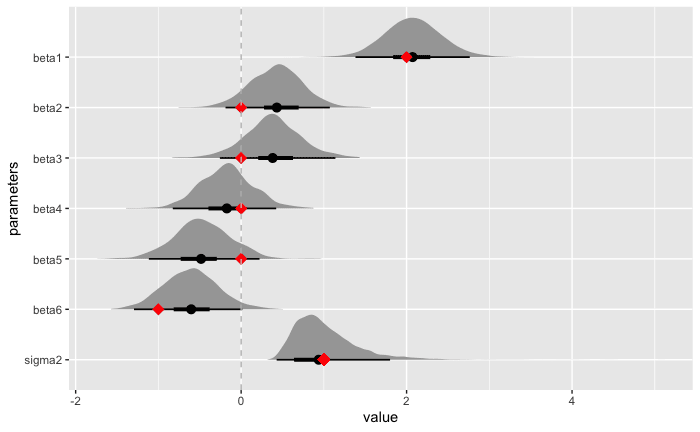
事後予測値
参考文献
-
直感的には、「緩やかな Spike-and-Slab 事前分布」において
\rho_k=0 \beta_k -
本実装は、著者の Matsuura さんからいただいたコメントがきっかけとなりました。改めて感謝申し上げます。 ↩︎
-
なお、「緩やかな」方の Spike-and-Slab 事前分布の場合でも、
\rho_k=0 \beta_k
Discussion