SnowflakeでStreamlitとSnowflake Notebooksを用いたIn silico創薬
はじめに
バイオインフォマティシャン兼データエンジニアのこれえだです。
近年、高度なコンピュータ技術とアルゴリズムの発展により、大規模なデータセットの解析が可能になりました。これにより、化合物の特性や相互作用を迅速に予測することができます。In Silico創薬とは、コンピュータを用いたシミュレーションや解析によって新しい薬の開発を行う手法です。従来の創薬プロセスは非常に高コストで時間がかかります。In Silico創薬では、初期段階での高額な実験を減らし、コンピュータシミュレーションによって有望な候補を絞り込むことができます。そのため、従来の実験ベースの方法と比較して効率的かつ経済的であるため、近年大きな注目を集めています。
SnowflakeでもSnowflakeのハイコンピューティングリソースを利用してIn silico創薬を行うことが可能です。今回は、StreamlitとSnowflake Notebooksを用いたIn silico創薬の一部を解説しようと思います。
StreamlitとSnowflake Notebooksについて
まず今回のIn silico創薬に使うStreamlitとSnowflake Notebooksについて解説します。どちらもSnowflakeが持つネイティブな機能です。それぞれを解説していきます。
Streamlitの特徴
StreamlitはPythonのオープンソースライブラリで、データをインタラクティブなWebアプリケーションとして素早く簡単に可視化することができます。これはHTML、CSS、JavascriptなどのWebアプリケーション開発に必要な知識がなくても構築可能です。SnowflakeではSnowflake上でStreamlitアプリを構築、展開、共有できるStreamlit in Snowflakeという機能も持っています。

Streamlit in Snowflake
Snowflake Notebooksの特徴
Snowflake NotebooksはPython および SQL 用のインタラクティブなセルベースのプログラミング環境を提供するSnowsightの開発インターフェイスになります。Streamlit などの他のライブラリを使用して、データをインタラクティブに視覚化することが可能です。Snowflakeにすでに存在するデータを探索したり、ローカル ファイル、外部クラウド ストレージ などからSnowflake にアップロードすることが可能です。
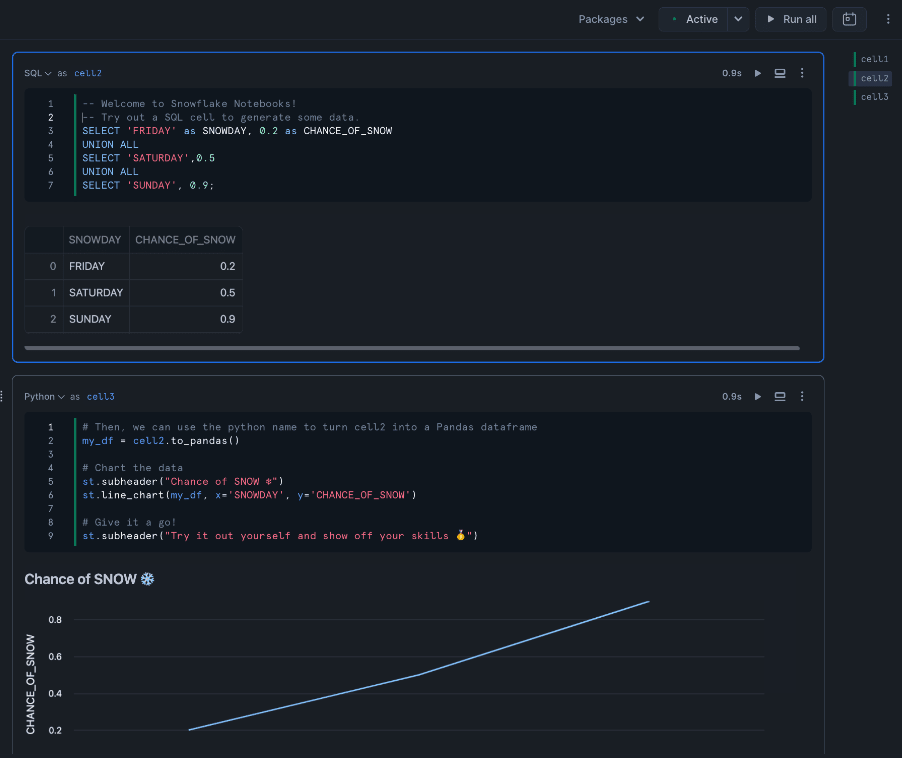
Snowflake Notebooks
StreamlitとSnowflake Notebooksを用いたバーチャルスクリーニング
それでは実際にsnowflakeでIn silico創薬をしていきたいと思います。今回は化合物の類似度を用いたバーチャルスクリーニングを紹介します。特許や論文で薬の候補となる化合物が発表された場合、より有望な類似化合物を探したいことがあります。ここではインフルエンザ治療薬であるノイラミニダーゼ阻害薬「ラニナミビル」に類似した化合物を、ZINC DBのデータからスクリーニングし、タニモト係数で類似度がTop10のものだけをピックアップしてきます。

ZINC DBのデータからバーチャルスクリーニング
ZINC (ZINC Is Not Commercial) Databaseは、無料で利用できる化合物データベースで、主にバーチャルスクリーニングや薬剤発見の研究に使用されます。このデータベースは、特に計算化学やバイオインフォマティクスの分野で広く利用されています。ZINC DBには、何百万もの商業的に入手可能な化合物の3D構造が含まれており、ドラッグライクな性質を持つものや、リード化合物として有望なものが多く含まれています。また、化合物データは、PDB、SDF、MOL2、SMILESなどの多様なフォーマットでダウンロード可能です。
In silico創薬「化合物類似度の評価」
まずは、化合物類似度の評価を評価するサンプルコードを紹介します。下記例では、Cc1ccccc1:トルエン Clc1ccccc1:トリクロロベンゼン の類似度をタニモト係数を使って計測する例です。
import streamlit as st
from rdkit import Chem, DataStructs
from rdkit.Chem import AllChem, Draw
from rdkit.Chem.Draw import IPythonConsole
from PIL import Image
import io
mol1 = Chem.MolFromSmiles("Cc1ccccc1")
mol2 = Chem.MolFromSmiles("Clc1ccccc1")
img1 = Draw.MolToImage(mol1)
img2 = Draw.MolToImage(mol2)
st.image(img1, caption="Molecule 1")
st.image(img2, caption="Molecule 2")
DataStructs.TanimotoSimilarity(fp1, fp2)
メインで使うライブラリとしてRDKitを使っています。RDKitは、オープンソースの化学情報学ライブラリで、分子の操作、シミュレーション、分析、描画などを化学情報学、バイオインフォマティクス、薬剤開発などの分野で使われています。その他モジュールは以下の通りです。
Chem: 分子の作成や操作を行うモジュール。
DataStructs: 分子間の類似度計算を行うためのモジュール。
AllChem: 構造描画や計算に関する高度な機能を提供。
Draw: 分子の描画を行うモジュール。
IPythonConsole: IPythonでの分子描画をサポートするモジュール。
化合物の分子構造をSMILES表記から生成していきます。Chem.MolFromSmiles 関数を使用して、SMILES表記から分子オブジェクトを作成します。次に、分子のフィンガープリントを計算します。ここではMorganフィンガープリント(ECFP)を使用します。生成した分子構造の画像を描画し、Streamlitを使って表示しています。最後にタニモト係数でトルエンとトリクロロベンゼンの類似度の評価を行っています。結果としては「0.5384615384615384」でした。
In silico創薬「バーチャルスクリーニング」
それでは実際にバーチャルスクリーニングに入っていきます。ラニナミビルの分子量が約350,ALogPが-2.92なので、ZINCの分子量350-375,LogPが-1未満の340万化合物の区画を選択し、最初の1セットだけダウンロードして使っていきます。下記コマンドを自身のターミナルで実行してファイルをダウンロードしてください。
wget http://files.docking.org/2D/EA/EAED.smi
ダウンロードが完了したら、notebooks上にアップロードをしてください。
次に解析に使うコードを全文紹介します。
# cell 1
spl = Chem.rdmolfiles.SmilesMolSupplier("EAED.smi")
len(spl)
# cell 2
laninamivir = Chem.MolFromSmiles("CO[C@H]([C@H](O)[C@H](O)CO[C@H]1OC(=C[C@H](NC(=N)N)[C@H]1NC(=O)C)C(=O)O")
laninamivir_fp = AllChem.GetMorganFingerprint(laninamivir, 2)
def calc_laninamivir_similarity(mol):
fp = AllChem.GetMorganFingerprint(mol, 2)
sim = DataStructs.TanimotoSimilarity(laninamivir_fp, fp)
return sim
# cell 3
similar_mols = []
for mol in spl:
sim = calc_laninamivir_similarity(mol)
if sim > 0.2:
similar_mols.append((mol, sim))
# cell 4
similar_mols.sort(key=lambda x: x[1], reverse=True)
mols = [x[0] for x in similar_mols[:10]]
# cell 5
def visualize_mols(mols, grid_size=(5, 2)):
st.header("Visualized Molecules")
img_per_row, img_per_col = grid_size
mol_imgs = [Draw.MolToImage(mol) for mol in mols]
width, height = mol_imgs[0].size
canvas_width = width * img_per_row
canvas_height = height * img_per_col
canvas = Image.new('RGB', (canvas_width, canvas_height))
for i, img in enumerate(mol_imgs):
row = i // img_per_row
col = i % img_per_row
x = col * width
y = row * height
canvas.paste(img, (x, y))
buf = io.BytesIO()
canvas.save(buf, format='PNG')
st.image(buf.getvalue(), use_column_width=True)
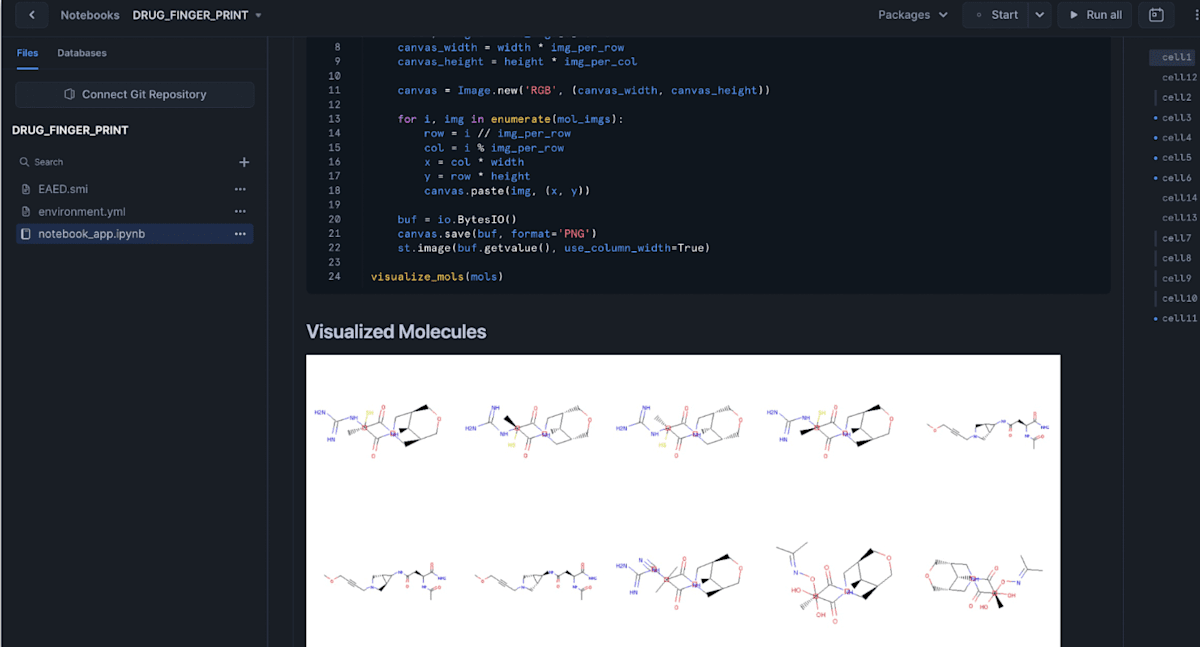
visualize_mols(mols)

実行風景
実行結果
上記のコードを実行すると、Snowflake Notebooks上でノイラミニダーゼ阻害薬「ラニナミビル」に類似した化合物Top10をスクリーニングし、Streamlitで分子構造を可視化することができます。
コードの解説
このPythonスクリプトは、化学分子の類似度を計算し、類似分子を可視化するStreamlitアプリケーションです。以下に各セルの解説を示します。
セル 1: SMILESファイルの読み込み
spl = Chem.rdmolfiles.SmilesMolSupplier("EAED.smi")
len(spl)
このセルでは、EAED.smiというファイルからSMILES形式の化学分子を読み込みます。SmilesMolSupplierを使用してファイル内のすべての分子をロードし、リストsplに格納します。len(spl)は、読み込んだ分子の数を返します。
セル 2: 基準分子のフィンガープリント計算
laninamivir = Chem.MolFromSmiles("CO[C@H]([C@H](O)[C@H](O)CO[C@H]1OC(=C[C@H](NC(=N)N)[C@H]1NC(=O)C)C(=O)O")
laninamivir_fp = AllChem.GetMorganFingerprint(laninamivir, 2)
def calc_laninamivir_similarity(mol):
fp = AllChem.GetMorganFingerprint(mol, 2)
sim = DataStructs.TanimotoSimilarity(laninamivir_fp, fp)
return sim
ここでは、基準となる化学分子「ラニナミビル」をSMILES文字列から生成し、その分子のフィンガープリントを計算します。さらに、他の分子との類似度を計算する関数calc_laninamivir_similarityを定義します。この関数は、引数として渡された分子のフィンガープリントを計算し、ラニナミビルのフィンガープリントとのタニモト類似度を返します。
セル 3: 類似分子のフィルタリング
similar_mols = []
for mol in spl:
sim = calc_laninamivir_similarity(mol)
if sim > 0.2:
similar_mols.append((mol, sim))
このセルでは、splリスト内のすべての分子に対して、ラニナミビルとの類似度を計算します。類似度が0.2を超える分子をsimilar_molsリストに追加します。
セル 4: 類似分子のソートと選択
similar_mols.sort(key=lambda x: x[1], reverse=True)
mols = [x[0] for x in similar_mols[:10]]
ここでは、similar_molsリストを類似度の降順でソートし、最も類似度が高い10個の分子を選択します。選択された分子はmolsリストに格納されます。
セル 5: 分子の可視化
def visualize_mols(mols, grid_size=(5, 2)):
st.header("Visualized Molecules")
img_per_row, img_per_col = grid_size
mol_imgs = [Draw.MolToImage(mol) for mol in mols]
width, height = mol_imgs[0].size
canvas_width = width * img_per_row
canvas_height = height * img_per_col
canvas = Image.new('RGB', (canvas_width, canvas_height))
for i, img in enumerate(mol_imgs):
row = i // img_per_row
col = i % img_per_row
x = col * width
y = row * height
canvas.paste(img, (x, y))
buf = io.BytesIO()
canvas.save(buf, format='PNG')
st.image(buf.getvalue(), use_column_width=True)
visualize_mols(mols)
最後に、選択された分子をグリッド形式で可視化する関数visualize_molsを定義し、実行します。この関数は、与えられた分子リストを画像に変換し、指定されたグリッドサイズ(ここでは5x2)に従ってキャンバスに配置します。キャンバスの画像はバッファに保存され、Streamlitを使用して表示されます。
SnowflakeでIn silico創薬をするメリット
SnowflakeでIn silico創薬のバーチャルスクリーニングを試してみたことで、色々とメリットがあることに気づいたのでまとめてみようと思います。
1. 機械学習フレームワークの利用
Snowpark MLなどのフレームワークを利用することで、複雑なインフラの設定、ローカルでの環境構築が不要になります。Snowflakeは、自動的にスケーリングするため、大規模なデータセットや複雑な機械学習モデルの訓練にも対応可能です。ユーザーは必要に応じてリソースを拡張でき、計算能力を最大限に活用できます。
2. 大量の化合物データセットを効率よく格納
創薬プロセスにおいて必要となる大量の化合物データや生物学的データを一元管理が可能です。これにより、データの可視性が向上し、研究者は必要なデータに迅速にアクセスできます。またSnowflakeの強力なクエリエンジンを使用して、膨大な化合物データセットを高速に検索およびフィルタリングすることができます。
3. ハイコンピューティングリソースが要求される解析まで一貫して解析が可能
Snowpark Container Serviceを利用すれば、分子動力学(MD)シミュレーションやドッキングシミュレーション、タンパク質の3次元構造予測、PyMOLによる分子構造の立体的な可視化など、高度な計算リソースを必要とする解析を実行できます。ハイコンピューティングリソースが要求される解析までエンドツーエンドで解析することが可能です。これにより、研究者は計算負荷の高いシミュレーションをエンドツーエンドの解析ワークフロー実行できます。

PyMOL ref:https://pymol.org/animate.html?
終わりに
いかがだったでしょうか。Streamlit in SnowflakeやSnowflake Notebooksはデータサイエンティストのみならず、ケモインフォマティクスやバイオインフォマティクスに取り組むインフォマティシャンにとっても使える機能です。今回はNotebooks上にアップロードしたSMILES形式データを使いましたが、あらかじめテーブルにアップロードしておけばもっと高速でスクリーニングをかけることが可能になりますし、こちらの記事のRegistry of Open Data on AWSに存在するChEMBLデータを外部ステージとして取り込める状態にしておけば公共データをうまく使うこともできます。より最適なIn Silico創薬環境を整えて行きたいと思います。
Discussion