【論文】Biomni - A General-Purpose Biomedical AI Agent
はじめに
ライフサイエンス界隈で話題になっているBiomni。
スタンフォードが公開したAI Scientistです。
プレプリントがbioRxivに投稿されたのが2025年6月2日。同時にウェブアプリケーションも公開。
7月9日にOSS (0.0.1) がGitHubで公開されました(Apache-2.0)。
- 公式サイト: Biomni - A General-Purpose Biomedical AI Agent
- 論文: Biomni: A General-Purpose Biomedical AI Agent | bioRxiv
- GitHub: snap-stanford/Biomni: Biomni: a general-purpose biomedical AI agent
次に読む論文リスト
背景
2024年8月12日にSakana AIからThe AI Scientistがリリースされてから、いわゆるAI Scientistが盛り上がっていると感じます。もちろんそれ以前もAI科学者という概念はありました。有名どころで言えばSonyの北野さんのNobel Turing Challengeなど。ただ、大規模言語モデル、生成AIの普及に伴い現実味が増してきて、それに伴い分野の盛り上がりを感じています。
2025年2月18日にはGoogleからAI co-scientist、5月19日にはFutureHouseからRobin、そして6月2日にスタンフォード大学からBiomniが報告されています。
Sakana AIのThe AI Scientistは機械学習研究を事例として用いており、ライフサイエンス研究への言及はわずかでした。GoogleのAI co-scientistはライフサイエンス研究での成果も報告されていましたが、利用は一部の研究者に限定されており、かつソースコードが公開されていないため実態は不明でした。
そんな中、FutureHouseのRobin、スタンフォード大学のBiomniはソースコードが公開されており、ライフサイエンス研究におけるAI Scientistの全容を把握するのには最適な教材と言えます。
私自身、今年は実験科学へのAIエージェントの活用に取り組んでおり、大いに参考にさせていただこうということで論文およびソースコードを紐解きます。
利用登録
とりあえず、公式サイトにアクセス。
利用者登録が必要。
聞くところによると数日以内には利用できるようになるらしい。
公式サイト
トップページ
トップで「A General-Purpose Biomedical AI Agent... with 150 specialized tools, 59 databases, and 105 software to automate」と標榜。
AI Scientistのツール数は、多く見えても1つのデータベースごとに複数のツールが用意されていて利用可能なDB数は少なかったりするのですが、Biomniは59のデータベースを接続しています。これはなかなかな数です。
これだけ多くのツールをどうやって使いこなしているか、実装に興味が湧きます。
しかし、Slack、X、Linkedinとコミュニティ形成に余念がないです。最近のAI研究はGitHub Pagesで綺麗に紹介サイトを作っていたり、研究の中心部分以外にも力を入れていると感じます。

利用の様子を動画で確認できます。ざっとどんな風に使えるかが高速に流れます。
印象的なのは、パスウェイやヒートマップなど、ライフサイエンス研究特有の表現形式にも対応していることです。これはChatGPTやManusなどの汎用的AIエージェントでは明確に弱い部分ですね。

About
ちょっと詳しめの解説が載っています。
Key componentsとして2つが紹介されています。
- Biomni-E1: バイオメディカルエージェントが利用可能な統一された環境。さまざまなデータベースやツールへのアクセスが可能な環境。
- Biomni-A1: 広範なバイオメディカル領域にまたがるタスクを(あらかじめ定められたテンプレートなしに)遂行可能な汎用的なエージェント
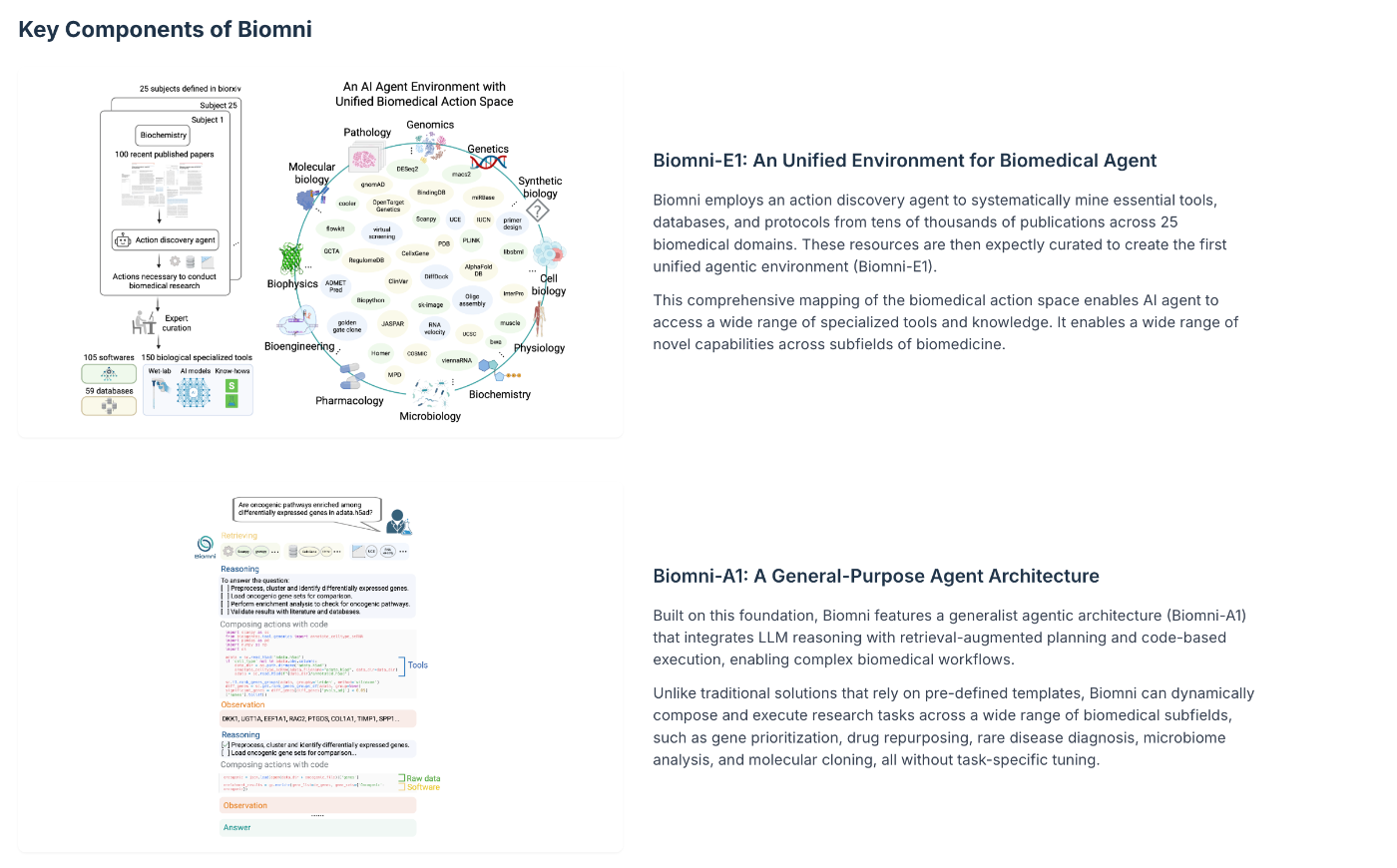
これが肝。
Enviroment
上述のE1に内包されるツール群。データベース、ツール、ソフトウェアという呼び分けをしている。

ツールとソフトウェアの違いは?
Team
Blog
FAQ
データ利用のポリシーや、アクセス、プライシングに関する回答。

計算資源に関する質問は気になる。
What is the computing instance in the platform?
The platform is running on AWS Fargate with 16GB of memory, 4 vCPUs, and 60GB of ephemeral storage.
ローカルで動かす時はこれくらいを目安にすれば良いか、
HPはこんなもん。
論文読んでGitHubのソースコードを見たり、動かしたりしてみよう。
プレプリント
以下からフルテキストが閲覧可能。AI界隈のオープンな文化最高。
ざっと読み
背景として、バイオメディカル(生物医学)研究ではデータセット、分析ツール、論文の増加、実験の複雑化が、新たな発見を阻んでいる。ことを挙げています。
そこで著者らが作ったのがBiomniです。Biomniは
a general-purpose biomedical AI agent designed to autonomously execute a wide spectrum of research tasks across diverse biomedical subfields.
多様な生物医学サブフィールドにわたる幅広い研究タスクを自律的に実行するように設計された汎用生物医学AIエージェント
と紹介されています。
Biomniのエージェントとしての特徴は初の統合的エージェント環境(the first unified agentic environment)であると言えます。これは生物医学のアクション空間を体系的にマッピングしたものであり、これをエージェントに扱わせるのがBiomniの大きな特徴と言えます。
対して、Biomniは多数のツールを集めた"環境"を用意し、汎用エージェントが"事前定義されたテンプレートや硬直的なタスクフローに頼ることなく、複雑な生物医学ワークフローを動的に構成し実行する"と主張しています。
エージェントシステムでよく言われるのは「ツールが増えすぎるとエージェントがツールの使い分けができなくなる」という問題です。そのような状況に陥ったら、マルチエージェントを導入するのが良いというのが通説(というほど確立していないかもしれないが)でした。
果たしてBiomniではどのようにして多数なツールをエージェントに適切に使いこなさせているのか、ソースコードは特にその部分に注目したいです。
またプロンプトチューニングなしに広範なタスクに対応できるというのもかなり気になります。幸いソースコードが公開されているので、詳らかにしましょう。
Biomni-E1 (エージェントの相互作用のための生物医学アクション空間を定義する基盤環境)
Biomniの統合的エージェント環境Biomni-E1に含めるツールを選定するために、生物学文献リポジトリの25の分野にまたがる数万の論文からデータベースやツールを選定するアクション発見エージェントを構築している。
Biomni-E1には、150の専門的な生物医学ツール、105のソフトウェアパッケージ、59のデータベースが含まれている。
Biomni-A1 (幅広い生物医学タスクを柔軟に実行できる汎用エージェントアーキテクチャ)
E1が提供するツールやデータベースを利用して、A1はタスクを遂行する。
A1はざっくり以下の進め方でタスクを遂行する。
- ユーザーのクエリが与えられる
- 検索システムを使用して、最も関連性の高いツール、データベース、ソフトウェアを特定
- LLMベースの推論とドメインの専門知識を適用して、詳細なステップバイステップの計画を生成
- 各ステップは実行可能なコードを通じて表現
ケーススタディ
- 458ファイルのウェアラブルセンサーデータを分析して新しい洞察を生成
- 単一細胞RNA-seqやATAC-seqデータなどの大規模な生データセットに対して包括的なバイオインフォマティクス分析を迅速に実行し、新しい洞察と仮説を生成
- ウェットラボの研究者を支援するために実験室プロトコルを自律的に設計
Figureざっと見
Figure1. Overview of the unified biomedical action space and agent environment in Biomni.
aはバイオメディカルアクション空間のキュレーション。大量の論文から自動でデータベース、ソフトウェア、ツールを抽出したことがわかる。
bはそうして作られたアクション空間(Biomni-E1)。
cはエージェントのタスク遂行の様子。
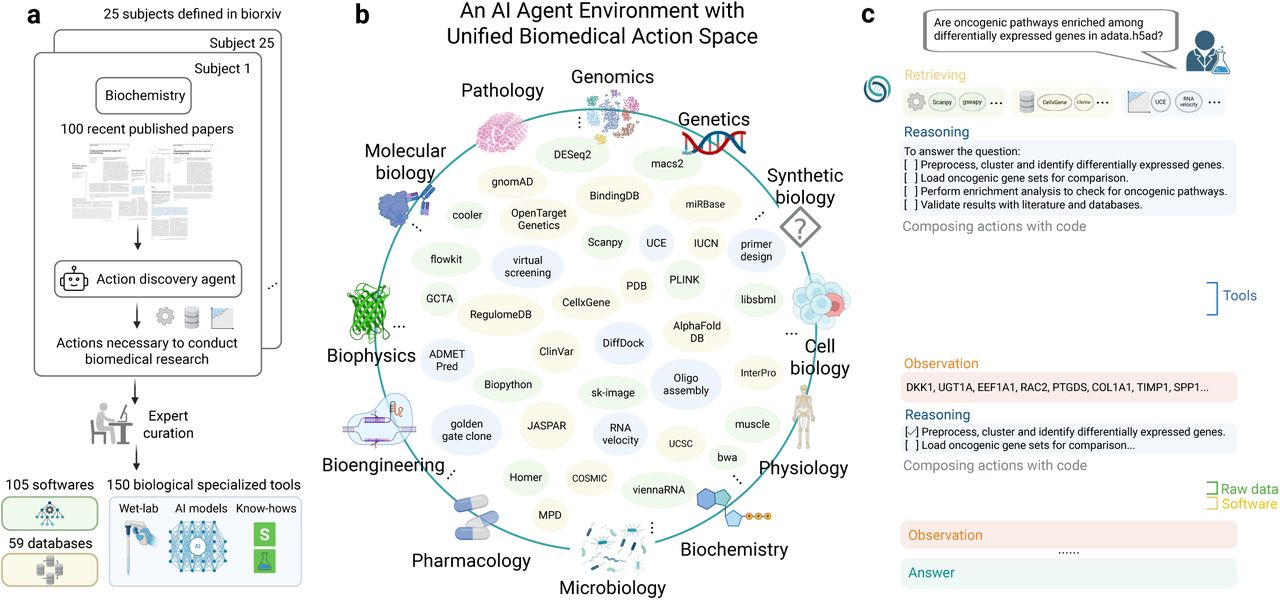
Figure2. Zero-shot generalization of Biomni across diverse realistic biomedical tasks.
aは広範な高度な知的タスクに関するQAのHumanity's Last Exam、ライフサイエンス研究に特化したQAのLAB-Benchの2つで評価。LAB-BenchはDbQA、SeqQAというそれぞれDB操作、シークエンス理解に関するQAのみ利用。
bはunseenなタスクについての評価。タスク固有のファインチューニング、プロンプトエンジニアリングなしに評価した。リアルなバイオメディカルな問題として、変異体の優先順位付けとGWAS因果遺伝子検出(遺伝学とゲノミクス)、摂動スクリーン設計(機能ゲノミクス、免疫学)、患者遺伝子の優先順位付け、希少疾患診断(臨床ゲノミクス)、薬物再利用(薬理学)、マイクロバイオーム疾患-分類群バイオインフォマティクス分析(微生物学)、および単一細胞RNA-seq細胞アノテーション(単一細胞生物学)を対象とした。

Figure3. Biomni autonomously executes complex multi-modal biomedical analyses to generate hypothesis.
Biomniは458枚の生のExcelシートから成る30人の個人からのCGM由来の熱産生応答データと活動データ、約336,000個の核液滴からの単一細胞マルチオミクスデータを解析し、独自の知見を述べた。

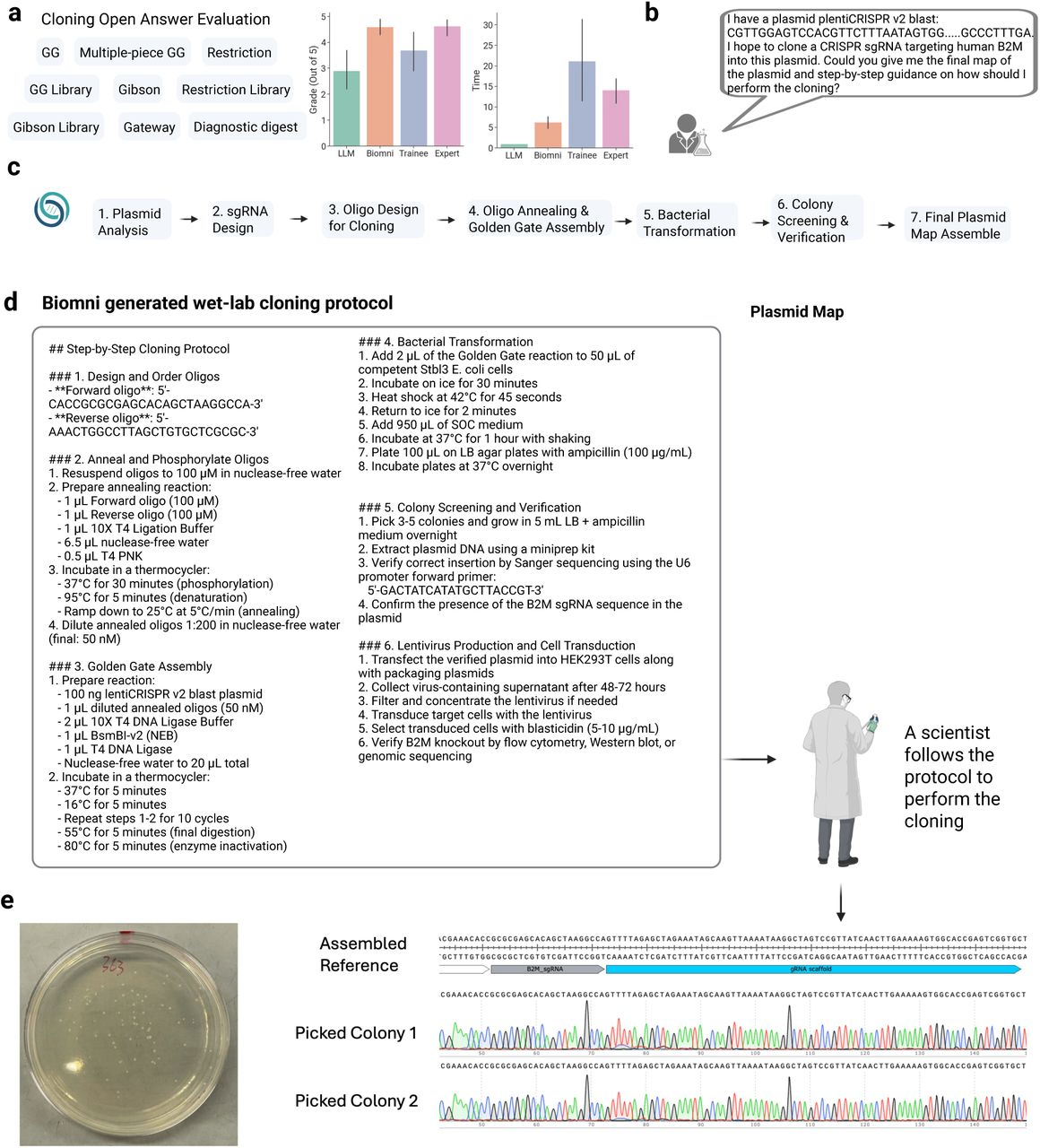
Figure4. Biomni designs wet-lab experimental protocol.
ウェットラボの実験プロトコルをデザインした。専門家レベルと同等の設計能力を示し、かつかかる時間は専門家よりもはるかに少ないと述べている。
Results
概要は頭に入ったので、Resultsを読み進める。
アクション空間のキュレーション自体をAI-drivenなアプローチで実施した。
25のカテゴリーごとに最新の100件の論文を選定し、An action discovery LLM agent (アクション発見LLMエージェント) が各論文から研究を再現するために必要なタスク、ツール、データベース、ソフトウェアを抽出した。
Biomni-E1は150の専門的な生物医学ツール、105のソフトウェア、59のデータベースを含む。
なお、それぞれの違いは以下の通り(by Claude)
- ツール(Tools) - 150個
- 特定の生物医学タスクを実行するための専門的な機能やアルゴリズム
- 具体例:
- BLAST検索を実行する機能
- タンパク質の3D構造を予測するAlphaFold
- 遺伝子オントロジー(GO)解析ツール
- 系統樹作成アルゴリズム
- プライマー設計ツール
- 特徴:
- 特定の目的に特化
- 入力→処理→出力が明確
- Biomniが直接呼び出して使用
- データベース(Databases) - 59個
- 生物医学情報を保存・提供するデータリポジトリ
- 具体例:
- PDB:タンパク質3D構造データ
- GenBank:DNA配列データ
- ClinVar:臨床的変異情報
- KEGG:代謝経路情報
- DrugBank:薬物情報
- 特徴:
- 情報の保存と検索が主目的
- 定期的に更新される
- クエリを送って情報を取得
- ソフトウェア(Software) - 105個
- より広範な機能を持つ汎用的なプログラム環境
- 具体例:
- R:統計解析言語環境
- Bioconductor:Rの生物情報学パッケージ群
- samtools:配列データ処理ソフト
- GATK:ゲノム解析ツールキット
- ImageJ:画像解析ソフト
- 特徴:
- 複数の機能を含む統合環境
- コマンドラインやスクリプトで操作
- 他のツールの基盤となることも多い
データベースは利用方法で2つ分かれる。
1つ目はWeb API経由で利用可能なもの(e.g. PDB, OpenTarget, ClinVar)。データベースごとに統一された関数を実装。各関数は自然言語を受け取り、クエリを動的に生成実行する。
2つ目はWebインターフェースを持たないもの。これらはデータレイクにダウンロードし、pandas DataFrameで前処理。
そして肝のBiomni-A1。個別のタスクに特化したプロンプトエンジニアリングやファインチューニングなしに、さまざまなタスクに対して対応できる。
Results - 評価
比較対象は汎用的なアーキテクチャ。
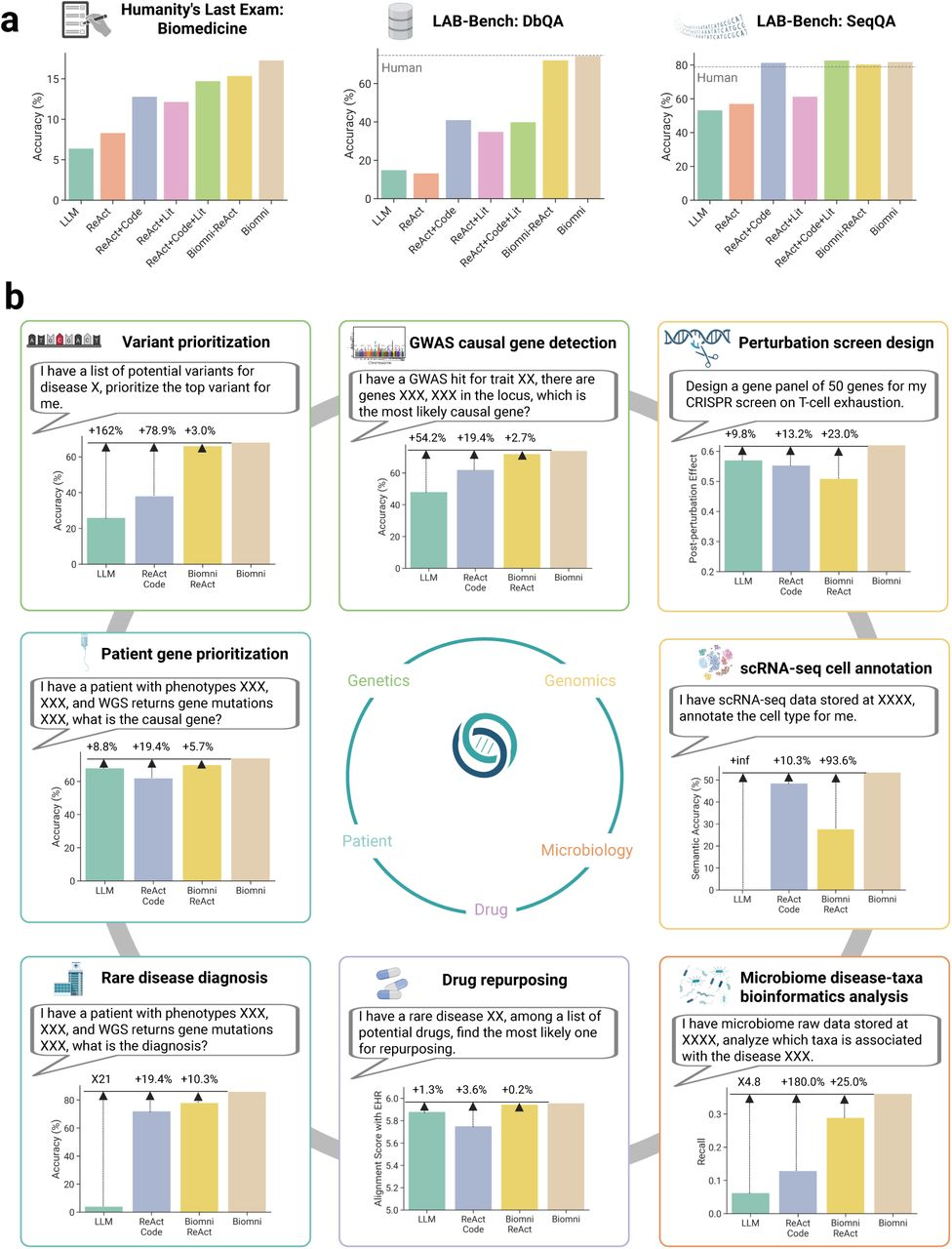
aで利用したベンチマークはHumanity's Last Exam (HLE) とLAB-Bench。
HLEは生物学14分野から52問を抜粋。
比較対象の特徴整理 by Claude
LLM - Base LLM(ベース大規模言語モデル)
ReAct - 推論と行動を組み合わせた手法
ReAct+Code - ReActにコード実行機能を追加
ReAct+Lit - ReActに文献検索機能を追加(Literature Agent)
ReAct+Code+Lit - コード実行と文献検索の両方を持つ
Biomni-ReAct - Biomni環境でReAct手法を使用(コード実行なし)
Biomni - 完全版Biomni(すべての機能統合)
bでは著者らが新たに作成した「実世界の研究目標を反映するような」ベンチマーク。
実行軌跡を分析したところ、
平均して、Biomniはタスクあたり6から24の異なるステップを実行し、0-4の専門ツール、1-8のソフトウェアパッケージ、0-3のユニークなデータレイクアイテムの組み合わせ
ていることがわかった。
リソース使用はタスクタイプによって異なります:CRISPR摂動スクリーン設計やGWAS因果遺伝子同定などの情報統合タスクは、データベースクエリ(例:KEGG、Reactome)と文献検索(例:PubMed、Google)に大きく依存しますが、マイクロバイオームプロファイリングや単一細胞アノテーションなどのバイオインフォマティクス分析タスクは、データベース使用は最小限ですが、scanpyなどのソフトウェアライブラリを使用した広範なコード実行を伴います。
Results - データ解析
データ解析と仮説の生成ができた。
【3つの解析課題】
-
体温と血糖の解析(458ファイル)
- 30人、数ヶ月分のウェアラブルデータ
- 発見:食後に平均2.19°C体温上昇(個人差大)
- 代謝タイプの違いを示唆
-
睡眠データ解析(227夜分)
- 10人の睡眠パターンを分析
- 発見:水曜日が最も睡眠効率良好、日曜日は低下
- 重要な知見:睡眠の規則性>睡眠時間
-
マルチオミクス統合解析
- 脂質652、代謝物731、タンパク質1,470項目
- 血糖データと統合して相関分析
- 代謝制御の全体像を解明
その後もいろんなことができるよ、と書いてある(割愛)。
ユーザインタフェース
ウェブアプリケーションとして公開したことに言及。
Discussion
以下に言及
- アクション空間のサブセット分野のみで検証しており、それ以外の主要な分野での性能は検証できていない
- アクション発見エージェントが最新論文のみを見ているので、より基礎的なものが漏れている可能性
- 微妙な臨床判断、新規の実験的推論、分析的発明、または深い生物学的思考と統合を必要とする領域ではまだ苦労している
- 強化学習で生物医学推論エージェントを訓練することで、計画と実行における継続的な自己改善が可能になるかも
Methods - Biomni-E1
-
初期フィルタリング:
- 25分野から15分野に絞り込み(創薬・臨床医学に関連)
- 約1,900の反復タスクを特定
-
厳格な選別基準:
- 除外:簡単なコードで実装可能なタスク
- 採用:専門知識が必要な複雑なタスク
-
実装プロセス:
各専門ツールを実装 ↓ 厳密な検証(テストケース必須) ↓ 150の専門ツール完成
ツールの品質保証(5つの必須要件):
- 明確な名前:「blast_sequence_search」など
- 詳細なドキュメント:使い方、パラメータ、出力形式
- LLM最適化出力:AIが理解しやすい構造化ログ
- テストケース:必ず動作確認済み
- 専門性:本当に必要なツールのみ
データベース統合の工夫:
Web APIありの場合:
- 自然言語→LLMが解釈→適切なクエリに変換
- 例:「p53の変異を探して」→ SQL/APIクエリ
Web APIなしの場合:
- 事前にダウンロード・整形
- pandas DataFrameで高速アクセス
コンテナ化環境の利点:
-
依存関係の解決:
- 105のソフトウェアが競合なく共存
- バージョン管理も統一
-
マルチ言語対応:
- Python、R、Bashを同時サポート
- 研究者の好みの言語を使用可能
-
再現性:
- 同じ環境で誰でも同じ結果
- 科学研究に不可欠
Methods - 検証方法
以下の3つで包括的に検証。
- Q&Aベンチマークでの性能評価
- 実際の研究・医療応用を想定したベンチマーク
- 実際の実験(ウェットラボ)での評価と検証
Funder Information Declared
Hitachiの名前がある。何かのプロジェクトがあったのだろうか?
「Hitachi Biomni」で検索してもそれらしい情報はヒットしない。
論文はここまで。
GitHub
主要な注目ポイント (整理by Claude Opus 4)
ここまでのメモをもとに注目ポイントを整理してもらった。
1. 多数のツールの使い分けメカニズム
- 150のツール、105のソフトウェア、59のデータベースという膨大な数をどのように適切に使い分けているか
- 「ツールが増えすぎるとエージェントがツールの使い分けができなくなる」という一般的な問題をどう解決しているか
- マルチエージェントシステムを使わずに、単一の汎用エージェントでどう実現しているか
2. 汎用性と精度の両立方法
- プロンプトチューニングなしに広範なタスクに対応できる仕組み
- 事前定義されたテンプレートに頼らない動的なワークフロー構成の実装
- タスク固有のファインチューニングなしでの高性能を実現する設計
3. データベース統合の実装詳細
- データベースごとの「統一された関数」の具体的な実装
- 特にChEMBLのようなテーブル数が多いデータベースでのText-to-SQLの実装
- Webインターフェースを持たないデータベースのpandas DataFrame化の処理
4. アクション発見エージェントの実装
- 論文から自動でツール・データベース・ソフトウェアを抽出する仕組み
- AI-drivenなアクション空間キュレーションの具体的な実装
5. データ処理と可視化の実装
- パスウェイやヒートマップなど、ライフサイエンス特有の表現形式への対応方法
- 入力データの構造を自律的に補正する仕組みがあるか
- 定められたフォーマットに当てはめているだけか、動的に対応しているか
6. Biomni-A1エージェントアーキテクチャの核心部分
- 汎用エージェントがどのような設計で実装されているか
- 検索システムによる関連ツール特定の仕組み
- ステップバイステップの計画生成の具体的な実装
環境構築
とりあえず動かしてみたい。
が、Python仮想環境がconda管理されていてテンションが下がる。
環境構築のドキュメント。
E1をフルで構築するには10時間と30GBのストレージが必要。なかなかハード。
ローカルでやるのは微妙だな。クラウドに立ち上げるか。
Ubuntu 22.04, 64bitでしか動作検証されていないみたいだし。
一旦読み進める。
最低限AnthropicのAPIキーがあれば動くっぽい。
# Required: Anthropic API Key for Claude models
ANTHROPIC_API_KEY=your_anthropic_api_key_here
# Optional: OpenAI API Key (if using OpenAI models)
OPENAI_API_KEY=your_openai_api_key_here
# Optional: AI Studio Gemini API Key (if using Gemini models)
GEMINI_API_KEY=your_gemini_api_key_here
# Optional: AWS Bedrock Configuration (if using AWS Bedrock models)
AWS_BEARER_TOKEN_BEDROCK=your_bedrock_api_key_here
AWS_REGION=us-east-1
お〜、Biomni-E2へのコントリビュータを募集している。熱いな。
Tutorials
Notebook形式のBiomni 101がある。
さて、とりあえずGCPでフル環境を作ろう。
e2-standard-4で作成。Python仮想環境も地味にストレージ食ったりするので余裕を持って100GBで作成。
% gcloud compute instances create biomni-instance \
--zone=asia-northeast1-a \
--machine-type=e2-standard-4 \
--boot-disk-size=100GB \
--boot-disk-type=pd-standard \
--image-family=ubuntu-2204-lts \
--image-project=ubuntu-os-cloud \
--metadata=enable-oslogin=true \
--tags=biomni-server \
--maintenance-policy=MIGRATE \
--scopes=https://www.googleapis.com/auth/cloud-platform
ただし10時間かかるとのことなので放置して、今日はコードリーディングだけだ。
e2-standard-4で作成。
Tutorial
ざっと読んでみる。
読むまでもなかった。
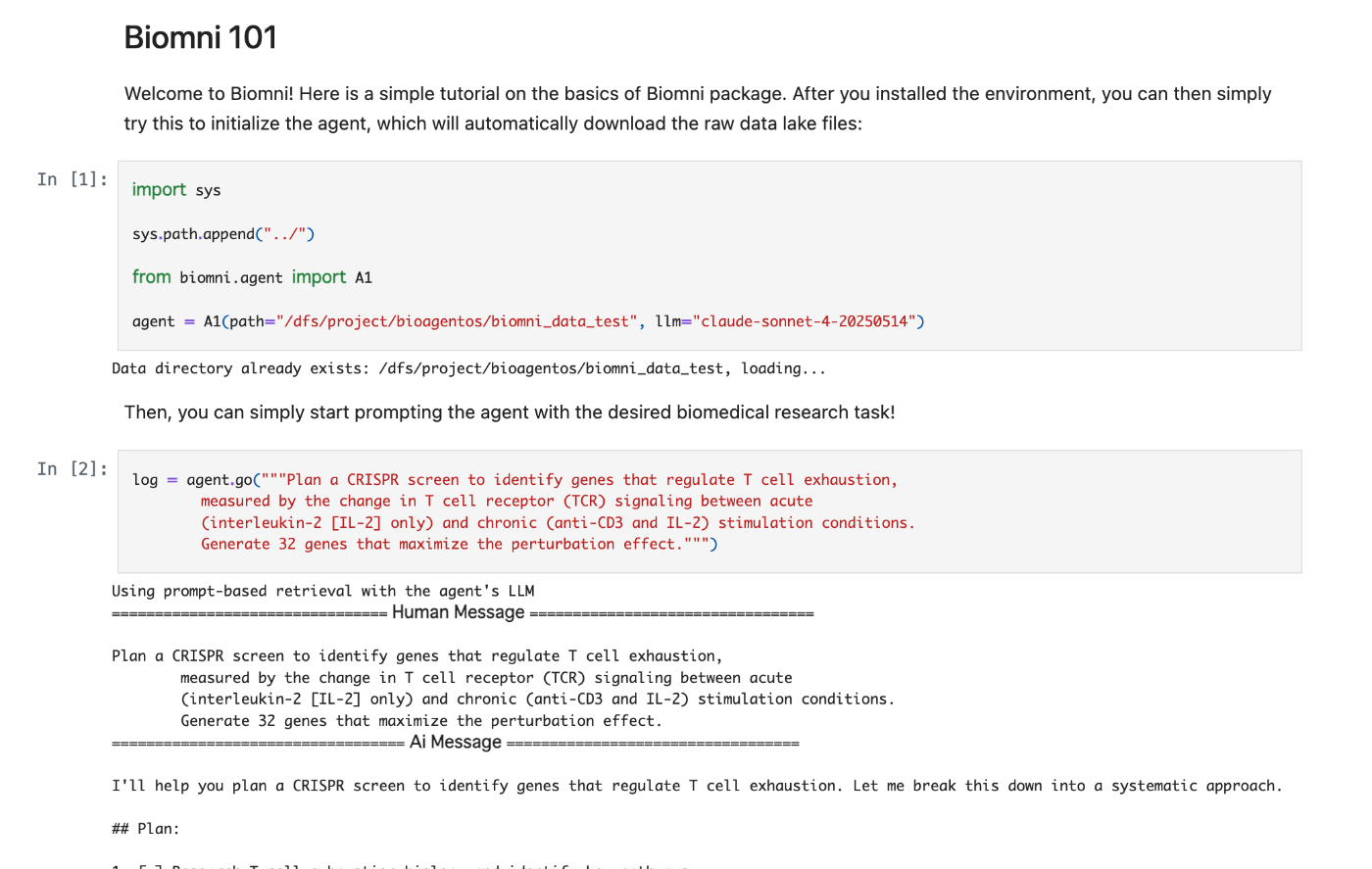
from biomni.agent import A1
agent = A1(path="/dfs/project/bioagentos/biomni_data_test", llm="claude-sonnet-4-20250514")
log = agent.go("""Plan a CRISPR screen to identify genes that regulate T cell exhaustion,
measured by the change in T cell receptor (TCR) signaling between acute
(interleukin-2 [IL-2] only) and chronic (anti-CD3 and IL-2) stimulation conditions.
Generate 32 genes that maximize the perturbation effect.""")
A1を呼び出して、クエリを入力するだけ。
18のメッセージでタスクを遂行していることがわかった。
biomini.agent.A1
GitHubのChat with Copilotで解説してもらう。
LangGraph製のようだ
ツール分析 - PubMed
PubMed検索は
-
Biomni/biomni/tool/literature.py at 0f9dbf925a0512292a2486c87f87152484a9d18a · snap-stanford/Biomni
に定義されている。
def query_pubmed(query: str, max_papers: int = 10, max_retries: int = 3) -> str:
"""Query PubMed for papers based on the provided search query.
Parameters
----------
- query (str): The search query string.
- max_papers (int): The maximum number of papers to retrieve (default: 10).
- max_retries (int): Maximum number of retry attempts with modified queries (default: 3).
Returns
-------
- str: The formatted search results or an error message.
"""
from pymed import PubMed
try:
pubmed = PubMed(tool="MyTool", email="your-email@example.com") # Update with a valid email address
# Initial attempt
papers = list(pubmed.query(query, max_results=max_papers))
# Retry with modified queries if no results
retries = 0
while not papers and retries < max_retries:
retries += 1
# Simplify query with each retry by removing the last word
simplified_query = " ".join(query.split()[:-retries]) if len(query.split()) > retries else query
time.sleep(1) # Add delay between requests
papers = list(pubmed.query(simplified_query, max_results=max_papers))
if papers:
results = "\n\n".join(
[f"Title: {paper.title}\nAbstract: {paper.abstract}\nJournal: {paper.journal}" for paper in papers]
)
return results
else:
return "No papers found on PubMed after multiple query attempts."
except Exception as e:
return f"Error querying PubMed: {e}"
pymedという知らないライブラリを使っていた。210スターと結構マイナーなツールだ。
シンプルな実装。一言で言えばesearchとefetchのラッパー。
クエリにヒットするidを取得、ヒットしたidのxmlを取得、の2つの機能が主。
あとは取得したxmlをArticleクラスにパースしている程度。
これくらいシンプルで十分なのかな?
Biomni内のtool_descriptionでもquery_pubmedについては以下のような情報しかない。
{
"description": "Query PubMed for papers based on the provided search query.",
"name": "query_pubmed",
"optional_parameters": [
{
"default": 10,
"description": "The maximum number of papers to retrieve.",
"name": "max_papers",
"type": "int",
},
{
"default": 3,
"description": "Maximum number of retry attempts with modified queries.",
"name": "max_retries",
"type": "int",
},
],
"required_parameters": [
{
"default": None,
"description": "The search query string.",
"name": "query",
"type": "str",
}
],
},
PubMedの検索方法は内部知識に依存している?
だからクエリを間違えるのか。納得。
URL: https://eutils.ncbi.nlm.nih.gov/entrez/eutils/esearch.fcgi?db=pubmed&term=2024[pdat]&retmax=3000&retmode=xml
Found 1734669 total papers for 2024
上記のURLだと2024という文字列が含まれる文献がヒットしていまうので、1734669件という非常に多くの論文がヒットしてしまっています。
実際には、https://eutils.ncbi.nlm.nih.gov/entrez/eutils/esearch.fcgi?db=pubmed&term=filter[all] AND “journal article”[pt]&retmax=10000&retmode=xml%mindate=2024/01/01&maxdate=2024/12/30というURLである必要があります。
【AI for Science試行録】第2回 特殊な要件を満たす文献セットの取得 - Science Aid Tech Blog
複雑なクエリを正確に実行できるようにするにはtool descriptionを拡充するか、ツールの引数を増やすかするのが良いのではないだろうか。
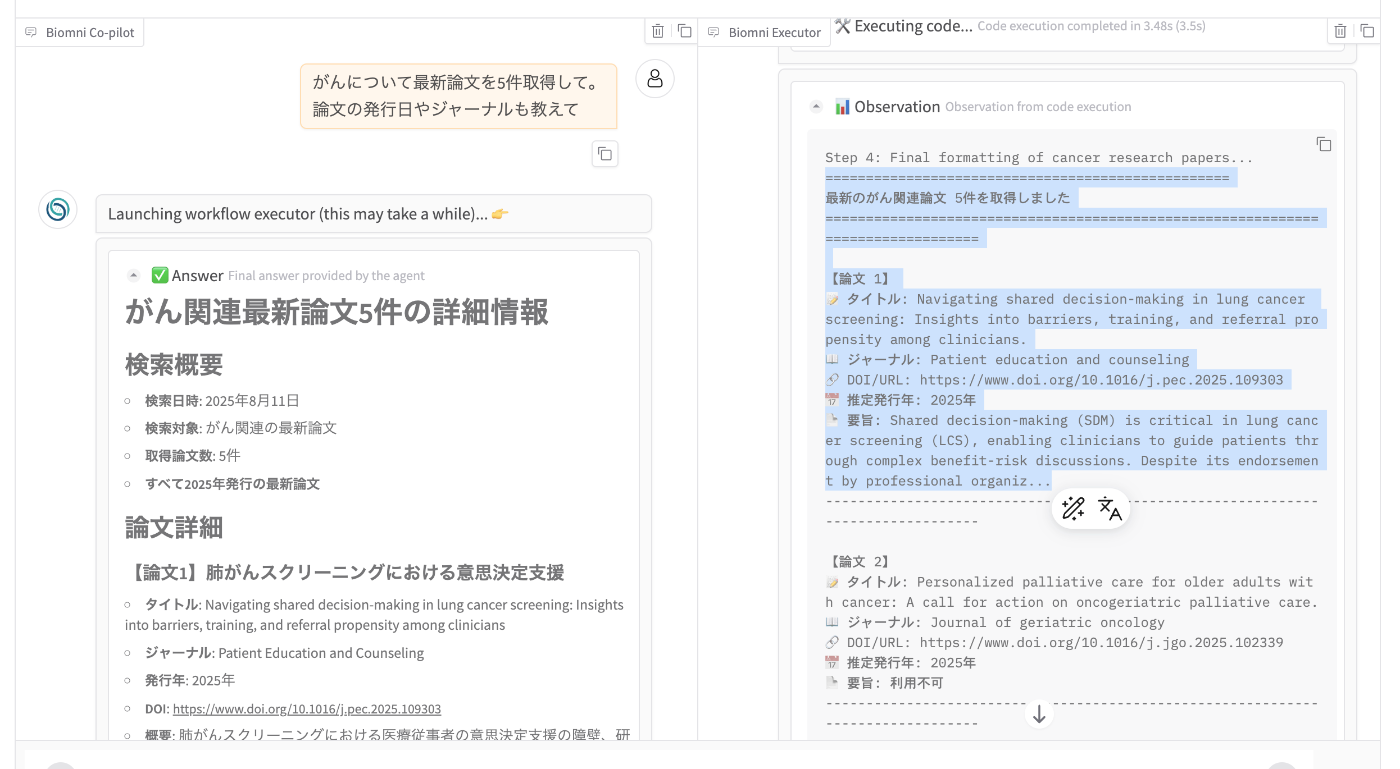
挙動確認。どのようにツール利用とCodeActを併用しているか。
PubMed検索後、単語の頻度分布を解析するよう指示。
以下の挙動を期待
- query_pubmedツールを使って検索
- 取得したデータを解析
方針は期待通り。

だが実際の挙動は予想と異なる。
from biomni.tool.literature import query_pubmed
import pandas as pd
import numpy as np
from collections import Counter
import re
import matplotlib.pyplot as plt
import seaborn as sns
# PubMedで検索実行
print("PubMedで検索を実行中...")
query = '(moyamoya) AND (("2025/8/11"[Date - Publication] : "3000"[Date - Publication]))'
print(f"検索クエリ: {query}")
# 最大50件の論文を取得
results = query_pubmed(query, max_papers=50)
print(f"検索結果のタイプ: {type(results)}")
print("検索結果:")
print(results)
query_pubmedでタイトル、アブストラクト、ジャーナルしか返していない。
results = "\n\n".join(
[f"Title: {paper.title}\nAbstract: {paper.abstract}\nJournal: {paper.journal}" for paper in papers]
)
よって当然論文の日付は取得できていない。