snp distance matrixを系統樹のラベル順に並び替える
はじめに
以前の記事の続きになります。
系統樹のラベル順に並んだデータを利用してsnp distance matrixを並び替えます。
系統樹と並べて可視化に利用するのが最終目標です。
Pythonのpandasを使用して加工していきます。
動作確認 (Hardware)
-MacBook Pro
-チップ Apple M1 (Rosetta2 インストール済み)
-macOS Ventura 13.6.6
-メモリ 16GB
動作確認 (Software)
-Python 3.10.8
-pandas 1.5.3
および
-Python 3.13.3
-pandas 2.2.3
-snp-dists 0.8.2
手順
1.snp distance matrixを作成
core genome SNP treeを作成するためのゲノムのfastaファイルのアライメント済みのファイルを使って、まずsnp distance matrixを作成します。
アライメント結果ファイルはmulti-fasta形式のファイルになっており、seaviewのようなプログラムで開くと、今回使う例ではこのような感じです。
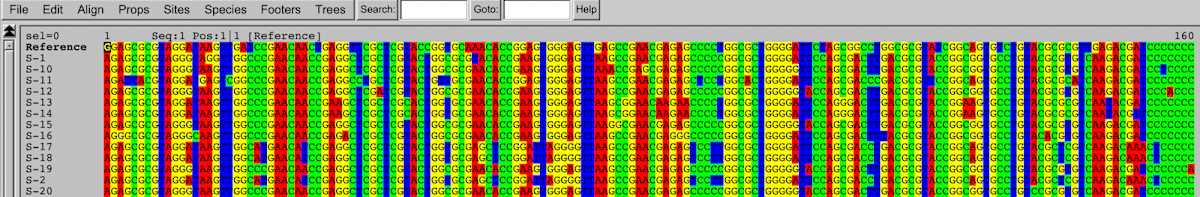
snp distance matrixの作成にはsnp-distsを使用しました。
condaの仮想環境を作成してインストールしました。
仮想環境の作成時にはcondaより処理の速いmambaを使用します。
その仮想環境内に入った後snp-distsをインストールします。
(※自分の環境ではRosettaを使用しています、ARM64での動作は未検証)
mamba create -n snp-dists
conda activate snp-dists
mamba install bioconda::snp-dists
コマンドを確認します。
snp-dists -h

fastaファイルのアライメント結果ファイルを指定して実行します。
snp-dists path_to/alignment_file > matrix.tsv
仮想環境から出ます。
conda deactivate
タブ区切りのmatrix.tsvファイルは距離行列の形になっています。
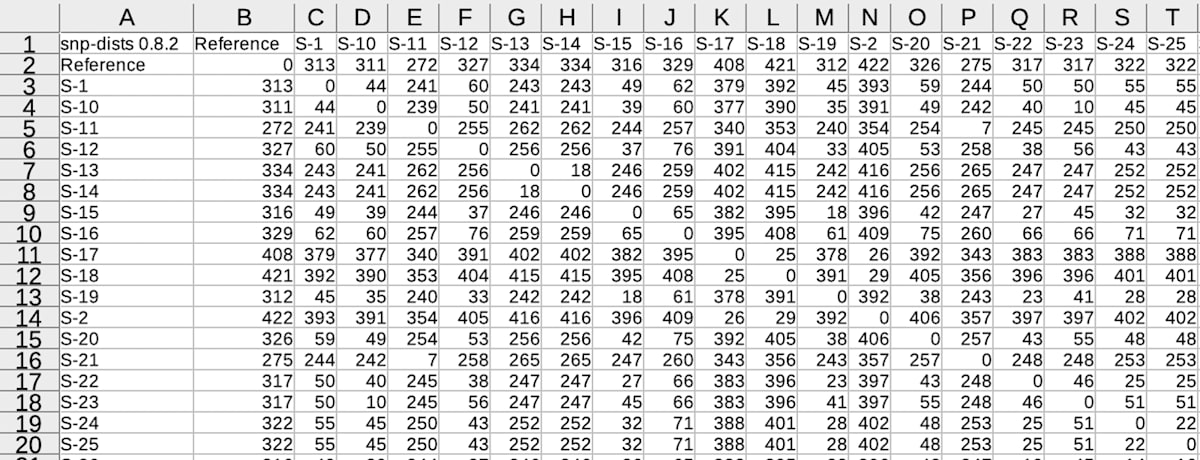
このsnp distance matrixは上からラベル(株名)の名前順に並んでいるため、以前の記事の処理で得たorder.csvファイルを利用してPythonのpandasを使って並べ替えます。
2.pandasでの処理
Pythonで処理していきます。
import pandas as pd
#order.csvファイルの読み込み
df1 = pd.read_csv('path_to/order.csv', header=0)
#後でmatrix.tsvファイルの内容と結合操作を行うため
#カラム名を'snp-dists 0.8.2'にする
df1.columns=["snp-dists 0.8.2"]
#matrix.tsvファイルの読み込み
df2 = pd.read_table('path_to/matrix.tsv', index_col=0)
#各データフレームの'snp-dists 0.8.2'列を結合時のキーとして
#結合方法(how)を'left'に指定して左外部結合を実行
df3 = pd.merge(df1, df2, on='snp-dists 0.8.2', how='left')
一旦df3の中身を確認してみます。
df3
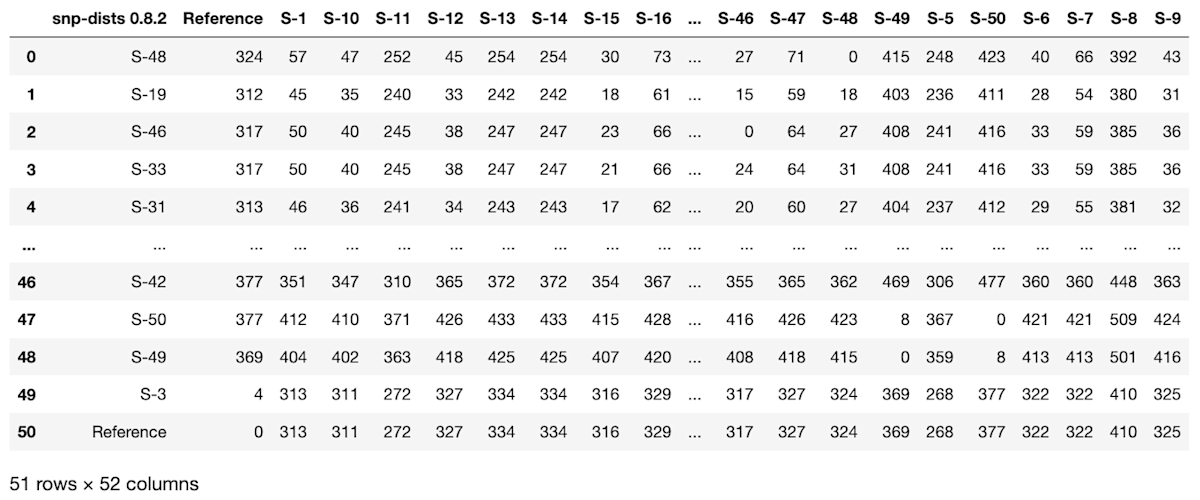
まず、行方向の並び替えができました。
この後転置させてもう一度左外部結合させれば良いと考えたのですが、indexの処理がうまくできなかったため以下の記事のコードを参考にさせていただきました。
処理の続きです。
#転置した結果の先頭行をカラム名、2行目以降をデータとしてデータフレームを作成
df4 = df3.T.reset_index().values
df4 = pd.DataFrame(df4[1:,:], columns=df4[0,:])
#左外部結合を実行
df5 = pd.merge(df1, df4, on='snp-dists 0.8.2', how='left')
df5の中身を確認してみます。
df5

目的の行列ができました。
必要に応じて先頭にある'snp-dist2 0.8.2'の文字列を置換します。
後でヒートマップとして描図する際に残らないようにするには空欄に置き換えます。
df5.rename(columns={'snp-dists 0.8.2': ''}, inplace=True)
csvファイルに書き出して保存します。
df5.to_csv('distance_matrix.csv', index=False)
カレントディレクトリに distance_matrix.csv ファイルができます。
おわりに
もっとスマートな方法はあるのだろうと思いますが...。
Discussion