TreeTimeでtime tree作成の練習
はじめに
TreeTimeを使ってtime tree(time-scaled phylogenetic tree)を作成する練習の記録です。
TreeTime: Maximum-likelihood phylodynamic analysis
解析環境 (Hardware)
-MacBook Pro
-チップ Apple M1 (Rosetta2 インストール済み)
-macOS Sequoia 15.5
-メモリ 16GB
解析環境 (Software)
-TreeTime 0.11.4
-ncbi-acc-download 0.2.8
-MAFFT 7.525
-IQ-TREE 3.0.1
手順
1. データの用意
Nature Scientific Reportsに掲載の、こちらの文献に記載されている株を例にとります。
Analysis of complete genomes of the rubella virus genotypes 1E and 2B which circulated in China, 2000–2013
こちらの文献中のSupplemental Table 2にある32株の風疹ウイルスの配列データを使用します。
TableにGenbankのアクセッション番号が記載されているので、以前の記事に記した方法でncbi-acc-downloadを使用してfastaファイルをダウンロードします。
ダウンロードする際のファイル名を「Genotype.国.年」に揃えました(この形式で名前が重複する株には末尾に'.1''.2'を付加しました)。
株リスト
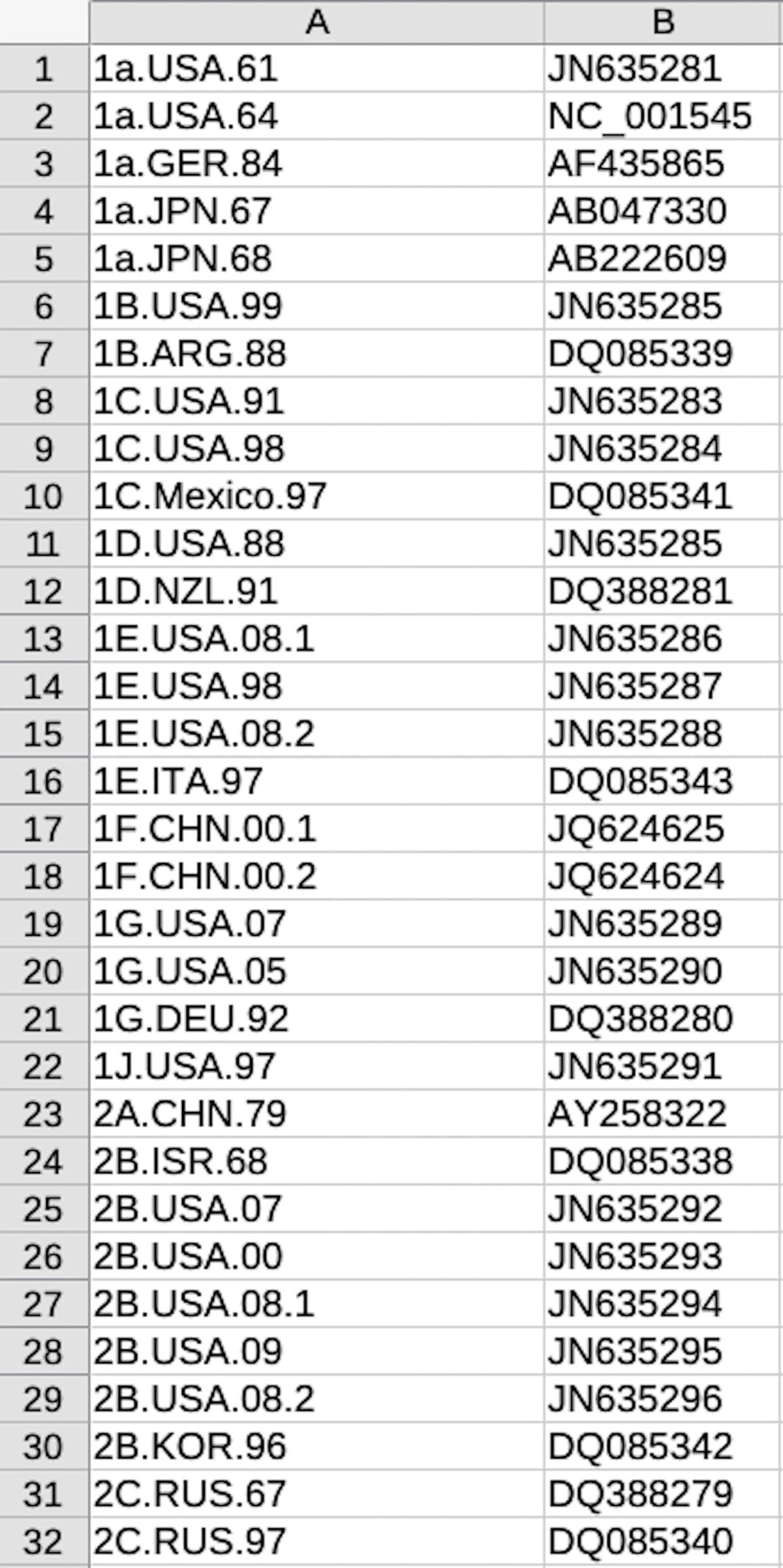
風疹ウイルスのComplete Genomeのサイズは約10Kbpなので、fastaファイルのサイズもおおよそ10KBです。
fastaファイル
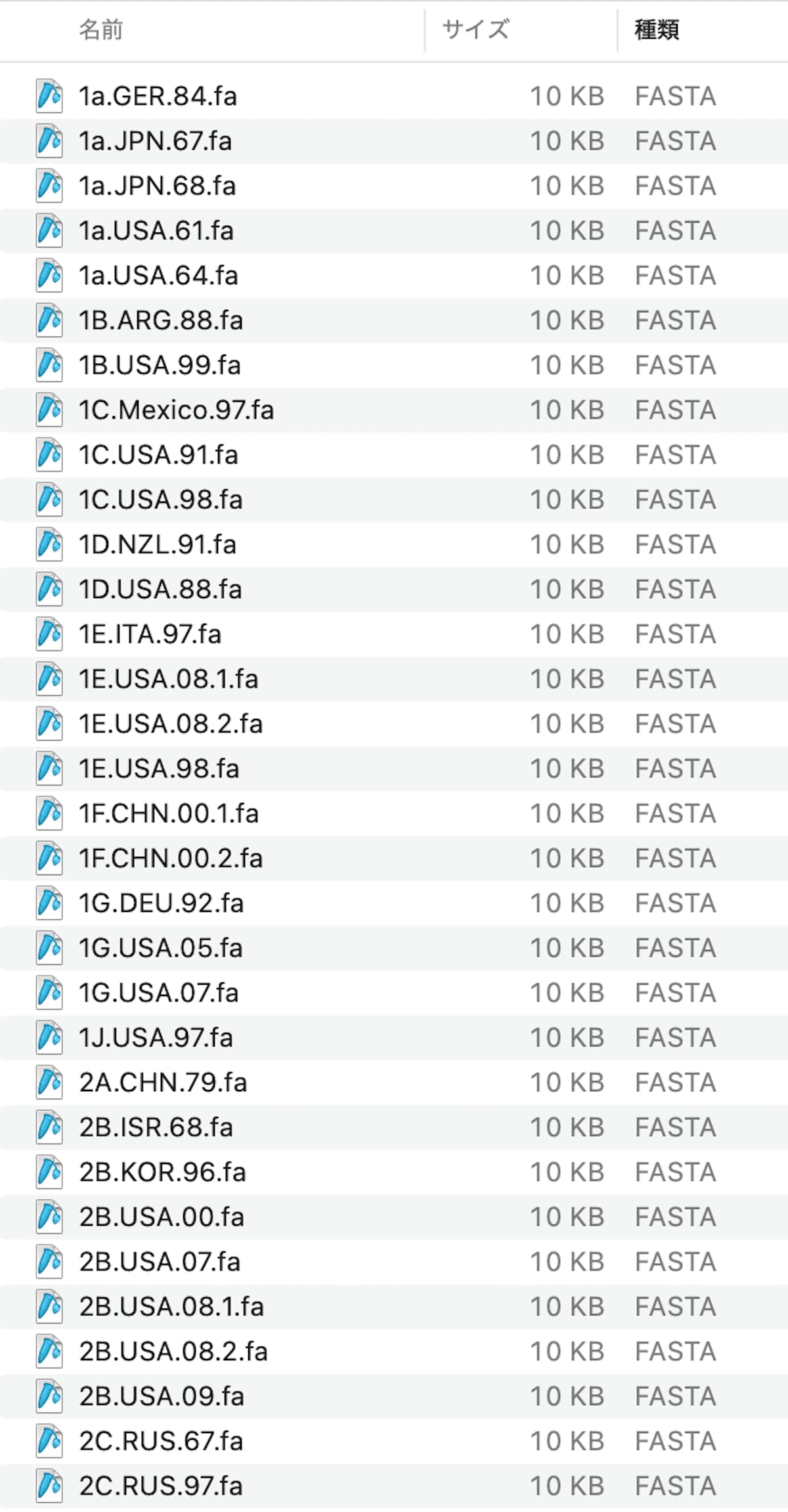
各fastaファイルのヘッダ行を、ファイル名と同じものに変更しました(アライメントファイルや系統樹での表示名になります)。
ヘッダ行変更例

↓

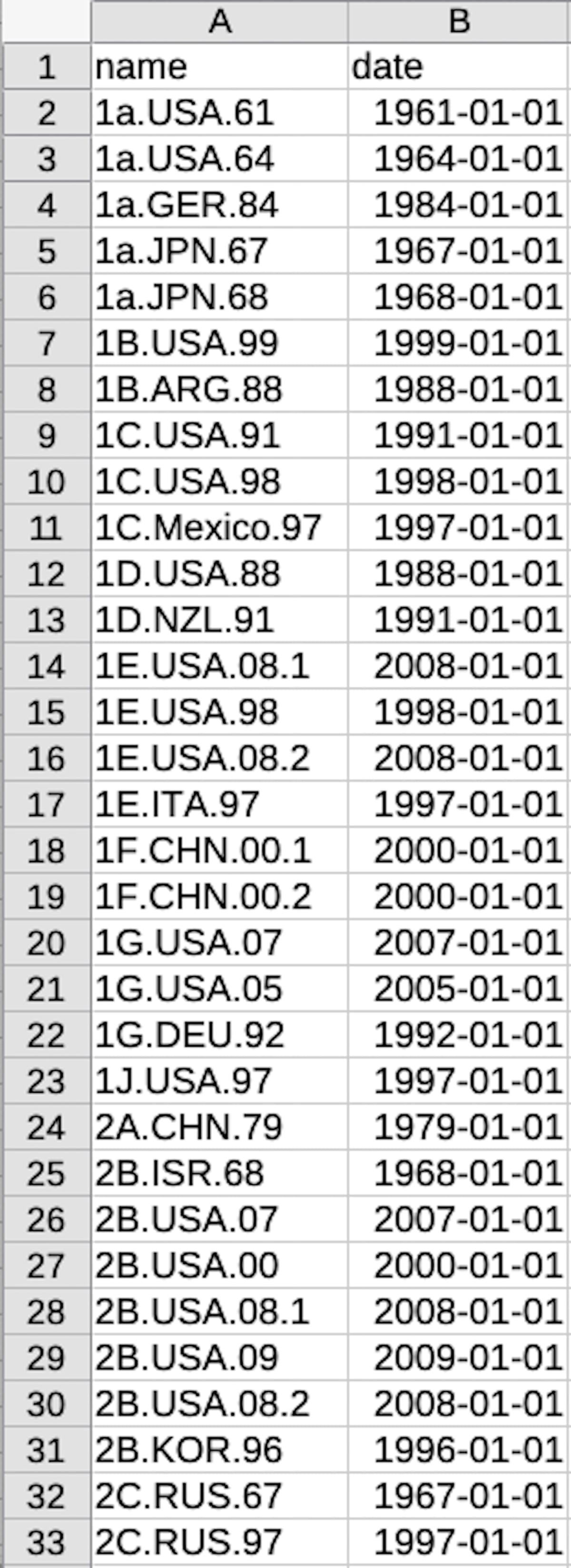
「dateファイル」を用意します。
1列目に列名を「name」として株名を記載して(fastaファイルのヘッダ行に記載の内容と揃える)、2列目に列名を「date」として株の分離年月日を記載します。
今回の例では月・日はすべて仮に1月1日としました。
csvファイルで保存します。
dateファイル(date.csv)
2.プログラムのインストールと実行
先日、以下の記事を参考にCondaの環境を再構築しました。
Macでのバイオインフォマティクス解析の環境構築 2025版
よって、各プログラムのインストールはARMベースのターミナルから起動して、仮想環境作成時にRosettaの利用が必要なケースでは--platform osx-64 を指定して作成する形になります。
これまでのターミナル
$arch
i386
環境を再構築した後のターミナル
$arch
arm64
Rosettaを介してターミナルを起動している場合は--platform osx-64 のオプションを付けずに仮想環境を作成することになると思います。
2-1. MAFFTのインストールとマルチプルアライメントの実行
マルチプルアライメントの実行にMAFFTを使用します。
MAFFT - a multiple sequence alignment program
#仮想環境の作成とインストール
mamba create --platform osx-64 -n mafft
conda activate mafft
mamba install bioconda::mafft
対象のfastaファイルを集めているディレクトリに移動して、各株のfastaファイルを結合して1つのmulti-fastaファイルを作成した後、MAFFTを実行します。
cd path_to/fasta_folder
cat *.fa > mafft_input.fasta
#MAFFTを実行
mafft --auto mafft_input.fasta > output.aln
conda deactivate #仮想環境から出る
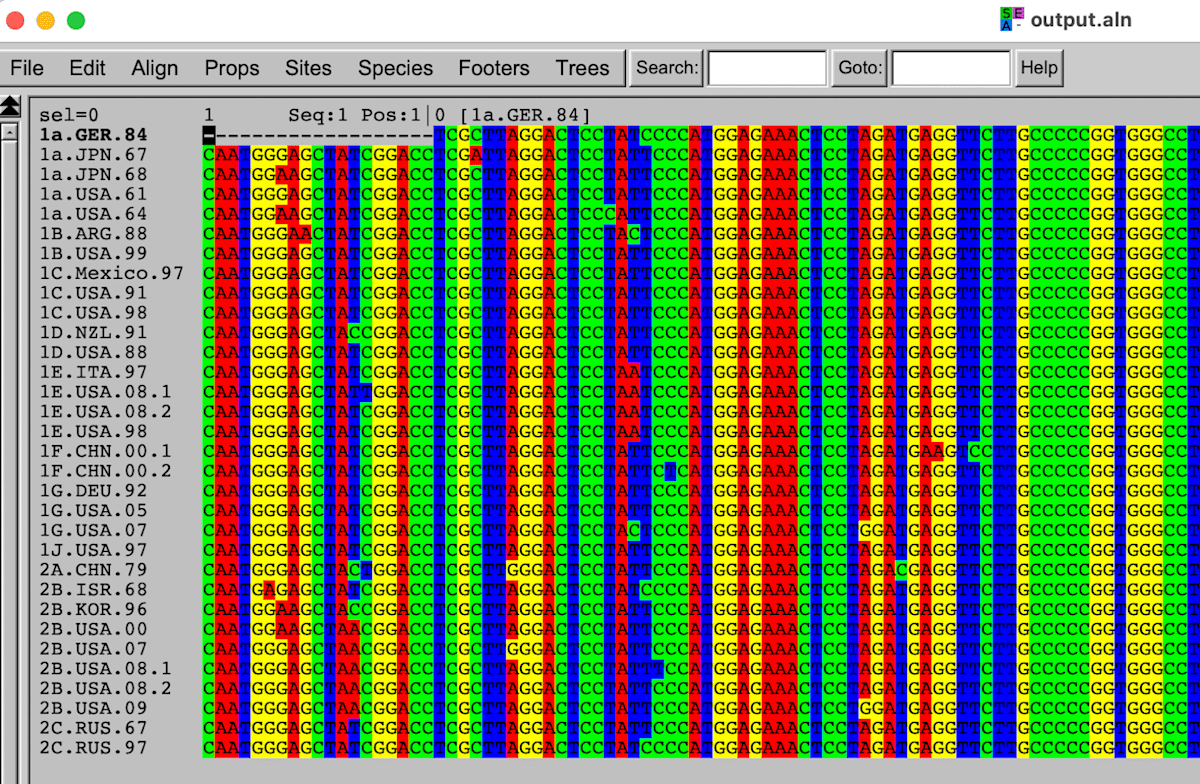
マルチプルアライメントの出力ファイル(output.aln)はmulti-fasta形式のファイルで、seaviewのようなプログラムで開いてアライメント結果を確認できます。
seaviewで開いた例
2-2. TreeTimeのインストールと系統樹作成
2-2-1. TreeTimeのインストール
#仮想環境の作成とインストール
mamba create --platform osx-64 -n treetime
conda activate treetime
mamba install bioconda::treetime
2-2-2. IQ-TREEのインストール
TreeTimeの実行時に--treeオプションでNewick形式の系統樹ファイルが必要になりますが、TreeTimeのDocumentationによると「ファイルが指定されていない場合は(インストールされていれば)fasttree、iqtree、あるいはraxmlを使用して系統樹の作成を試みる」とのことでしたので、TreeTimeの仮想環境内にIQ-TREEもインストールしておきます(もちろん、事前にNewick形式のファイルを別途用意しておいてからTreeTimeを実行しても可です)。
#TreeTimeの仮想環境に入った状態でインストールを実行
mamba install bioconda::iqtree
2-2-3. TreeTimeの実行
#TreeTimeの仮想環境に入った状態で実行
treetime --aln output.aln --dates date.csv --outdir treetime_out \
--plot-tree treetime.png --tip-labels
conda deactivate #仮想環境から出る
- --aln:alignment file (fasta)
- --dates:csv file with dates for nodes with 'node_name, date' where date is float (as in 2012.15) or in ISO-format (YYYY-MM-DD). Imprecisely known dates can be specified as '2023-XX-XX' or [2013.2:2013.7]
- --outdir:directory to write the output to
- --plot-tree:filename to save the plot to. Suffix will determine format (choices pdf, png, svg, default=pdf)
- --tip-labels:add tip labels (default for small trees with <30 leaves)
Newick形式のファイルを用意していない状態で実行すると、以下のようなメッセージが出てiqtreeが動きました。
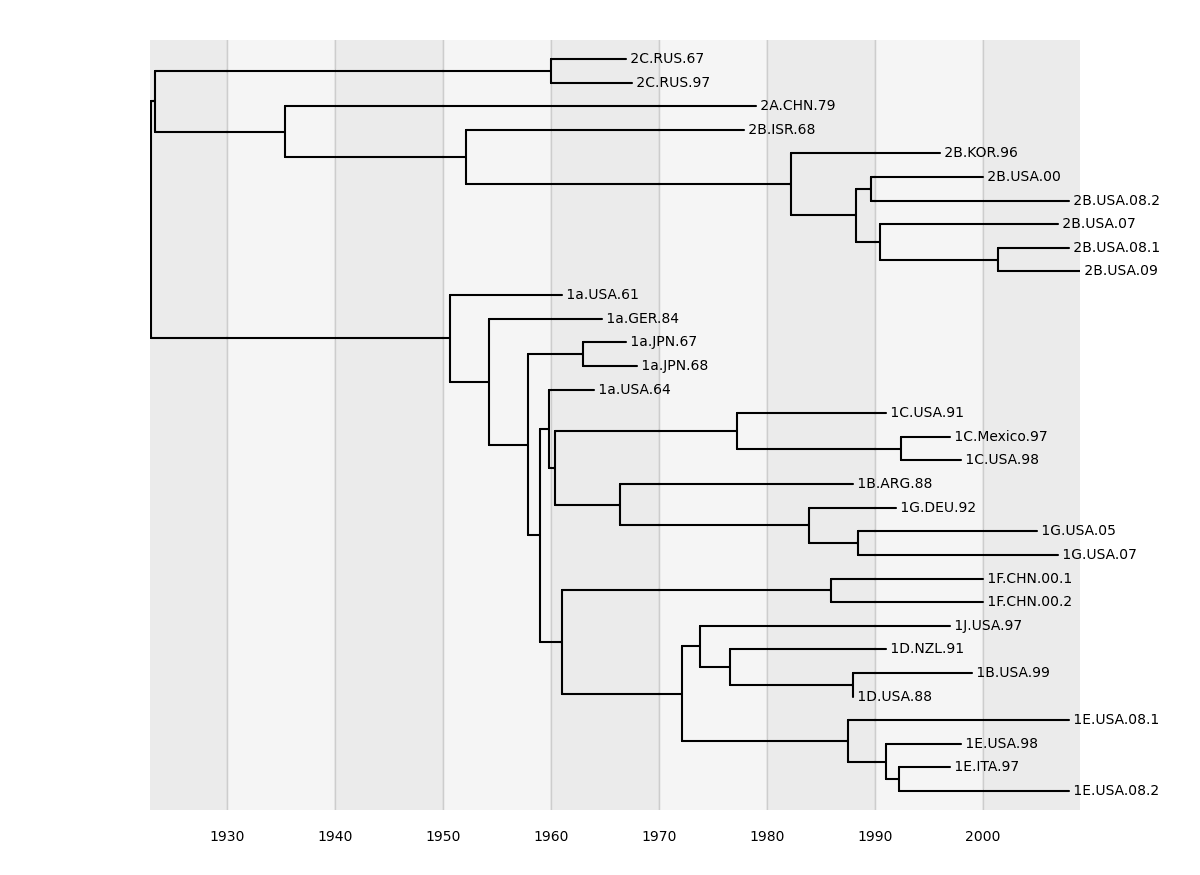
最終的にtime treeのPNG画像は100dpiで出力されました。
treetime.png
おわりに
留意点:
- 株名が長すぎると、分離年が新しめの株のラベルが系統樹の画像から見切れてしまうようです(なので、今回の例では短めにしました)
TreeTimeには他にもオプションがたくさんあるのでほんの一例ですが、使い方の参考になれば幸いです。
Discussion