[forceatlas2]error: unknown file type '.pxd' (from 'fa2/fa2util.pxd')
背景
グラフ描画のためのレイアウトアルゴリズムには様々なものがありますが,ForceAtlas2 (fa2) (Github)は高速で正確といわれています. bioinformaticsの分野では,single-cell RNAseq解析の最もメジャーなプラットフォームとしてScanpyをよく使用しますが,描画の際には内部でfa2が動くことが想定されています. しかしながら,ご経験の方も多いかと思うのですが,新たにScanpyの環境を構築しようとするとfa2のインストールに失敗すると思います. ネットで検索するとpythonのバージョンアップが原因であるとされています. グラフ描画については他のアルゴリズムもあるため,Scanpy自身は動作します. なのでこれまでfa2を諦めるか古いバージョンのコンテナを使用していました. ところが現環境でfa2でグラフ描画したいモチベーションが再び生まれたので,この時代Claudeに聞けばいいかと思い聞いたらあっさり解決したという話です.
環境
OS: Debian GNU/Linux 12
Python: 3.10.12
Cython: 3.0.10
事象
適切にcythonはインストールの上,pip install fa2とすると以下のエラー.
...
running build_ext
building 'fa2.fa2util' extension
error: unknown file type '.pxd' (from 'fa2/fa2util.pxd')
...
結論
git clone https://github.com/bhargavchippada/forceatlas2.gitの上,setup.pyを以下のように編集.
# 19行目
- from Cython.Build import build_ext
+ from Cython.Build import cythonize
# 28-30行目
if USE_CYTHON:
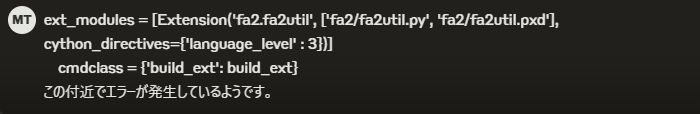
- ext_modules = [Extension('fa2.fa2util', ['fa2/fa2util.py', 'fa2/fa2util.pxd'], cython_directives={'language_level' : 3})]
- cmdclass = {'build_ext': build_ext}
- opts = {"ext_modules": ext_modules, "cmdclass": cmdclass}
+ ext_modules = cythonize([
+ Extension('fa2.fa2util', ['fa2/fa2util.py'],
+ cython_directives={'language_level': 3})
+ ])
+ opts = {"ext_modules": ext_modules}
編集後,インストール
cd forceatlas2
pip intall .
(経緯) Claude3.5 Sonnetに聞いてみた

最初の回答としては,Cythonがインストールされているかなどを聞かれたがうまくいかなかった.
エラーから判断すると,setup.pyのbuild_extで詰まっている様子だったので,そこを具体的に聞いてみた.
すると,

なるほど. ただ,4.については,きちんと展開されるようにはなっていたので,無視しました.
感想
fa2を諦めていたのが数年前で,使いたい時には古いPythonバージョンのコンテナを使用していました. LLMにエラー対応をお願いするのが当たり前になった今,改めて挑戦するとあっさり解決. いい時代です.



Discussion