インシリコ創薬の大規模化合物ライブラリの構築を論文に沿ってやってみる。【in silico創薬】
本記事では大規模化合物ライブラリの構築を行います。本記事ができるようになると、In silicoスクリーニングに必要な化合物ライブラリの作成の仕方がわかるようになります。
動作検証済み環境
Windows 11 Home, 13th Gen Intel(R) Core(TM) i7-13700, 64 ビット オペレーティング システム、x64 ベース プロセッサ, メモリ:32GB
本論文は以下の論文のフォローになります。
標的の選定方法からスクリーニング、スクリーニングした薬物候補化合物の毒性、物性予測、MD シミュレーションまでしており、ほぼ無料のツールですることができます。in silico創薬の基本が詰まっているので、一緒に勉強していきましょう。
宣伝
本記事を見てくださり、ありがとうございます。
インシリコ創薬についてより学びたい方は
拙著 で学び、さらに色々な方法で新薬探索を楽しんでいただければと思います!
また化合物の評価を行いたい場合は を見ていただければと大変嬉しいです。
In silico創薬とその流れ
In silico創薬、in silicoスクリーニングとは
インシリコ創薬(in silico drug discovery)は、コンピュータを駆使しし、シミュレーションやデータ解析を用いて新薬を設計・発見する手法です。
これにより、従来の実験的な方法に比べてコストと時間を大幅に削減できるとされています。
その中でもin silicoスクリーニングは、膨大なデータベースから有望な候補物質を迅速に特定し、その効果や副作用を予測することで、創薬の初期段階での効率化が図られます。
一見難しそうなin silico創薬ですが、現在では様々なアプリケーションやwebサイトがあり、それらを駆使すれば、誰でも簡単に創薬をすることもできます。本記事では、それらのアプリケーションを駆使し、in silico創薬を行った論文をもとに、手法をわかりやすく説明していきます。
In silico創薬の流れ
- ターゲット選定、準備:疾患の原因となる分子(ターゲット)を特定し、その3D構造を解析します。
- 化合物ライブラリの構築:in silicoスクリーニングに使用する化合物集団(=ライブラリ)を作成します。
- in silicoスクリーニング:コンピュータ上で数百万の化合物を対象に、ターゲット分子との結合親和性を評価します。
- ドッキングシミュレーション:候補化合物がターゲット分子にどのように結合するかを詳細にシミュレーションします。
- 分子動力学シミュレーション:候補化合物とターゲットの相互作用を動的に解析し、安定性や効果を予測します。
- 物性、毒性評価:過去のデータを用いて、新たな候補物質の特性や副作用を予測するモデルを構築します。
- (実験的検証):in silico解析で得られた候補物質を実験室で合成し、実際の効果や安全性を検証します。
本記事は2.化合物ライブラリの構築を解説します。
以下の論文をフォロー指定いきます。
該当箇所はMaterial and methodの「CMNPD preparation」の部分です。
本記事で用いられるツール一覧
CMNPD: 化合物ライブラリー作成に使用するデータベース
FAFDrug4 server:化合物ライブラリから薬剤として適しているものをフィルタリング
CMNPDからの抽出
CMNPD(Comprehensive Marine Natural Products Database)は、海洋天然物研究に特化したオープンアクセスのデータベースです。31,000以上(本論文では47000と書いてあるが、その値はどこ由来なのか不明)の化学物質を収録し、物理化学的および薬物動態的特性、生物活性データ、分類学、供給元生物の地理的分布、詳細な文献引用などの情報を提供します。
CMNPDは、構造の再確認、リード化合物の発見、構造活性相関のデータマイニング、化学的生態学の調査に役立ちます。
ユーザーは、ウェブインターフェースを通じてアクセスでき、研究者や広範な科学コミュニティに無料でデータ共有プラットフォームを提供しています。これにより、海洋由来の新規リード化合物の発見や薬剤開発が促進されます。
CMNPDにアクセスしてください。
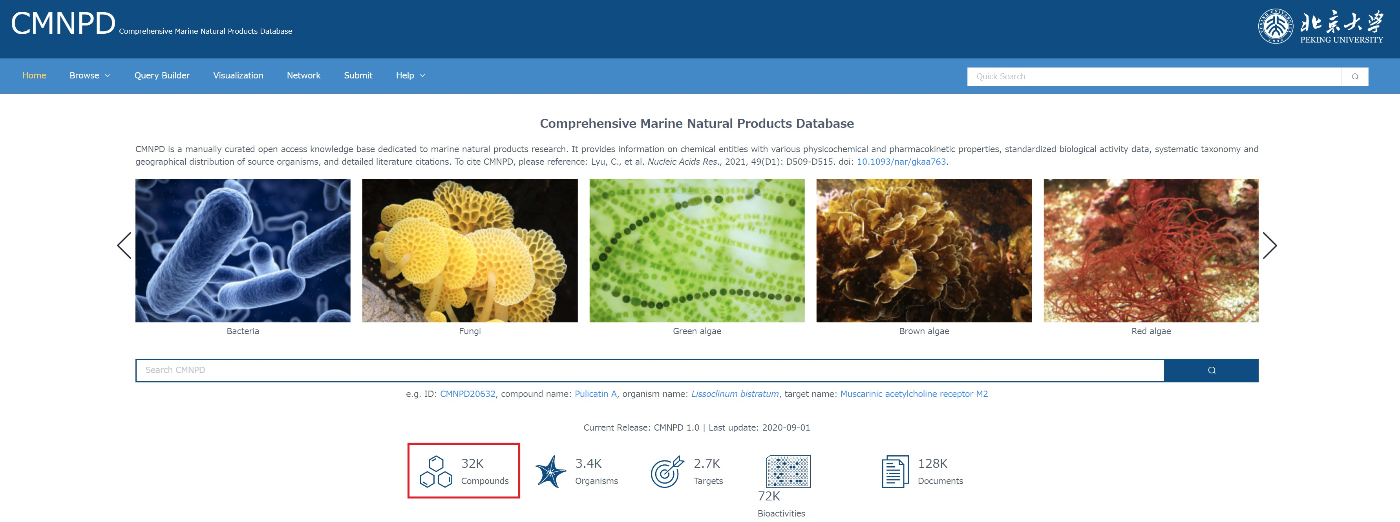
32K compoundsを押すとCMNPDにあるすべての化合物が見れます。
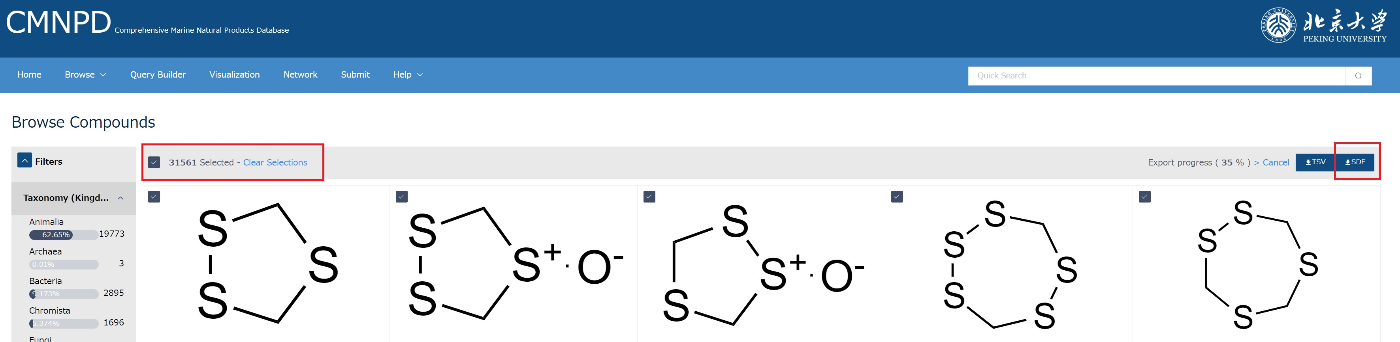
今回はとりあえずすべての化合物の情報がほしいので、チェックをいれて、SDFファイルをダウンロードしてください。
CMNPD-export として保存されると思います。
FAFDrug4 serverによるフィルタリング
FAF-Drugs4は、化合物コレクションの準備と注釈を行うオンラインサーバーで、ドラッグディスカバリーや化学生物学の研究を支援します。物理化学的性質の予測、有害化学群(トキシコフォア)の検索、ユーザー設定のフィルターによる化合物のフィルタリングを行います。新しいFAF-QEDサービスにより、薬剤適合性の評価も可能です。迅速な処理とカスタマイズ可能なフィルターにより、質の高い化合物を効率的に選定し、コストと時間を節約します。
ここではFAF-Drugs4によって、より薬物化合物になりそうなものをフィルタリングしていきます。
FAFDrug4 serverにアクセスしてください。
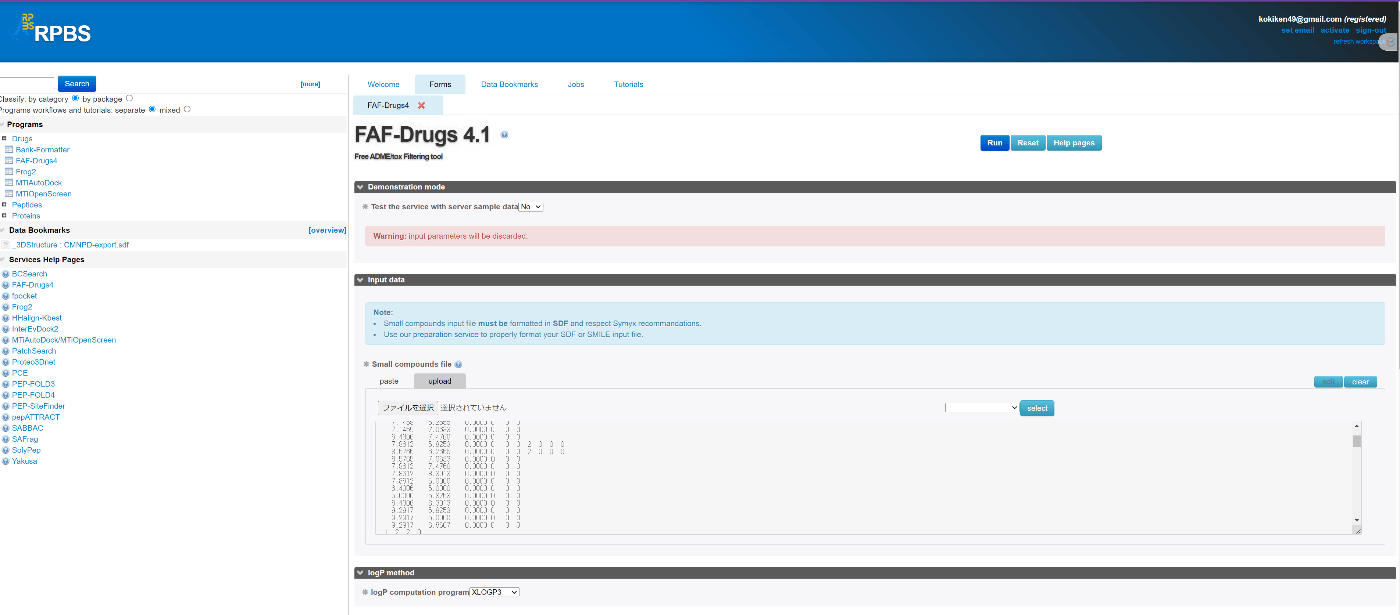
先ほど保存したCMNPD-exportファイルをアップロードして、selectを押してください。
論文に沿って、以下を設定してください。
In house [*] and published physchem filters [+] → Drug-like soft
Filter undesirable substructures moieties → Yes (これがToxicophoresによるフィルタリングになります)
Filter Pan Assay Interference Compounds (PAINS) Filter A, B, C → Yes
Lilly MedChem Rules (only detection, no triage)→ regular
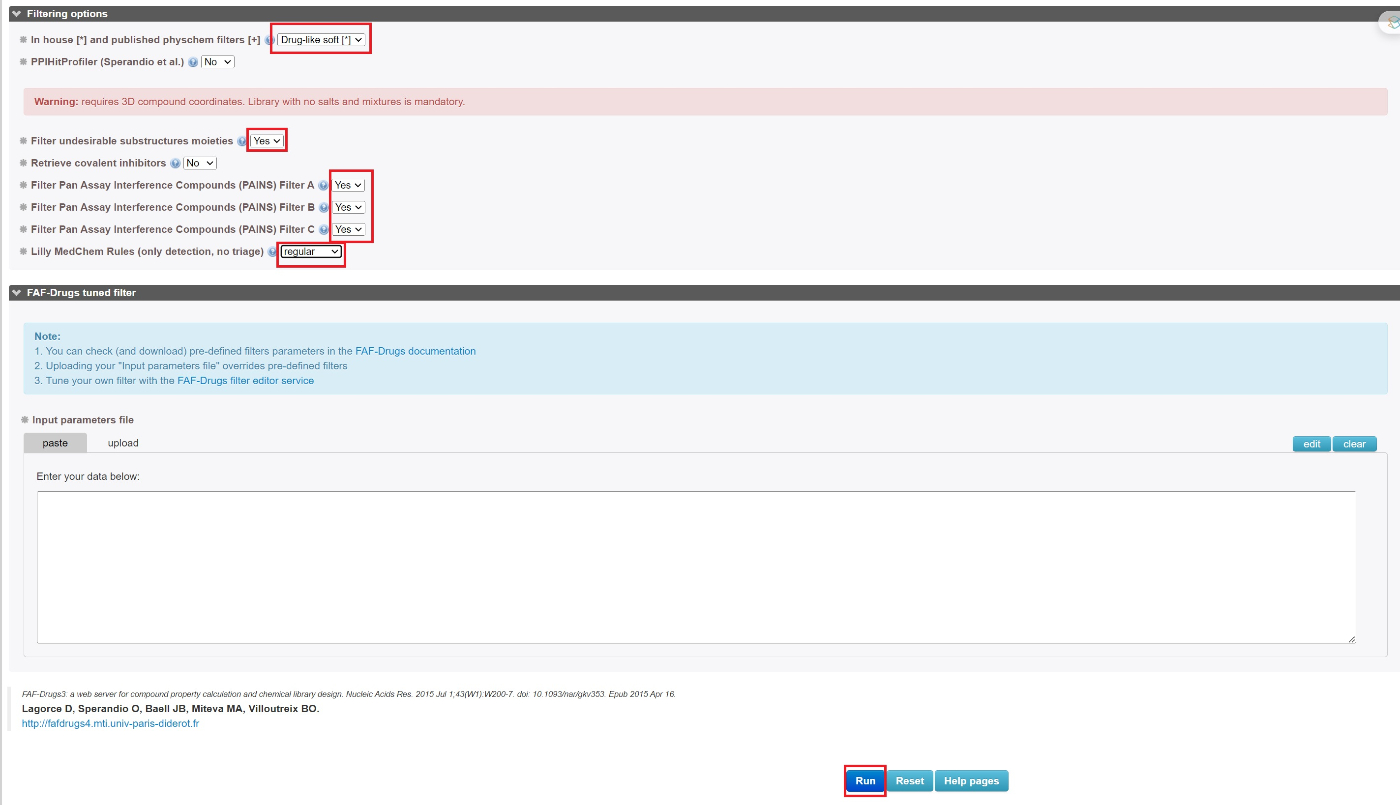
(最後にRunを押すと、activation keyがないのに、activation keyが求められたりしますが、そのときはメールアドレスを変えるなどしてみてください。)
終わると以下のようにJobsにチェックが入ります。
3時間くらいで終わります。
また以下のような結果が表示されます。
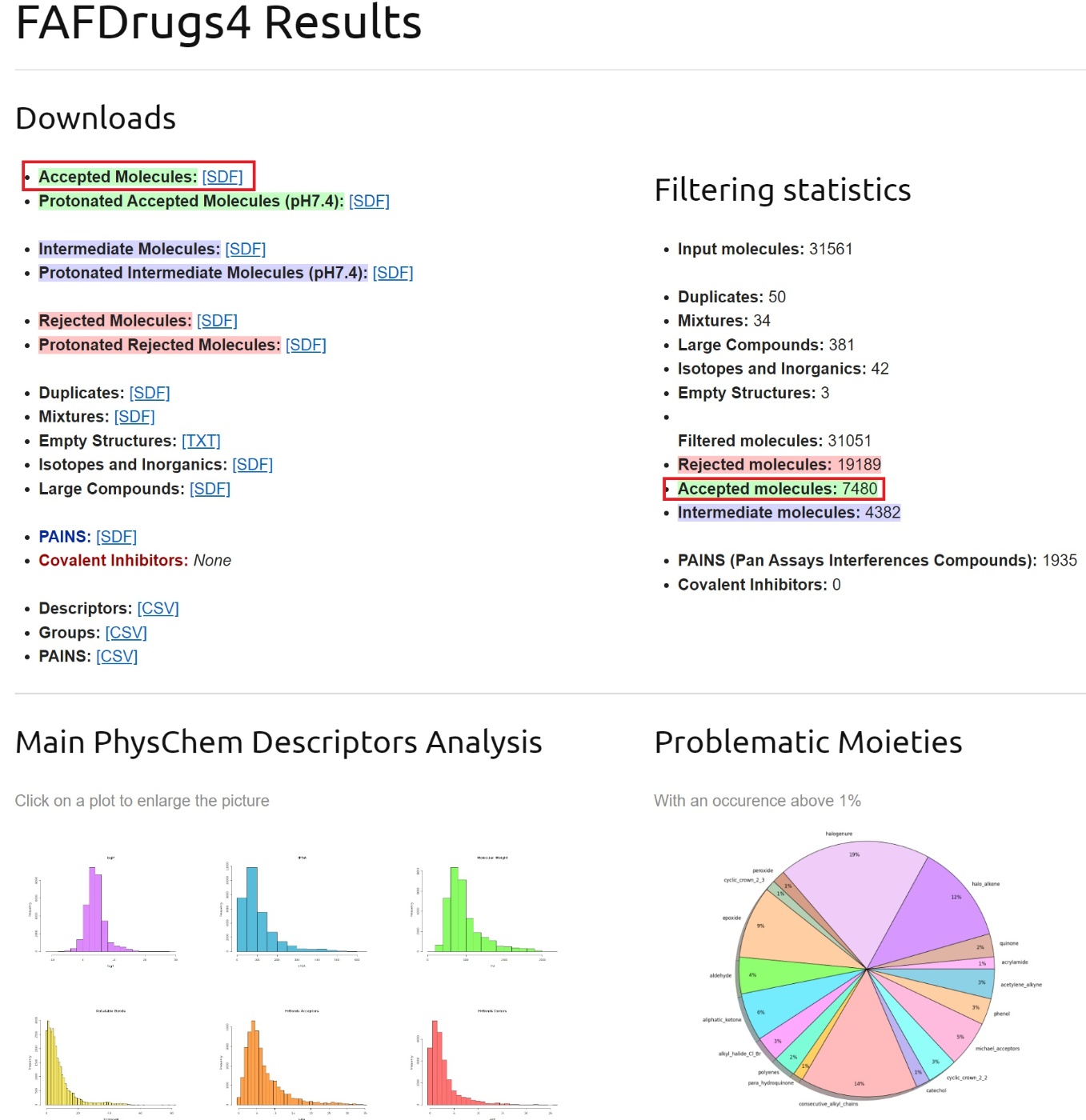
Accepted moleculesが7480となっているので、7480種類の化合物ライブラリーに減りました。
(大体4分の1くらい)
Accepted MoleculesのSDFファイルをダウンロードしてください。
(新しいwindowが開くので、コピーして、入力に使ったsdfファイルにコピペして、CMNPD-filteredとして保存してください)
新しいウィンドウが開くので、すべてコピーしてください。

CMNPDからexportしたsdfファイルCMNPD-export をコピーし、CMNPD-filtered という新しいファイルを作って、そこにコピーしておいた情報をペーストしてください。
これで化合物ライブラリの構築は完成です。
さて、結果論ではありますが、とりあえずこの論文をフォローすることが目的なので、
CMNPD-filteredにこの論文で取れてきた以下の化合物があることを確認します。
CMNPD31561→なし
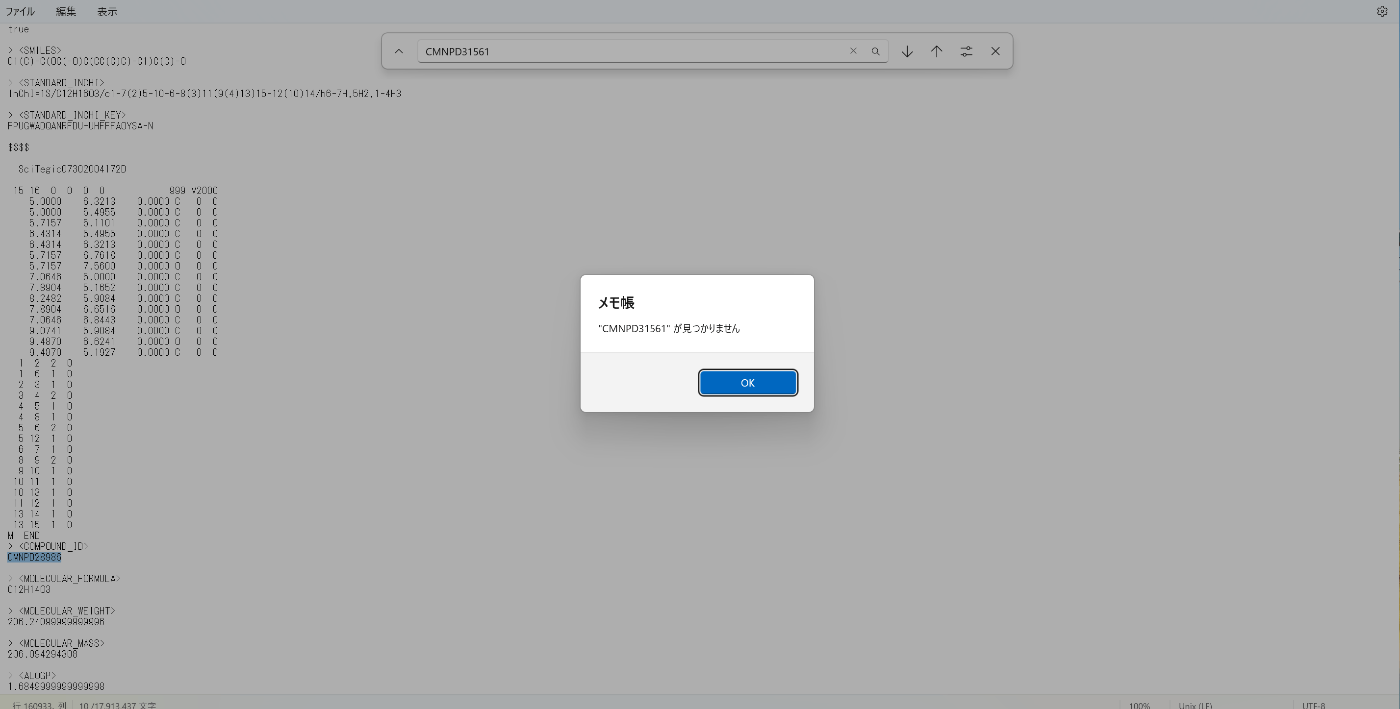
CMNPD28986→あり

CMNPD28985→なし
という結果になりました。
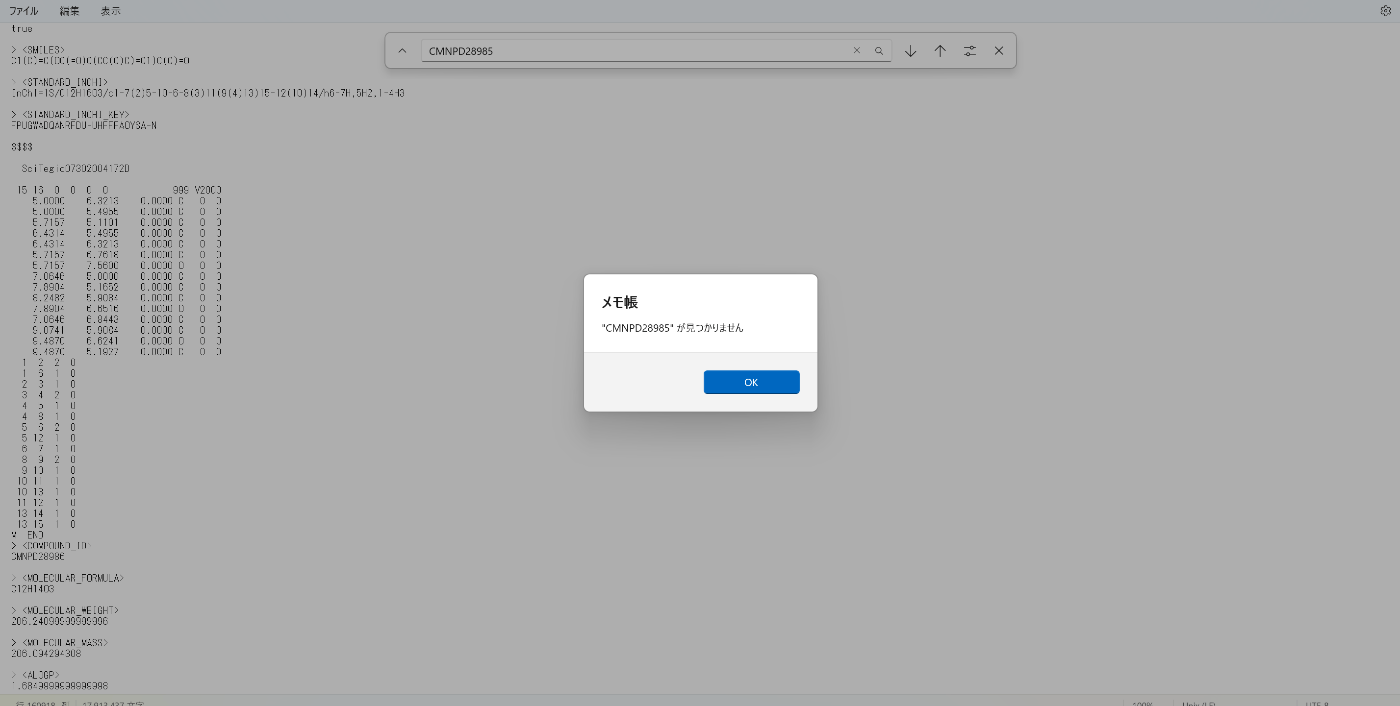
予期に反して、化合物が二つここでスクリーニングされて抜け落ちていることがわかります。
論文では詳細なやり方が書かれていないので、フォローできませんが、
場合によってはFilteringのオプションをもう少し緩やかにしてみてください。
(ソフトウェアのバージョン違いという可能性もありますので、このライブラリで次のインシリコスクリーニングを行なっていきます。)
最後に
いかがでしたでしょうか?化合物ライブラリを構築できたので、次からスクリーニングを試していきます!今回はCMNPDというデータベースを使いましたが、ほかにもたくさんデータベースはあるので、いろいろなDBを試してみてください!
参考文献
宣伝
本記事を見てくださり、ありがとうございます。
インシリコ創薬についてより学びたい方は
拙著 で学び、さらに色々な方法で新薬探索を楽しんでいただければと思います!
また化合物の評価を行いたい場合は を見ていただければと大変嬉しいです。
Discussion
CMNPDからSDFファイルを抽出する際に、
Request failed with status code 500
が表示され、ダウンロードできません。
どうしたらいいでしょうか。
こちらサーバー側の問題のようですので、しばらく待ってみて再度試していただければと思います。
2025/02/10時点でも本問題は解決していません。
以下に他のライブラリの紹介をしましたので、こちらを参考にしてライブラリを構築していただければと思います。