Open4
Staggered DID の実装について
Staggered DIDについてまとめる。
参考文献
Rでの実装
did というパッケージが使いやすそう。
att_gt では、介入開始が同じ値のグループごとに、介入前後の係数が推定される。
library(did)
data(mpdta)
out1 <- att_gt(yname="lemp",
tname="year",
idname="countyreal",
gname="first.treat",
xformla=NULL,
data=mpdta)
summary(out1)
群 x 時間のパネルデータを用意する必要がある。
gnameに渡すカラムの形式に注意。
- y_name: 被説明変数
- tname: 時間を表す変数
- idname: 個体を表す変数。数値に変換する必要あり。
- gname: 介入開始の時間が格納された変数。
- 介入群の場合、全ての時間tについて介入開始の時間が格納される。
- 介入がない群の場合、全ての時間tについて0が格納される。
- xformla: 説明変数
ggdid というメソッドを使えば、係数の時系列推移(event studyでよく見るやつ)をplotしてくれる。
ggdid(out1)
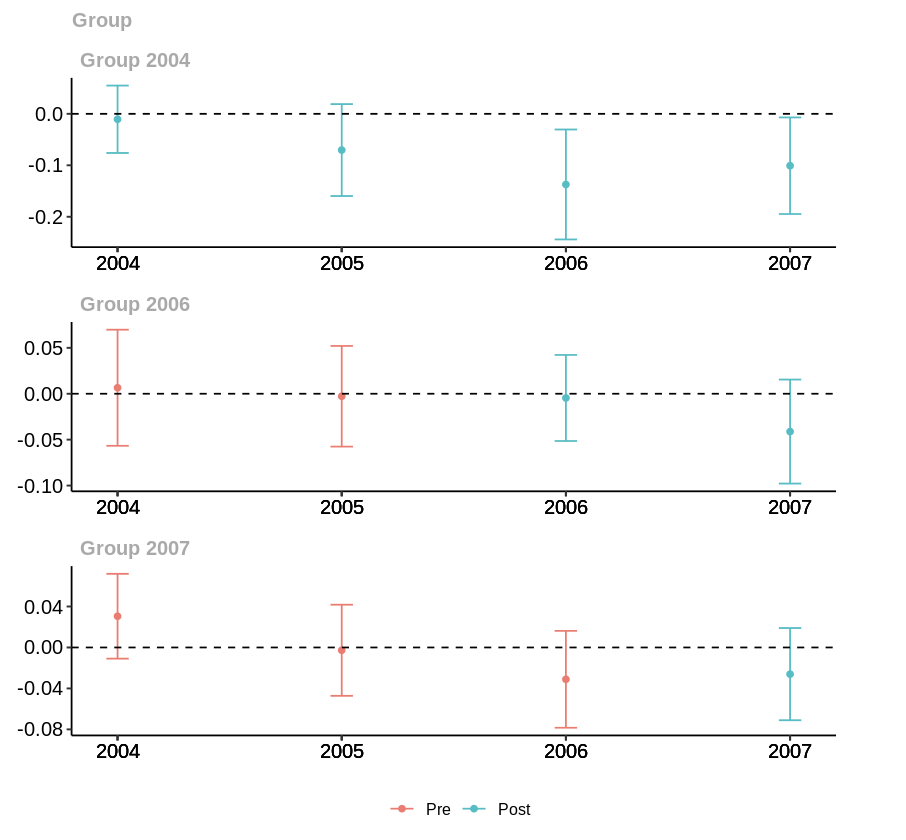
unbalancedなパネルデータを使う場合、引数の panel=FALSE とする。
データ数が多いと実行に時間がかかる模様。
どういう時にStaggered DIDが使えるか
以下の時に利用できる。
- 異なるグループが異なる時点で介入/処置を受ける
- 介入は開始してから継続する
- それぞれの介入効果に異質性がある
また、処置効果に異質性がある場合、TWFE/Event Studyだと常にバイアスが生じる。
この時にStaggered DIDが使える。
一方で、効果が同質的 or 処置が異質性と無相関であれば、TWFEでも問題ないらしい。
Rで固定効果モデルを回す際に大活躍の fixest にもStaggered DIDが実装されている。
こちらは、Sun and Abraham (2020) をもとに実装されたもの。
回帰の式の中に sunab(介入開始時間を表すカラム, 時間のカラム) を加えて上げることで、推定が実行される。
library(fixest)
data(base_did)
res_sa20 = feols(y ~ x1 + sunab(year_treated, year) | id + year, base_stagg)
summary や iplot に推定結果を渡すことで、係数が算出される。こちらは did の att_gt とは異なり、各コホート x 時間で算出された係数が時間ごとに集計されたものが表示される。
did の att_gt と同じように、各コホート(介入開始時間が同一の群)ごとに係数を確認したい場合は、agg=FALSE を指定すると良い。
# 時間ごとに係数が表示される
summary(res_sa20)
# 各コホート(介入開始時間が同一の群) x 時間 ごとに係数が表示される
summary(res_sa20, agg=FALSE)