Taxonkitを使って魚種名から対応するtaxidを取得する
はじめに
polishingがPCスペック問題でうまく行かないので箸休めな記事です。
メモレベルですが、表題そのままの内容になっています。入り乱れる分類情報をいい感じに操作してくれるtaxonkitは便利です。
Taxonkit
taxonkitは、taxonomy dumpファイルを効率的に操作可能なコマンドラインツールです。他のツールと比べて、多機能でパフォーマンスに優れています。
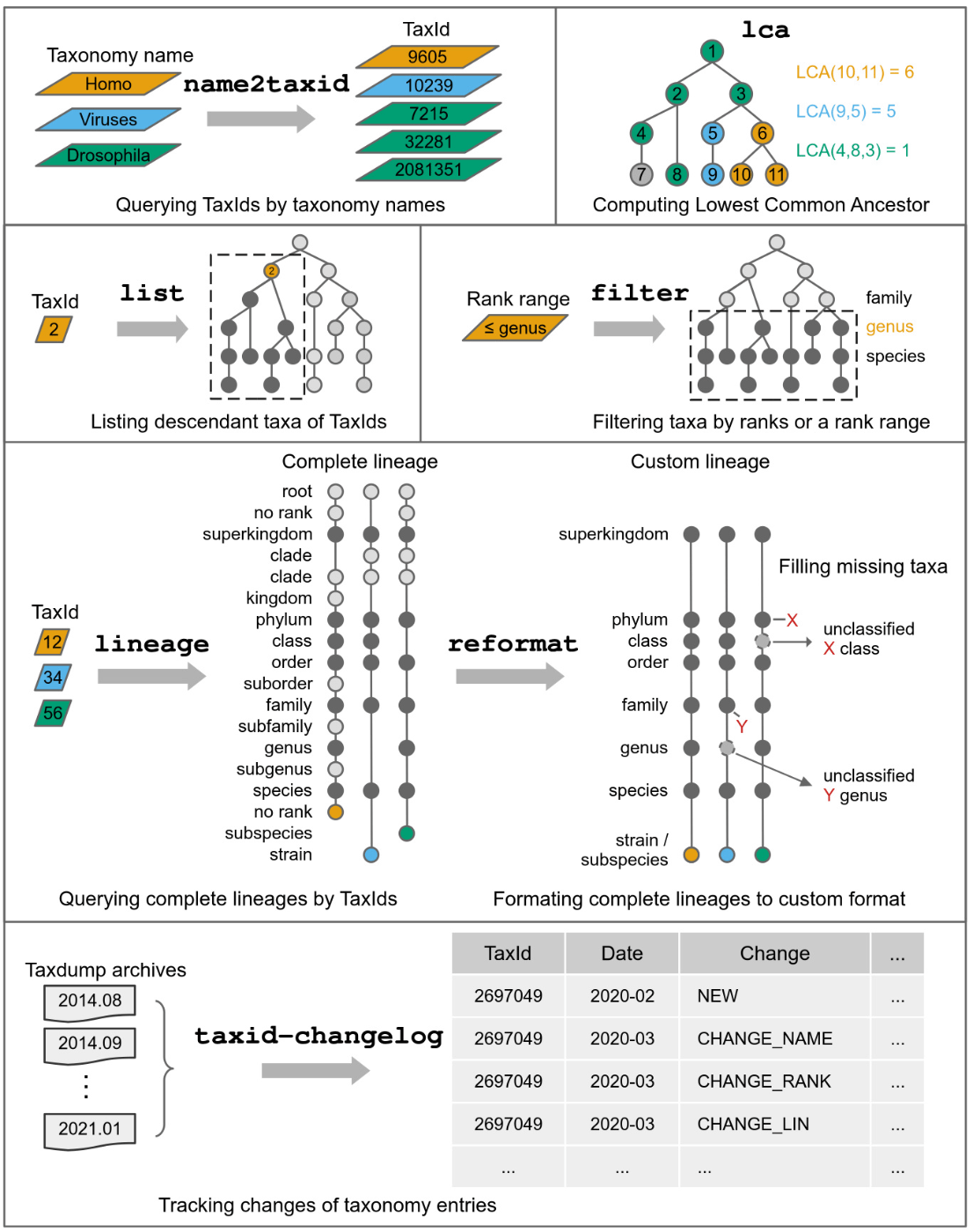
taxonokitは、NCBIのTaxonomyデータベースtaxonomy dumpファイルを使うことで、種名⇔IDの相互変換や分類階級に関する情報を得ることができます。
私は今回、以下のcsv形式のデータの種名に対応するtaxidの情報が欲しいので、種名→taxidに変換可能なname2taxidを使用する方法について記載します。
| species | region |
|---|---|
| Abbottina rivularis | 12s |
| Acanthogobius flavimanus | 12s |
| Acanthogobius hasta | 12s |
| Acanthogobius lactipes | 12s |
| Acanthopagrus latus | 12s |
| Acanthopagrus schlegelii | 12s |
| Acentrogobius sp. 2 | 12s |
| ... | ... |
私が操作しているファイルと完全一致ではないですが、以下のリポジトリにcsvファイルをおいてますので使ってください。
また一部、以下の記事を参考にしています。
Install
仮想環境を作成して、mambaでインストールしました。
# Make virtual env and install taxonkit
mamba create -n taxonkit -c bioconda taxonkit -y
# activate
mamba activate taxonkit
USAGE
TaxonKit - A Practical and Efficient NCBI Taxonomy Toolkit
Version: 0.15.0
Author: Wei Shen <shenwei356@gmail.com>
Source code: https://github.com/shenwei356/taxonkit
Documents : https://bioinf.shenwei.me/taxonkit
Citation : https://www.sciencedirect.com/science/article/pii/S1673852721000837
Dataset:
Please download and uncompress "taxdump.tar.gz":
ftp://ftp.ncbi.nih.gov/pub/taxonomy/taxdump.tar.gz
and copy "names.dmp", "nodes.dmp", "delnodes.dmp" and "merged.dmp" to data directory:
"/home/naoki/.taxonkit"
or some other directory, and later you can refer to using flag --data-dir,
or environment variable TAXONKIT_DB.
When environment variable TAXONKIT_DB is set, explicitly setting --data-dir will
overide the value of TAXONKIT_DB.
Usage:
taxonkit [command]
Available Commands:
cami-filter Remove taxa of given TaxIds and their descendants in CAMI metagenomic profile
create-taxdump Create NCBI-style taxdump files for custom taxonomy, e.g., GTDB and ICTV
filter Filter TaxIds by taxonomic rank range
genautocomplete generate shell autocompletion script (bash|zsh|fish|powershell)
lca Compute lowest common ancestor (LCA) for TaxIds
lineage Query taxonomic lineage of given TaxIds
list List taxonomic subtrees of given TaxIds
name2taxid Convert scientific names to TaxIds
profile2cami Convert metagenomic profile table to CAMI format
reformat Reformat lineage in canonical ranks
taxid-changelog Create TaxId changelog from dump archives
version print version information and check for update
Flags:
--data-dir string directory containing nodes.dmp and names.dmp (default "/home/naoki/.taxonkit")
-h, --help help for taxonkit
--line-buffered use line buffering on output, i.e., immediately writing to stdin/file for
every line of output
-o, --out-file string out file ("-" for stdout, suffix .gz for gzipped out) (default "-")
-j, --threads int number of CPUs. 4 is enough (default 4)
--verbose print verbose information
Use "taxonkit [command] --help" for more information about a command.
taxdump fileの取得
wgetで取得して解凍します。
# ダウンロード
wget -c ftp://ftp.ncbi.nih.gov/pub/taxonomy/taxdump.tar.gz
# 解凍
tar -zxvf taxdump.tar.gz
初めてであればホームディレクトリにtaxdumpファイル用のフォルダを作成
mkdir -p $HOME/.taxonkit
先程解凍したファイル群のうち必要なものをコピーしときます。
cp names.dmp nodes.dmp delnodes.dmp merged.dmp $HOME/.taxonkit
Run : name2taxid
csvファイルの1列目の2行目以降のをawkで抽出して、それをtaxonkit name2taxidに渡すことで種名に対応したtaxidが得られます。
awk -F, 'NR>1{print $1}' | taxonkit name2taxid > species_taxid.tsv
出力されたspecies_taxid.tsvを見てみましょう。
| Abbottina rivularis | 75332 |
| Acanthogobius flavimanus | 86203 |
| Acanthogobius hasta | 267130 |
| Acanthogobius lactipes | 1231916 |
| Acanthopagrus latus | 8177 |
| Acanthopagrus schlegelii | 72011 |
| Acentrogobius sp. 2 | |
| ... | ... |
Acentrogobius sp. 2 通称 ツマグロスジハゼは、taxidが出力されていない状態です。
NCBIのTaxonomy browserで本当にツマグロスジハゼに関するtaxidがないか確認してみます。
Acentrogobiusで検索してみると、一番下にそれっぽいのがおりました。
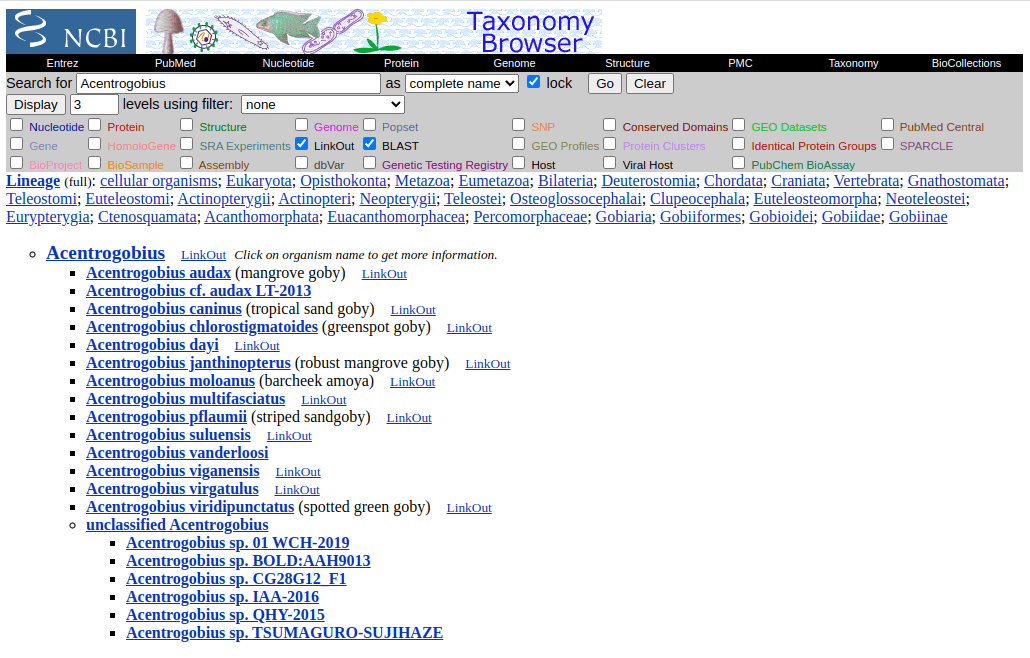
Acentrogobius sp. TSUMAGURO-SUJIHAZEでname2taxidを実行するとtaxidが得られるのか試してみます。
echo 'Acentrogobius sp. TSUMAGURO-SUJIHAZE' | taxonkit name2taxid
Acentrogobius sp. TSUMAGURO-SUJIHAZE 2057106
得られました。
taxid無いなと思ったら、taxonomy browserに登録されている種名に変更して実行してみてください。
今回はこれでおしまい。
Discussion