ONTロングリードシーケンシングのデータ解析 (1)
バイオインフォマティクス技術まとめ、Oxford nanopore technologiesの通称Nanoporeシーケンスから得られた、ロングリード配列データの解析をまとめます。
Zennで記事を書くのは初めてですので、お手柔らかによろしくお願いいたします。
サンプルデータ
RNAをONTでダイレクトにシーケンシングした、この論文のデータを解析します。
Direct detection of RNA modifications and structure using single-molecule nanopore sequencing
Stephenson W, et al. Cell Genomics, 2 100097, 2022.
データのダウンロード
Nanoporeは生データが電流(current)の波形データであることからfast5という特殊なフォーマットになっています。そのため、現状のシーケンスデータのアーカイブの多くでは残念ながら未対応になっています(参考 - hdf5規格もあるのでそこに併合される可能性?)。sratoolkitなど通常の方法でダウンロードした場合も、ベースコール後の配列ファイルしか得られません。
よって、生データが欲しい場合には、例えば以下のようにtarファイルをダウンロードしてきて、解凍する必要があります。
NCBI

プロジェクトIDがわかる場合には、Sequence Read Archiveの上部のタブ「Study」から検索をすると、関連ファイルのダウンロードリンクの一覧を表示することもできました。
フォルダ構造
ダウンロード後解凍済の中身は以下のようになります。
具体的にはworkspace以下にfast5ファイルが置かれており、ベースコーラーのログと出力結果のfastqファイルが見つかります。fastqには複数のfast5ファイルの解析結果が保存されています。
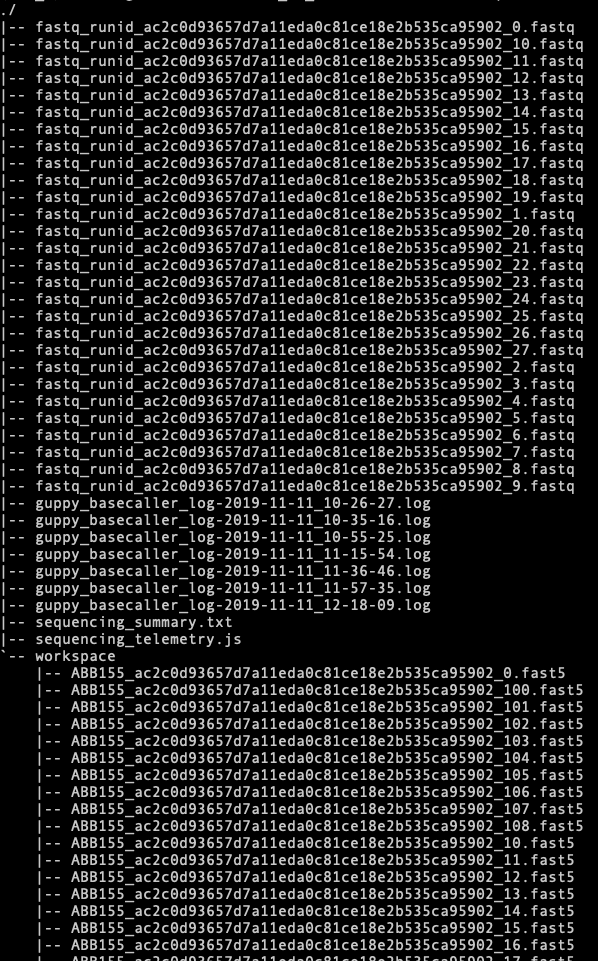
ただし、規定があるかわからないので、データセットごとに多様性があるかもしれません。
実際にこちらのRNAのダイレクトシーケンシングによる修飾検出論文では、生データはENAからのダウンロードが推奨されていました。
ベースコーラーの違い
ONTからはguppyとbonitoが提供されていますが、前者はフルアカウントを持つ人だけにバイナリで提供されるもの、後者はDeep learningかつオープンソースベースコーラーであるという違いがあります。両者ともGPUを用いたベースコールが可能です。guppyに関してはbidirectional flowを内部に持つディープラーニング、ということですがどんなアルゴリズムかは不明です。内部でいくつかバージョンも存在するようです(情報ご存知の方がいたら教えて下さい)
昔はbonitoの方が速度が遅かったようですが、大幅に速度は改善しているようです(参考)。
本論文のデータではguppyによるベースコールの結果が保管されていますが、
Guppyでは5mc,6mA, CpGに対応しているとの情報あり。
bonitoのインストールに関して
fastqのタグ説明
次回
リードのQC、basecallから修飾塩基のコーリングまでをまとめます。
Discussion