【AF2】WindowsユーザーのためのAlphafold2導入方法(デュアルブート)
こんにちは。zenzen@R-2です。
今までwindowsのOSしか使ってこなかった私がalphafold2の導入に苦戦し、結果的にデュアルブートでalphafold2を導入できたので、それをまとめた記事です。(2023年5月14日までの記録)
この記事はwindows環境を維持したままUbuntu環境を持ちたい。Alphafoldをローカル環境で使いたいという人向けです。
本家AF2ではなくLocalColabFoldという選択肢もありますので、ぜひそちらもご検討下さい。こちらは、データベースがダウンロード不要であったり、計算速度が速かったりと利点が多いです。
手軽に試したい方はGoogle Colab版のAlphafoldがおすすめです。
UbuntuなどのLinux環境はこれまで一度も触ったことがなくコマンドの使い方も1から勉強しました。ですので、この記事は出来るだけ初心者の方でも分かる内容になるように心掛けています。
PC構成
OS :windows11
マザーボード :ASUS PRIME Z690-A
CPU :i5-12600K
GPU :NVIDIA GeForce RTX 2070 SUPER
RAM :64GB
背景
バイオインフォマティクスを研究するにあたりAlphafoldを導入する必要がありました。
ですが、手持ちのパソコンはwindowsのOSのものしかありませんでした。
Google検索で"alphafold2 導入"などと検索すると日本語の記事や公式GitHubのページなどでalphafold2の導入方法が出てきますが、どれもネイティブがLinux環境での導入方法でwindowsユーザーが参考になる方法はありませんでした。
手持ちのWindows環境を維持しながらAlphafoldを使いたかったのです。
なので、こちらに私の動作事例を記載しようと思った次第です。
(注:記事の全てが参考にならないということではありません。)
はじめに
windows環境からAlphafold2を導入する時に 2つの問題 があります。(2023年5月現在)
①Windowsの仮想マシンでは動かない
Alphafold2を使うためにはLinux環境が絶対に必要なので、私はHyper-VやWSL2を使い導入を試みましたがどちらも失敗に終わりました。
Hyper-V
こちらの場合では、Hyper-Vの特性によりGPUを使っての計算が出来ませんでした。
Hyper-VでもGPUを使えるようにする方法の記事を見つけて試しましたが、少なくとも私の環境では動きませんでした。
WSL2
こちらの場合では、実行段階になりエラーが発生し、導入を断念しました。
出ていたエラーは こちらのHHblits falied でした。
もし、導入・実行できたとしても性能面ではこれから紹介するやり方がベストであると思われます。
②Dockerを使ったやり方では動かない
なぜDockerを使う方法で出来ないのか分かりませんが、私の試した限りでは使えませんでした。
Dockerを使ったやり方というのは公式GitHubの最初のページにある "Installation and running your first prediction" のことです。
解決策
上記の問題を解決するために取った方法です。
- 1つ目の課題を解決するのにはデュアルブート 。
- 2つ目の課題を解決するのにはDockerを使わない方法でのalphafold2の導入・実行。
これらについて順を追って説明していきたいと思います。
1.デュアルブート
デュアルブートとはなにか
コトバンクによると
1台のコンピューターで2つのOSを切り替えて起動できるようにすること。(中略)
たとえば、1台のパソコンに複数のOSを組み込んでおくことで、異なるOSに対応したソフトウェアや周辺機器をどちらも使える。
となっている。
今回の場合だとWindows11とUbuntu20.04でデュアルブートにする。
これによって、既存のWindowsPCの環境を変えることなくUbuntu20.04環境でAlphafold2を動かすことができる。
デュアルブートの長所と短所 (仮想マシンと比較して)
- 長所
- メモリ(RAM)がフルで使える
- ネイティブ環境になるので、ストレージへのアクセスが速い
- 環境問題がない、など…
- 短所
- 失敗するとPCがぶち壊れる
- ストレージ容量が減る(単一のストレージの場合)
- Windows環境と同時に作業出来ない、 など…
- 短所の対策(PCの故障への対策)
- 失敗するとPCが壊れるので、bootファイルに対しては復元ポイントの作成、重要なデータなどはクラウドや外付けのストレージへの保存などのバックアップ作業が考えられます。
- 新しいSSD/HDDを用意しそこにUbuntuをインストールすることで、間違ったストレージに割り当てることを回避する。(詳細は後ほど)
デュアルブートの準備
詳しくはこちらのサイトを参照してください。
準備するもの
- 8GB以上のUSB1本(Ubuntuのisoファイル用)
- 任意の外付けディスク(USB/HDD/SSD)(windowsのBootファイルバックアップ用)
- (任意)4TBのSSD/HDD 1つ(Ubuntuのブートディスク兼alphafold用)
デュアルブートの手順
1. Microsoft StoreからRufusというソフトをダウンロードします。
 Rufusを開くとこのような形
Rufusを開くとこのような形
2. Ubuntuのisoファイルをダウンロードする。
Ubuntu20.04LTSを使いたい方はこちら。Ubuntuのバージョンは22.04が最新ですが、私は何らかのパッケージが動かないなどのリスクを避けるために1つ古いものを導入しました。
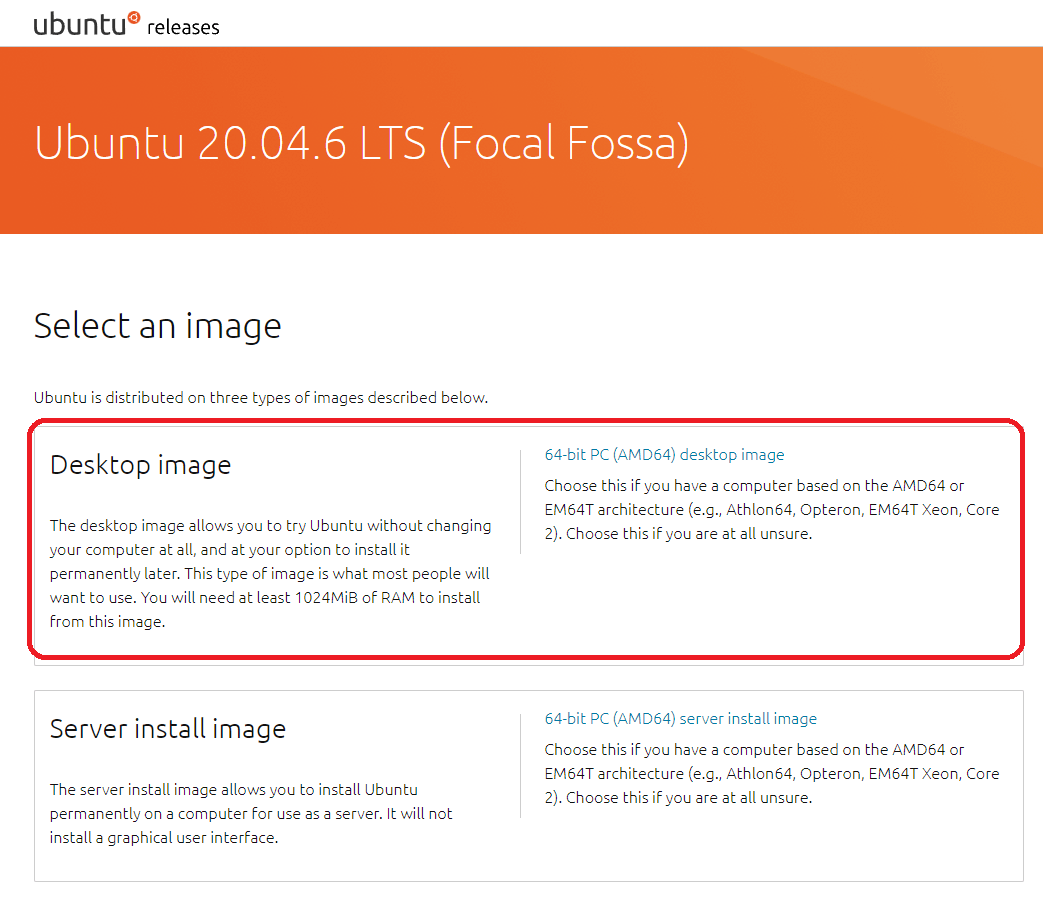 Ubuntu20.04の場合は赤枠の方
Ubuntu20.04の場合は赤枠の方
3. Rufusの選択から先ほどダウンロードしたisoファイルを選択する。
 赤丸の部分を押す
赤丸の部分を押す
4. RufusのデバイスからUSB(Ubuntuのisoファイル用)を押す。
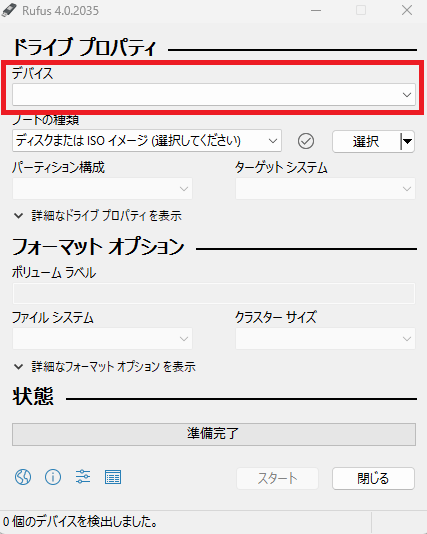 赤枠の部分
赤枠の部分
5. Rufusの準備完了ボタンを押す。
これで、Ubuntuのisoファイルが完成。
6. Bootのバックアップを取る。
Windowsの検索窓から復元ポイントの作成をクリック→作成をクリックし必要なディスクのバックアップを取る。(特に重要なものがなければbootだけで十分です。)
7. 外付けディスクにバックアップを取る。
Windowsのコントロールパネル→システムとセキュリティ→バックアップと復元(windows7)→システムイメージの作成→任意のディスクを選択→作成。(特に重要なものがなければbootだけで十分です。)
8. (任意)ディスクを準備する。
もし空のSSD/HDDがあればWindowsのディスク管理でフォーマットしておく。未割り当てにしておくと導入時にそのドライブに自動的にインストールされる。
9. 再起動をして、F2もしくはdelを押してbiosに入る。
10. Boot priorityでbootの優先順位をUbuntuの入っているUSBを上にする。
 赤枠部分をUbuntuを上にする
赤枠部分をUbuntuを上にする
11. advanced settingからsecure bootをother osにする。
 advanced setting→bootの画面。赤枠をクリックして変える画面に行きます。
advanced setting→bootの画面。赤枠をクリックして変える画面に行きます。

赤枠をother osに設定します。
12. Save&Exitでbiosを抜ける。
13. OSを選ぶ画面になるのでUbuntuを選ぶ。しばらくすると、導入画面が映る。
14. (導入の例)
Ubuntuをインストール→通常のインストール→UbuntuをWindows Boot Managerとは別にインストール→情報入力→導入完了
とても長かったと思いますが、以上でデュアルブート環境の構築は終了です。
お疲れ様です!!
もし、Windowsにしたい場合は再起動してbiosの画面に行きBoot settingをWindowsに、secure bootをWindows OSに変更して戻ればWindowsが立ち上がるようになります。
2. Alphafold2の導入
続いては、Alphafold2のインストールです。先に述べたように、公式GitHubのページで説明されているdockerを使ったやり方は出来ないので、Yoshitaka Moriwaki様が公開されているQiitaの記事を主に参考にさせていただきました。 また、こちらのalphafold_non_dockerも参考にさせていただきました。
導入の流れ
ここからはすべてUbuntu20.04のTerminalでのコマンドとなります。
Ubuntuのセットアップやコマンド解説などは今回は省略させていただきます。(そのうち記事にします。)
コマンドの簡単な説明を下部に添えてあります。
1. Alphafold2をダウンロード
特定のディスクにダウンロードしたい場合はcdコマンドを使って移動しておきましょう。
git clone https://github.com/deepmind/alphafold.git
#GitHubからファイルをダウンロードします。
cd ./alphafold
#cdコマンドでできたフォルダに移動する。
エラーなく移動できれば問題なし。
UIでもファイルからAlphafoldファイルができたことが確認できます。
2. NvidiaドライバとCUDAの確認
Nvidia製のGPUを使う場合はNvidiaドライバとCUDAを導入しなければ動作しません。
nvidia-smiと入力しエラーが出ることを確認してください。
nvidia-smi
#GPUのスタッツを確認するコマンドです。
#WindwosのタスクマネージャーのGPUだけ版です。
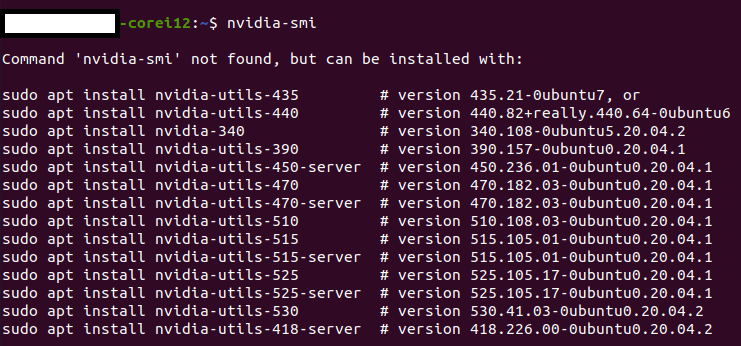 エラーだとこのようになります。
エラーだとこのようになります。
3. NvidiaドライバとCUDAのアンインストール
ここではこちらの記事を参考にしました。
Ubuntuでは既にNvidiaドライバが入っている場合はアンインストールしなければ別のものをインストールできません。なぜかは分かりません。
sudo apt purge nvidia-*
#nvidiaがつくものを削除します。
sudo apt purge cuda-*
#cudaがつくものを削除します
sudo apt autoremove --purge
#不要なパッケージを削除します。
4. NvidiaドライバとCUDAのインストール
CUDAを入れるとドライバも一緒にインストールされます。
これらは最新のもので大丈夫です。
下記のリンクから適切な物を選び導入してください。
#これは実行の例です。必ず上記リンクから適切なコマンドを取得してください。
wget https://developer.download.nvidia.com/compute/cuda/repos/ubuntu2004/x86_64/cuda-ubuntu2004.pin
sudo mv cuda-ubuntu2004.pin /etc/apt/preferences.d/cuda-repository-pin-600
wget https://developer.download.nvidia.com/compute/cuda/12.1.1/local_installers/cuda-repo-ubuntu2004-12-1-local_12.1.1-530.30.02-1_amd64.deb
sudo dpkg -i cuda-repo-ubuntu2004-12-1-local_12.1.1-530.30.02-1_amd64.deb
sudo cp /var/cuda-repo-ubuntu2004-12-1-local/cuda-*-keyring.gpg /usr/share/keyrings/
sudo apt-get update
sudo apt-get -y install cuda
5. NvidiaドライバとCUDAの動作確認
nvidia-smiとターミナルに打つと以下のような画像が表示されます。
ここで、NVIDIA-SMI、Driver Version、CUDA Versionが最新のものになっているかを確認しましょう。
特にCUDAのバージョンが11.1以上であることを確認しましょう。(CUDAのバージョンが11.1未満だと動きません。)
nvidia-smi
#GPUのスタッツを確認するコマンドです。

nvidia-smiを実行した場合
6. Chemical propertiesをダウンロード
Stero_Chemical_Propertiesという動作に必要なものをダウンロードします。
事前にcdコマンドを使ってalphafoldフォルダに移動しておきましょう。
#(例)homeディレクトリの下にAlphafoldをインストールした場合
cd alphafold
#wgetで指定したURLからファイルを指定したフォルダにダウンロードします。
wget -q -P alphafold/common/ https://git.scicore.unibas.ch/schwede/openstructure/-/raw/7102c63615b64735c4941278d92b554ec94415f8/modules/mol/alg/src/stereo_chemical_props.txt
7. Python(miniconda)を入れる
手順7,8は以下のコマンドをコピーしてbashファイルを作って実行させてもいいと思います。
まずはミニコンダをインストールします。
事前にcdコマンドを使ってAlphafoldフォルダに移動しておきましょう。やり方は手順6を参照してください。
wget -q -P . https://repo.anaconda.com/miniconda/Miniconda3-latest-Linux-x86_64.sh
#wgetで指定したURLからファイルを指定したフォルダにダウンロードします。
bash ./Miniconda3-latest-Linux-x86_64.sh -b -p $[Alphafold_Path]/conda
#bashでbashファイルを実行します。
rm Miniconda3-latest-Linux-x86_64.sh
#rmで指定したファイルを削除します。
8. 仮想環境を構築
続いて仮想環境を構築していきます。
source "[Alphafold_Path]/conda/etc/profile.d/conda.sh"
#[Alphafold_Path]にはalphafoldまでの上位フォルダ名を書きます。
#(例)
source "/home/usr/alphafold/conda/etc/profile.d/conda.sh"
#sourceでシェルが動きます。簡単に言えばWindowsのAnaconda Promptになります。
export PATH="[Alphafold_Path]/conda/condabin:${PATH}"
#export PATH= :${PATH}でPATH(環境変数)を追加する。
conda create -p [Alphafold_Path]/alphafold-conda python=3.8 -y
#conda create -pで仮想環境を作成し指定したファイルに書き込みます。
conda activate [Alphafold_Path]/alphafold-conda
#conda activateで指定した仮想環境に入ります。
conda update -n base conda -y
#conda update -nでAnaconda本体のアップデートを行います。
conda install -y -c conda-forge openmm==7.7.0 cudnn==8.2.1.32 cudatoolkit==11.1.1 pdbfixer==1.7 typing-extensions
#必要なパッケージをインストールしていきます。
#openmmのバージョンはもしかしたらエラーになる可能性があるので、出た場合は適宜変更してください。
conda install -y -c bioconda hmmer==3.3.2 hhsuite==3.3.0 kalign3
pip install pandas absl-py==1.0.0 biopython==1.79 chex==0.0.7 dm-haiku==0.0.9 dm-tree==0.1.6 immutabledict==2.0.0 jax==0.3.25 ml-collections==0.1.0 numpy==1.21.6 scipy==1.7.0 tensorflow-cpu==2.9.0 protobuf==3.20.1 pandas==1.3.4
pip install https://storage.googleapis.com/jax-releases/cuda11/jaxlib-0.3.25+cuda11.cudnn82-cp38-cp38-manylinux2014_x86_64.whl
#jaxをインストールします。このコード動かなければ、以下のコードを試して見てください。
#pip install --upgrade pip
#pip install --upgrade "jax[cuda12_pip]" -f https://storage.googleapis.com/jax-releases/jax_cuda_releases.html
pip install --upgrade jax==0.3.25
#jaxを指定のバージョンにします。
9. openmmを入れる
これは他のサイトにありませんが、私はこれをやらないと動きませんでした。
こちらのサイトを参照しながら導入します。
最初に導入すると何かしらのパッケージのバージョンが足りないと言われます。なので、そのパッケージもsudo apt install [パッケージ]でアップデートします。
(パッケージ名は忘れてしまいました。すみません。)
conda install -c conda-forge openmm-setup
#openmm-setupをインストールします。
10.データベースのダウンロード
予測に使うデータベースをダウンロードしていきます。基本的にダウンロードには24時間以上の時間がかかります。(特定のデータベースがボトルネックになっていると思います。)
気長に待ちましょう。(高速化の方法を知っている方はぜひご教授下さい。)
データベースのダウンロードとpython等のセットアップは前後しても問題ありません。
データベースについてはYoshitaka Moriwaki様が公開されているQiitaの記事がとても参考になります。
手順としては、ダウンロードコマンドをダウンロードし、Alphafoldファイルに移動した後、データベースのダウンロードをします。
sudo apt -y install rsync aria2
#rsyncとaria2をインストールします。
cd alphafold
#cdコマンドを使ってalphafoldフォルダに移動。上記は例です。
scripts/download_all_data.sh [Download_Dir]
#download_all_data.shですべてのデータベースをダウンロードしていきます。
#[Download_Dir]にデータベースの任意のダウンロード先を入力してください。
#alphafoldと同じディスクにあると早いそうです。でも、alphafoldの下にデータベースを作成すると遅くなるそうです。
#(例)ホームディレクトリの下のDATAフォルダを指定した場合
scripts/download_all_data.sh home/[user_name]/DATA
#[user_name]はあなたのユーザーネームです。
11. (任意)複合体を予測したい方は追加のデータベースのダウンロード
複合体を予測するには以下の2つの追加データベースが必要です。
-download_uniport.sh
-download_seqres
度々ですが、詳しくはYoshitaka Moriwaki様が公開されているQiitaの記事の該当部分がとても参考になると思います。
download_uniport.shは10と変わりません。
download_seqresはダウンロードが遅いので、速度の速い日本のサーバーに変更します。
以下は、download_seqres.shの34行目について、以下の2行目に書き換えてください。
- SOURCE_URL="ftp://ftp.wwpdb.org/pub/pdb/derived_data/pdb_seqres.txt"
+ SOURCE_URL="https://ftp.pdbj.org/pub/pdb/derived_data/pdb_seqres.txt.gz"
変更したらダウンロードをし、解凍します。
scripts/download_uniport.sh [Download_Dir]
#uniportをダウンロードします。(これは解凍などは不要です。)
scripts/download_pdb_seqres.sh [Download_Dir]
#pdb_seqresをダウンロードします。(解凍が必要です。)
cd [Download_Dir]/pdb_seqres
#pdb_seqresに移動します。
gunzip pdb_seqres.txt.gz
#gunzipを使って対象のファイルを解凍します。
12. Alphafoldの実行ファイルの作成
ここでは2通りのやり方がありますが、Yoshitaka Moriwaki様が公開されているQiitaの記事を参考にさせていただきます。(もう一つは後に書きます。)
Qiitaの記事にもあるようにalphafoldフォルダの中でbinフォルダを作成し、以下のbashプログラムをbinフォルダの中におきます。その後、作成したbashプログラムに実行権限を付けます。
上記をダウンロードもしくはコピーしてalphafold/binフォルダの下におきます。Ubuntuであれば、ファイルを開いてwindowsのようにファイル移動可能です。
cd [Alphafold_Path]/bin
#cdでアルファフォールドフォルダのbinフォルダに移動します。
chmod +x alphafold.sh
#chmod +x で実行権限を付与します。Windowsでは気にしないところ。
Alphafold2を実行する
あと少しです。
Alphafold2を実行してみましょう。
事前にfastaファイルを用意して適当な場所に置いておきましょう。
まずは、先ほどのbinフォルダに移動します。
cd [Alphafold_Path]/bin
#cdで作成したbinフォルダに移動します。
Alphafold.shを実行します。
bash alphafold.sh \
--fasta_paths=[Fasta_path]/[fastafile].fasta \
--max_template_date=2024-01-01 \
--output_dir=[Output_Path]
#fasta_pathはfastaファイルまでのパス
#max_template_dateはデータベースのデータをいつまでの分を含むのか
#出力するフォルダのパス
実行すると1時間~8時間程度で処理が終わります。(設定によって変わります。)
以上です!!お疲れ様です!!
まとめ
Windows環境を維持したまま、デュアルブートによってLinux環境を構築し、Alphafold2を実行することができた。
これによって、PCを新しく買わなくて済みました。
参考にさせて頂いたもの
- Alphafold公式GitHub
- Yoshitaka Moriwaki様が公開されているQiitaの記事
- alphafold nondocker setup
おまけ
Ubuntu上で手早く予測されたたんぱく質を見る方法などを書きます。
Ubuntuで素早くたんぱく質を確認
Ubuntuでも様々なたんぱく質構造分析ソフトがありますが、手っ取り早く見たいだけであればアプリストアでワンクリックでダウンロードできるPyMolがおすすめです。
(他にもおすすめのアプリがありましたらご教授ください。)
Discussion