【Python】scikit-learnを用いたパーセプトロンモデルの訓練
はじめに
この記事は株式会社インプレスの「Python機械学習プログラミング Pytorch&scilit-learn編」を読んで、私が学習したことをまとめています。
今回は3章2節の「scikit-learn活用へのファーストステップ:パーセプトロンの訓練」を読んで学んだことをまとめていきます。
【リンク紹介】
・【一覧】Python機械学習プログラミング Pytorch&scilit-learn編
・これまで書いたシリーズ記事一覧
第2章では分類のための学習アルゴリズムとしてパーセプトロンとADALINEを学びましたが、今回はscikit-learnライブラリ利活用の導入として、パーセプトロンの実装を行っていきます。用いるデータはIrisデータセットです。
インポート
# 場合によってインストール
pip install colorama > /dev/null
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
from sklearn import datasets
from sklearn.metrics import accuracy_score # 正解率
from sklearn.linear_model import Perceptron # パーセプトロンモデル
from sklearn.preprocessing import StandardScaler
from sklearn.model_selection import train_test_split # データセットの分割
from matplotlib.colors import ListedColormap
Irisデータセットの準備
# Irisデータセットをロード
iris = datasets.load_iris()
# データセットの確認
print(iris)
# 特徴量の取得
# 150個のデータの2, 3行目(花びらの長さと花びらの幅)を抽出
X = iris.data[:, [2, 3]]
# 目的変数の取得
y = iris.target
# 目的変数のunique値(以後正解ラベルという)を確認する
print('Class labels:', np.unique(y))
訓練データセットとテストデータセットに分割する
# データセットの分割
X_train, X_test, y_train, y_test = train_test_split(X, y,
test_size = 0.3, # 全体の30%をテストデータとする
random_state = 1, # 乱数を固定
stratify = y) # 層化抽出を実行
上の分割作業の中で引数statify = yと指定しましたが、これは層化抽出を行う指示となります。これで正解ラベルのバランスを維持してデータセットを分割することができ、今回はIris-setosa
print('Labels counts in y:', np.bincount(y))
print('Labels counts in y_train:', np.bincount(y_train))
print('Labels counts in y_test:', np.bincount(y_test))
Labels counts in y: [50 50 50]
Labels counts in y_train: [35 35 35]
Labels counts in y_test: [15 15 15]
訓練データを標準化する
前回と同様に、モデル学習の前に訓練データを標準化させていきますが、今回はscikit-learnのpreprocessingモジュールのStandardScalerクラスを使います。
# インスタンス化
sc = StandardScaler()
# 学習し、特徴量毎の平均値と標準偏差を推定する
sc.fit(X_train)
# 推定した平均値と標準偏差を用いて標準化を行う
X_train_std = sc.transform(X_train)
X_test_std = sc.transform(X_test)
パーセプトロンモデルによる学習を行う
それでは早速パーセプトロンを用いて学習を行っていきます。
# 学習率0.1でパーセプトロンのインスタンスを生成
ppn = Perceptron(eta0 = 0.1, random_state = 1)
# 学習を実施
ppn.fit(X_train_std, y_train)
次にpredictメソッドを用いて予測を行います。
# テストデータで予測を実施
y_pred = ppn.predict(X_test_std)
# 誤分類したデータ点の個数を表示
print('Misclassified examples: %d' % (y_test != y_pred).sum())
予測値と正解値を用いて正解率を算出してみましょう。
# 分類の正解率を表示
print('Accuracy: %.3f' % accuracy_score(y_test, y_pred))
# パーセプトロンに定義されているscoreメソッドを用いて正解率を求めることも可能
# sklearn.linear_model.Perceptronライブラリ:https://scikit-learn.org/stable/modules/generated/sklearn.linear_model.Perceptron.html
print('Accuracy: %.3f' % ppn.score(X_test_std, y_test))
実行結果
Accuracy: 0.978
Accuracy: 0.978
決定境界を可視化するための関数の実装
最後にplot_decision_regions関数を用いてパーセプトロンの決定領域を描画していきます。テストデータセットのデータ点を強調するために少し工夫を行います。
def plot_decision_regions(X, y, classifier, test_idx = None, resolution = 0.02):
"""マーカーとカラーマップの準備"""
markers = ('o', 's', '^', 'v', '<')
colors = ('#3F9877', # ジェードグリーン
'#003F8E', # インクブルー
'#EA5506', # 赤橙
'gray',
'cyan'
)
cmap = ListedColormap(colors[: len(np.unique(y))])
"""グリッドポイント(格子点)の生成"""
# x軸方向の最小値、最大値を定義
x_min = X[:, 0].min() - 1
x_max = X[:, 0].max() + 1
# y軸方向の最小値、最大値を定義
y_min = X[:, 1].min() - 1
y_max = X[:, 1].max() + 1
# 格子点の生成
xx, yy = np.meshgrid(np.arange(x_min, x_max, resolution),
np.arange(y_min, y_max, resolution)
)
# 確認用
#print(xx1)
#print(xx2)
"""各特徴量を1次元配列に変換(ravel())して予測を実行"""
# つまり2つの特徴量から0と予測された格子点と1と予測された値が格子点ごとにlabに格納される
lab = classifier.predict(np.array([xx.ravel(), yy.ravel()]).T)
# 確認用
#print(np.array([xx1.ravel(), xx2.ravel()]).T)
#print(lab) # 格子点の並びで0,1が格納されているが、この時点では1次元配列なので変換が必要
"""予測結果の元のグリッドポイント(格子点)のデータサイズに変換"""
lab = lab.reshape(xx.shape)
"""グリッドポイントの等高線のプロット"""
plt.contourf(xx, yy, lab,
alpha = 0.3, # 透過度を指定
cmap = cmap
)
plt.xlim(xx.min(), xx.max())
plt.ylim(yy.min(), yy.max())
"""決定領域のプロット"""
# クラスごとに訓練データをセット
for idx, cl in enumerate(np.unique(y)):
plt.scatter(x = X[y == cl, 0],
y = X[y == cl, 1],
alpha = 0.8,
c = colors[idx],
marker = markers[idx],
label = f'Class {cl}',
edgecolor = 'black'
)
"""テストデータ点を目立たせる(点を〇で表示)"""
if test_idx: # ここはbool(test_idx)と同義。つまりTrueを返す
# すべてのデータ点を描画
X_test = X[test_idx, :]
y_test = y[test_idx]
plt.scatter(X_test[:, 0],
X_test[:, 1],
c = 'none',
edgecolor = 'black',
alpha = 1.0,
linewidth = 1,
marker = 'o',
s = 100,
label = 'Test set'
)
if test_idx について
参考資料:bool関数の使い方(オブジェクトが真か偽か判定する)
決定境界を可視化
# 訓練データとテストデータの特徴量を行方向に結合
X_combined_std = np.vstack((X_train_std, X_test_std))
# 訓練データとテストデータのクラスラベルを結合
y_combined = np.hstack((y_train, y_test))
# 決定領域をプロット
plot_decision_regions(X = X_combined_std,
y = y_combined,
classifier = ppn,
test_idx = range(105, 150)
)
plt.xlabel('Petal length [standardized]')
plt.ylabel('Petal width [standardized]')
plt.legend(loc = 'upper left')
plt.tight_layout()
# グラフを保存
plt.savefig('fig3-1.png')
plt.show()
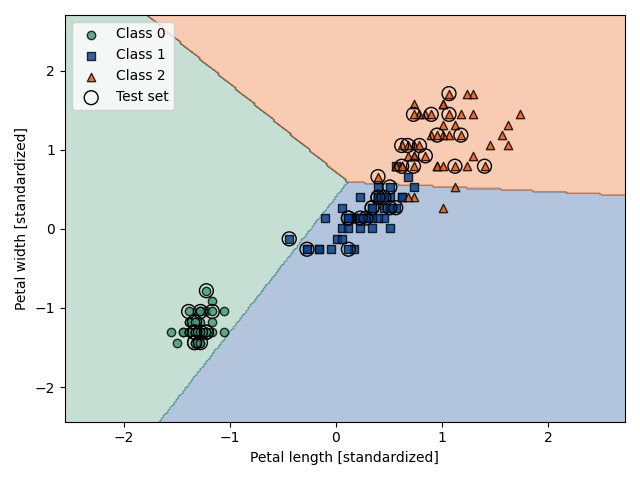
決定境界を可視化しましたが、やはり線形分離ができないデータセットにおいて、パーセプトロンアルゴリズムは収束できません。この問題点を改善する分類器として、ロジスティック回帰を考えていきいます。
参考文献
- Sebastian Raschka, Yuxi(Hayden)Liu, Vahid Mirjalili.Python機械学習プログラミング PyTorch&scilit-learn編.株式会社インプレス,2022
- Christopher M. Bishop.パターン認識と機械学習 上.丸善出版,2014
- Soledad Galli.データサイエンティストのための特徴量エンジニアリング.株式会社マイナビ,2023
- 寺田学, 辻真吾, 鈴木たかのり, 福島真太郎.Pythonによる新しいデータ分析の教科書 第2版.翔泳社,2023
- 三宅敏恒.線形代数学-初歩からジョルダン標準形へ-.培風館,2009
- 稲垣宣生,山根芳知,吉田光雄.統計学入門.裳華房, 2009
- 宮腰忠.高校数学+α 基礎と論理の物語.共立出版, 2013
ご協力のほどよろしくお願いします。

Discussion