💓
Neurokit アルゴリズム — 心電図 QRS/Rピーク検出
Neurokit アルゴリズム — 心電図 QRS / Rピーク検出
概要
NeuroKit2は、生理信号処理のためのPythonライブラリであり、心電図(ECG)信号からRピークを検出するためのデフォルトアルゴリズムを提供している。他のアルゴリズムより、所見では精度が良いので、中身を理解するために、処理の流れを整理する。
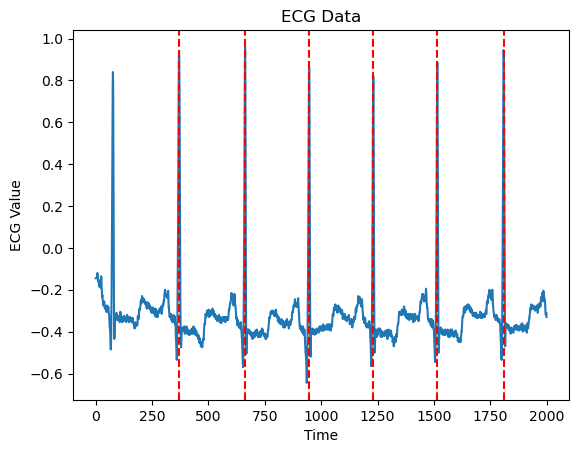
ライブラリの結果
import neurokit2 as nk
import pandas as pd
import wfdb
import matplotlib.pyplot as plt
record = wfdb.rdrecord('100', pn_dir='mitdb')
ecg_signal = record.p_signal[:,0]
ecg_df = pd.DataFrame(ecg_signal, columns=["ecg"])
signal,info = nk.ecg_peaks(ecg_df["ecg"].values, sampling_rate=record.fs, method="neurokit")
# データの可視化
plt.plot(ecg_df["ecg"].values[:2000]) # 最初の3000サンプルをプロット
#ピークの位置に赤帽線
for peak in info["ECG_R_Peaks"]:
if peak < 2000: # プロット範囲内のピークのみ表示
plt.axvline(x=peak, color='r', linestyle='--')
plt.title('ECG Data')
plt.xlabel('Time')
plt.ylabel('ECG Value')
plt.show()
アルゴリズムの流れ
NeuroKit2のRピーク検出アルゴリズムは、以下のステップで構成されている。
- 勾配計算: ECG信号の勾配を計算し、信号の変化率を強調する。これにより、急激な変化(QRS複合体)がより明確になる。
- 絶対値計算: 勾配の絶対値を計算し、信号の変化の大きさを強調する。これにより、正負の変化が区別されなくなる。
- スムージング(短時間): 短時間のスムージングを適用し、ノイズを低減する。boxcar カーネルを使用して、信号の短期的な変動を平滑化する。
- 平均化(長時間窓): 信号の振幅を一定の範囲にスケーリングすることで、ピーク検出の精度を向上させる。
- 閾値計算
- QRSの開始地点と終了地点の特定(たちあがり、たちさがり検出)
- 不良区間の除外(短すぎるQRSのスキップ)し,各QRS内でピーク検出
1. 勾配計算
ECG信号の勾配を計算し、信号の変化率を強調する。これにより、急激な変化(QRS複合体)がより明確になる。
import numpy as np
import matplotlib.pyplot as plt
import wfdb
from neurokit_ecg_findpeaks import signal_smooth, neurokit_findpeaks
# レコードと信号の読み込み(既に前のセルで読み込んでいれば省略可)
rec = wfdb.rdrecord('100', pn_dir='mitdb')
sig = rec.p_signal[:,0]
fs = rec.fs
plt.figure(figsize=(12, 8))
plt.plot(sig[:2000])
plt.legend(['Original Signal'])
plt.title('Original ECG Signal')
plt.xlabel('Samples')
plt.ylabel('Amplitude')
plt.grid()
plt.show()
#1.勾配計算(試しに2000サンプルで計算)
grad = np.gradient(sig[:2000])
plt.figure(figsize=(12, 8))
plt.plot(grad)
plt.legend(['Original Signal'])
plt.title('Original ECG Signal')
plt.xlabel('Samples')
plt.ylabel('Amplitude')
plt.grid()
plt.show()
| 心電図データ | 勾配計算 |
|---|---|
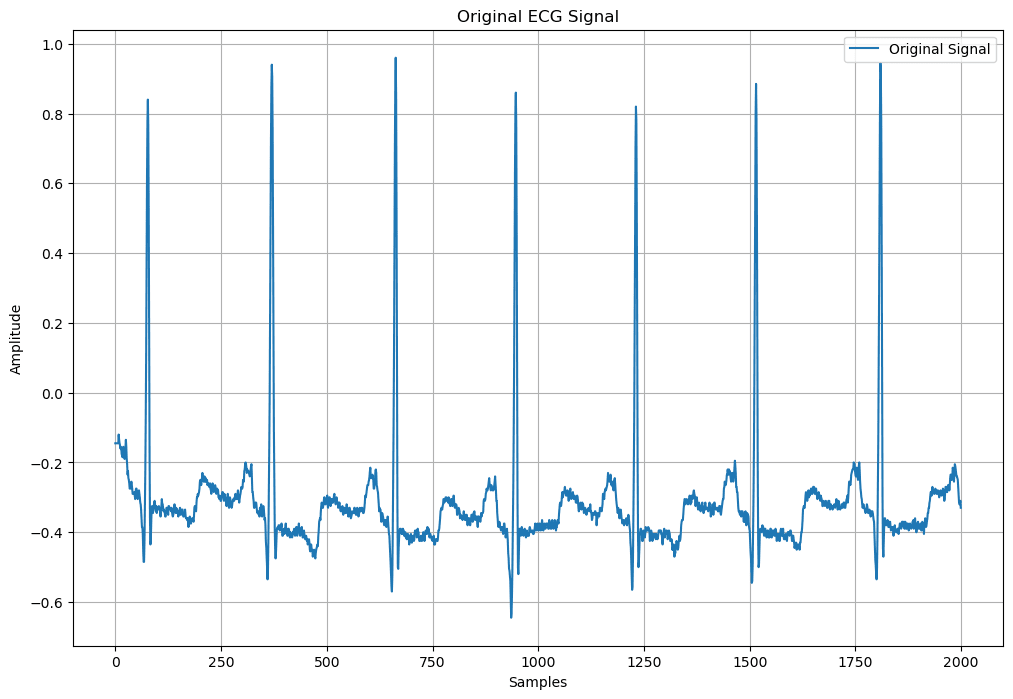 |
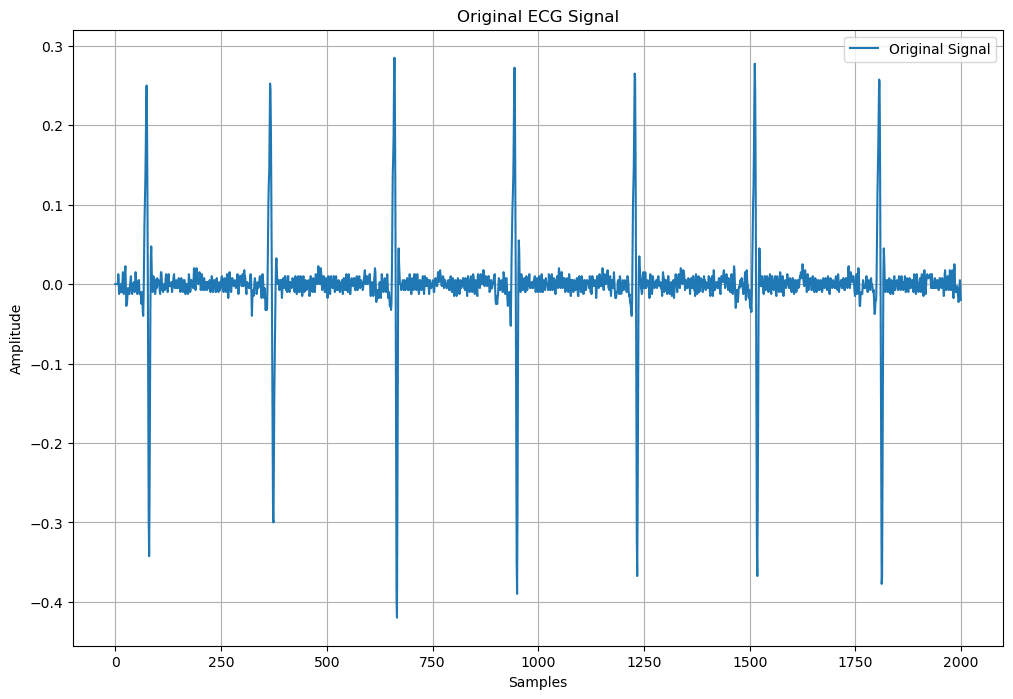 |
2. 絶対値計算
勾配の絶対値を計算し、信号の変化の大きさを強調する。これにより、正負の変化が区別されなくなる。
#絶対値に変換
absgrad = np.abs(grad)
plt.figure(figsize=(12, 8))
plt.plot(absgrad)
plt.legend(['Absolute Gradient'])
plt.title('Absolute Gradient of ECG Signal')
plt.xlabel('Samples')
plt.ylabel('Amplitude')
plt.grid()
plt.show()
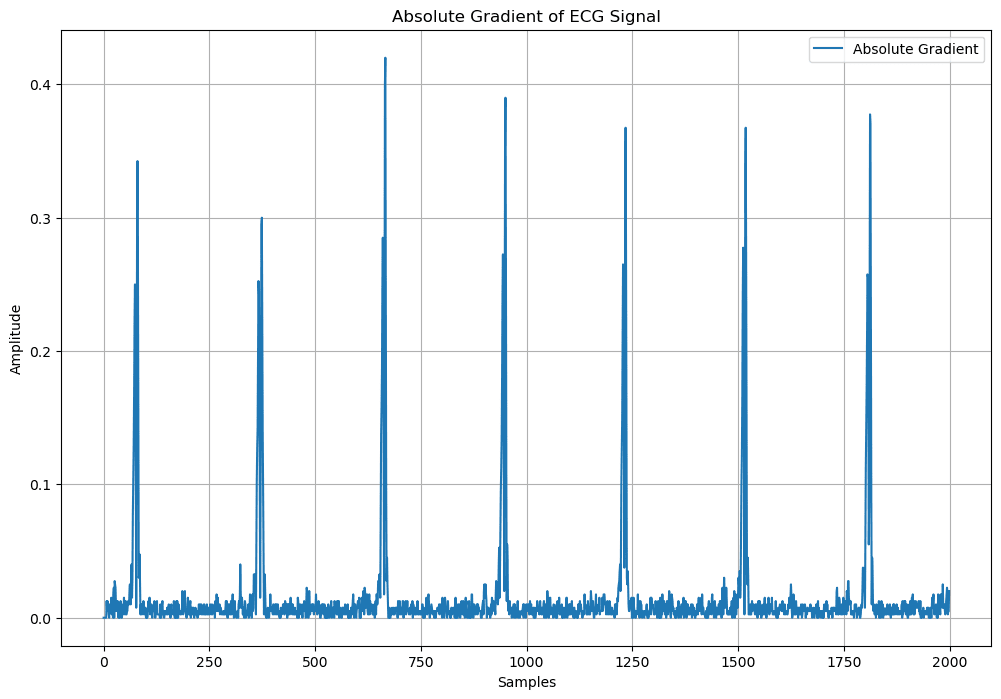
3. スムージング(短時間)
短時間のスムージングを適用し、ノイズを低減する。boxcar カーネルを使用して、信号の短期的な変動を平滑化する。
# 短時間スムージング
def signal_smooth(x, kernel='boxcar', size=3):
if kernel != 'boxcar':
raise ValueError('Only boxcar kernel supported in this minimal port')
if size <= 1:
return x
window = np.ones(size) / size
return np.convolve(x, window, mode='same')
smoothwindow = 0.1
smooth_kernel = int(np.rint(smoothwindow * fs))#fs上で使用した値
#平滑化
smoothgrad = signal_smooth(absgrad, kernel='boxcar', size=max(1, smooth_kernel))
#boxcar 平滑化の結果をプロット
plt.figure(figsize=(12, 8))
plt.plot(smoothgrad)
plt.legend(['Smoothed Gradient'])
plt.title('Smoothed Absolute Gradient of ECG Signal')
plt.xlabel('Samples')
plt.ylabel('Amplitude')
plt.grid()
plt.show()
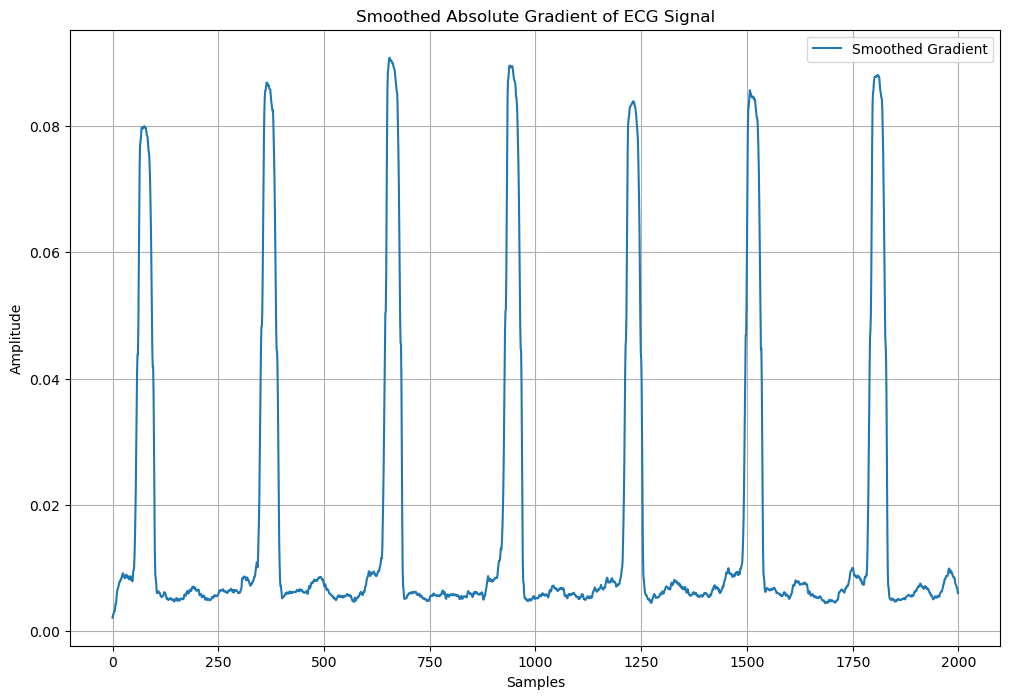
4. 平均化(長時間窓)
長時間の移動平均を適用し、信号の全体的な傾向を捉える。これにより、ピーク検出の基準となる信号が得られる。
def signal_smooth(x, kernel='boxcar', size=3):
if kernel != 'boxcar':
raise ValueError('Only boxcar kernel supported in this minimal port')
if size <= 1:
return x
window = np.ones(size) / size
return np.convolve(x, window, mode='same')
avgwindow=0.75
avg_kernel = int(np.rint(avgwindow * fs))
#平均化
avggrad = signal_smooth(smoothgrad, kernel='boxcar', size=max(1, avg_kernel))
plt.figure(figsize=(12, 8))
plt.plot(avggrad)
plt.legend(['Averaged Gradient'])
plt.title('Averaged Smoothed Absolute Gradient of ECG Signal')
plt.xlabel('Samples')
plt.ylabel('Amplitude')
plt.grid()
plt.show()
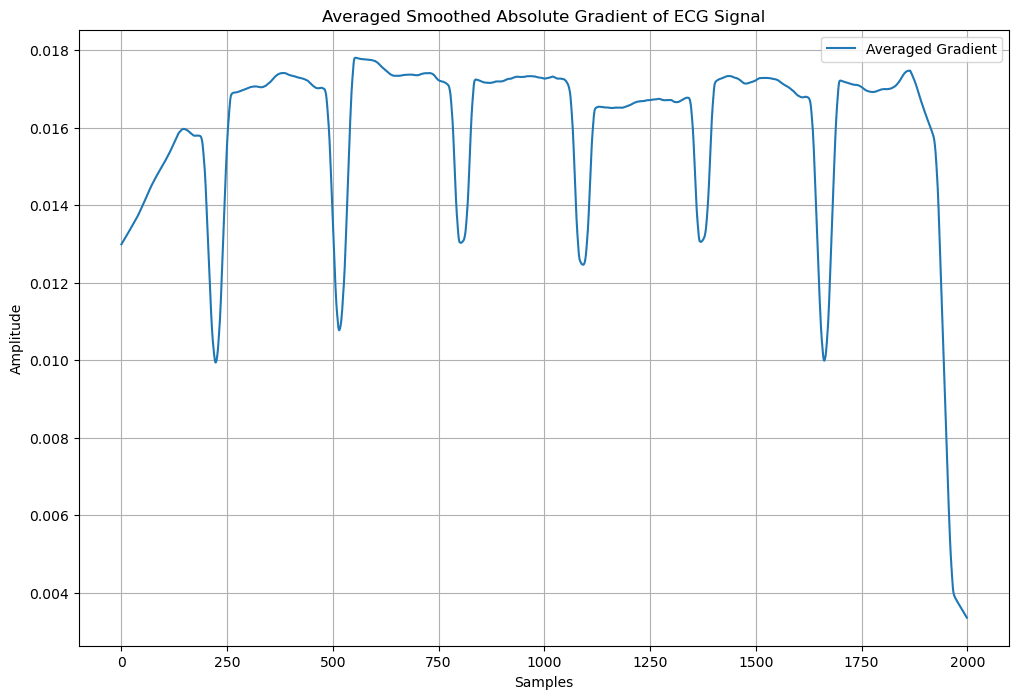
5. 閾値計算
QRSの開始地点と終了地点決定するための閾値を計算する。
平均化された信号の一定の割合を閾値として設定する。
gradthreshweight = 1.5
gradthreshold = gradthreshweight * avggrad
plt.figure(figsize=(12, 8))
plt.plot(avggrad, label='Averaged Gradient')
plt.plot(gradthreshold, label='Gradient Threshold', linestyle='--')
plt.legend()
plt.title('Averaged Gradient and Gradient Threshold')
plt.xlabel('Samples')
plt.ylabel('Amplitude')
plt.grid()
plt.show()
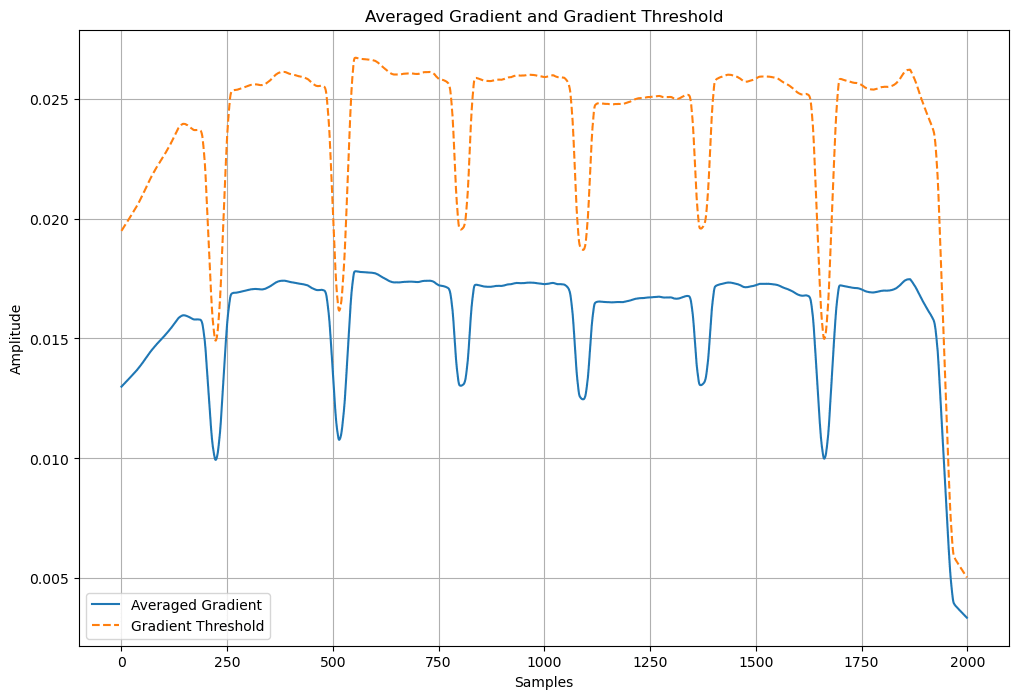
6. QRSの開始地点と終了地点の特定(たちあがり、たちさがり検出)
作成した閾値を使用して、QRS複合体の開始地点と終了地点を特定する。
これにより、各QRS複合体の範囲が明確になる
# QRSの開始地点と終了地点の特定できる閾値になる
plt.figure(figsize=(12, 8))
plt.plot(smoothgrad, label='Smoothed Gradient')
plt.plot(gradthreshold, label='Gradient Threshold', linestyle='--')
plt.legend()
plt.title('Smoothed Gradient and Gradient Threshold')
plt.xlabel('Samples')
plt.ylabel('Amplitude')
plt.grid()
plt.show()
#6.立ち上がり、立ち下がり検出
qrs = smoothgrad > gradthreshold #true/falseの配列
# beg_qrs: 最初の1つの立ち上がりのインデックス
# end_qrs: 最初の1つの立ち下がりのインデックス
beg_qrs = np.where(np.logical_and(np.logical_not(qrs[0:-1]), qrs[1:]))[0]
end_qrs = np.where(np.logical_and(qrs[0:-1], np.logical_not(qrs[1:])))[0]
end_qrs = end_qrs[end_qrs > beg_qrs[0]]
#beg_qrs end_qrs 可視化
plt.figure(figsize=(12, 8))
plt.plot(sig[:2000], label='ECG Signal')
for beg in beg_qrs:
if beg < 2000:
plt.axvline(x=beg, color='g', linestyle='--', label='QRS Onset' if 'QRS Onset' not in plt.gca().get_legend_handles_labels()[1] else "")
for end in end_qrs:
if end < 2000:
plt.axvline(x=end, color='m', linestyle='--', label='QRS Offset' if 'QRS Offset' not in plt.gca().get_legend_handles_labels()[1] else "")
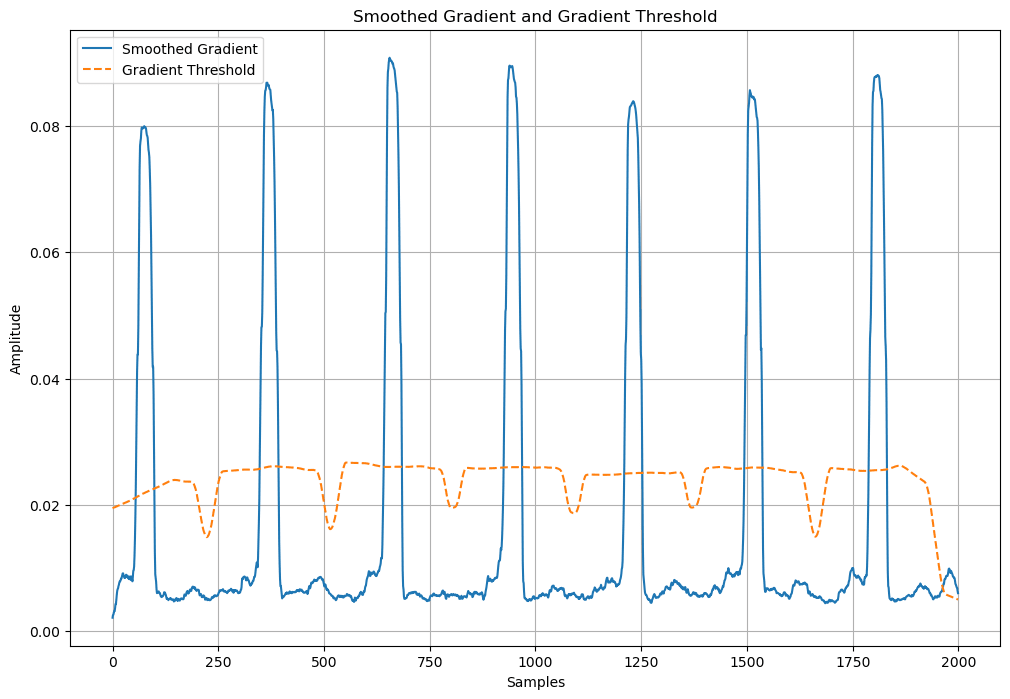
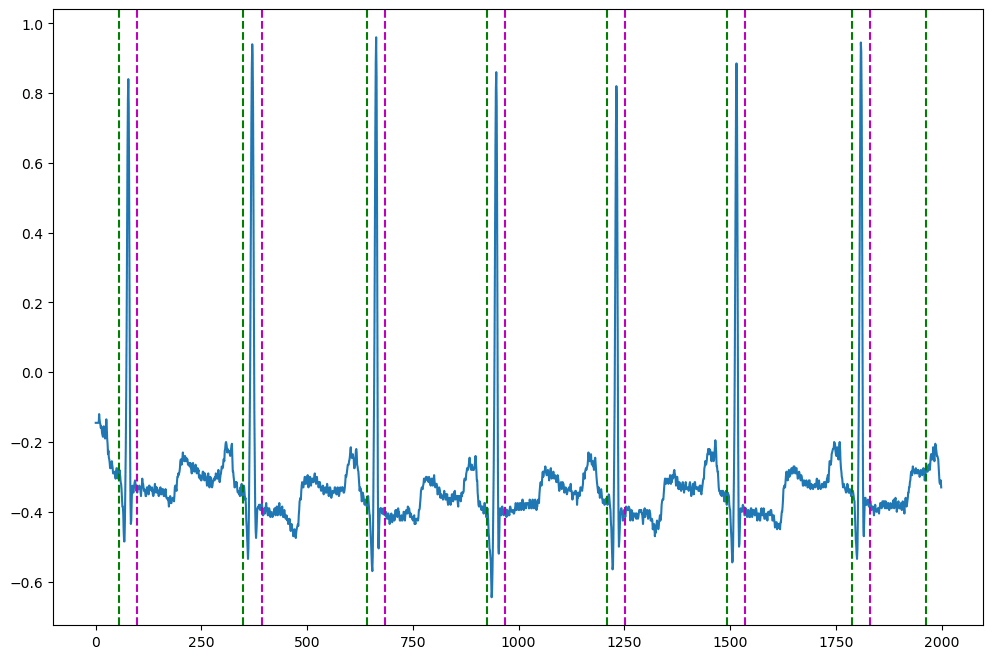
7. 不良区間の除外(短すぎるQRSのスキップ)し,各QRS内でピーク検出
最後に、各QRS複合体内でRピークを検出する。短すぎるQRS複合体は除外し、各QRS内で最も顕著なピークを特定する。
#分割した範囲ないでscipy.signal.find_peaksを使ってRピークを検出
import scipy.signal
minlenweight = 0.4
mindelay=0.3
# Identify R-peaks within QRS (ignore QRS that are too short).
num_qrs = min(beg_qrs.size, end_qrs.size)
mindelay = int(np.rint(fs * mindelay))
min_len = np.mean(end_qrs[:num_qrs] - beg_qrs[:num_qrs]) * minlenweight
peaks = [0]
for i in range(num_qrs):
beg = beg_qrs[i]
end = end_qrs[i]
len_qrs = end - beg
if len_qrs < min_len:
continue
# Find local maxima and their prominence within QRS.
data = sig[beg:end]
locmax, props = scipy.signal.find_peaks(data, prominence=(None, None))
if locmax.size > 0:
# Identify most prominent local maximum.
peak = beg + locmax[np.argmax(props["prominences"])]
#最も顕著な局所最大値を特定します。
if peak - peaks[-1] > mindelay:
peaks.append(peak)
peaks.pop(0)
# Rピーク検出結果を可視化
plt.figure(figsize=(12, 8))
plt.plot(sig[:2000], label='ECG Signal')
for peak in peaks:
if peak < 2000:
plt.axvline(x=peak, color='r', linestyle='--', label='R Peak' if 'R Peak' not in plt.gca().get_legend_handles_labels()[1] else "")
plt.legend()
plt.title('ECG Signal with Detected R Peaks')
plt.xlabel('Samples')
plt.ylabel('Amplitude')
plt.grid()
plt.show()
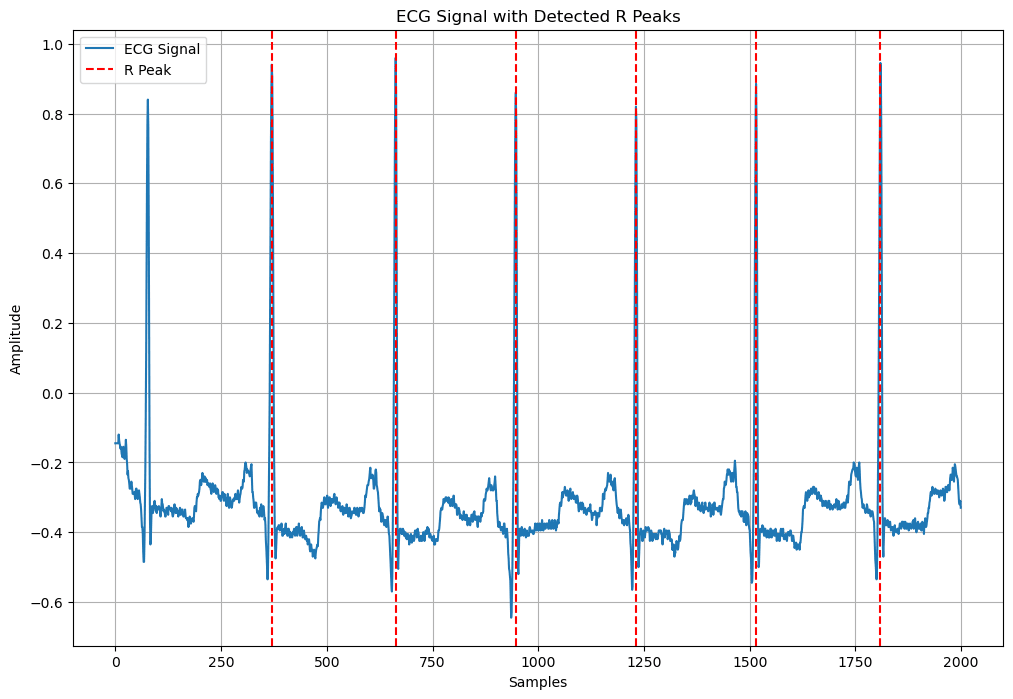
まとめ
ルールベースの検出になる。
また、結局はscipyのfind_peaksを使っているため、上に山なR波では検出できるが、マイナス側にも谷が大きくあるR波では、R波をご検出することがある。
Discussion