Low Performance PDF Parserのことはじめ。LLMとUnstructuredでPDFをパースする。
TL;DR
- 本文抽出は、PDFパーサーでパースしたやつをLLMにさらに綺麗にしてもらう。
- 画像とテーブルは、Unstructuredを使って、OCRベースでを抜き出す
- PDFをRAGが使いやすいようにできるけど遅い!だから使い所は予めドキュメントを準備するケース
- GPT3.5だと本文抽出微妙な部分もあるので、Haikuの方がいいかも(GUIからは圧倒的にHaikuでの抽出はうまくできてた)
はじめに
PDFをRAGで利用する際、単にPDFパーサーにかけてパースするだけでは、本文以外のフッターやヘッダー、図のキャプションなどさまざまなものが混入してしまうことがあります。これによりRAGの精度が落ちたり、ベクトル検索の結果が見づらくなったりすることがよくあります。
このような問題に対処するために、本文をコピペで取ってきて自力でパースするという方法もありますが、それは手間がかかり、やりたくありません。
そこで、LLMを使ってパースする方法を考えました!
試しに最近流行りのClaude3のHaikuでGUIを使ってPDFから本文抽出してみたところ、以下のようにうまくいきました。
いい感じにできるんですよ。Haikuは高速で、体験めっちゃいいです。
Claude3内では、PDFをドロップした瞬間にPDFパーサーかなんかを使ってパースして保持しているようです。
それを見ながらHaikuが本文抽出してくれるので、これとおんなじスキームを使えば本文抽出もいい感じにできるのでは?と思い実装してみました。
さらに、Unstructuredを使えば、画像やテーブル、キャプションの抽出までできてしまいます。一般的なPDFパーサーと比べると、処理速度は劣るかもしれませんが、高精度なPDFパーサーが作れるのです。
以下が実装の全体です。
成果物として、以下のものが出力されます。
- PDFから本文のみ抽出したファイル
- PDFからFigureとTableを抽出した画像
- Figureとそのキャプションの対応が記載されてるJSONファイル
では実装の詳細を簡単に見ていきましょう。
※今回の実装は二つほどのPDFを用いてうまくいったかどうかしか試せていません。コーナーケースを考慮した実装になっていませんのでご容赦ください。
Low Performance PDF Parserもどきを実装
では実装を一部解説していきます。
実際に実装しているものからかなり省略して書いていきますので、正常に動かない可能性があります。
その場合は、GitHub側のコードを見てください。
テストも特に書いてないので怪しいコードもありますので、ご容赦ください。
実装は、Colab上でPythonでLangchain,unstructured,PyMuPDFを使います。
LLMはGPT3.5を使用します。(Haikuを使っていない理由は後述してます。)
必要ライブラリをInstall
まずは、必要なライブラリをinstallします。
!apt-get install -y libmagic-dev poppler-utils tesseract-ocr
%pip install "unstructured[pdf]" unstructured langchain PyMuPDF openai langchain_openai
LLMを使った文章抽出を実装。
以下のステップで文章を抽出してきたいと思います。
- PyMuPDFでpdfをパースする。
- パースしたPDFを分割する
- 一つ前の会話を用いて、HaikuでパースしたPDFのテキストから本文抽出
1. PyMuPDFでpdfをパースする
基本最初のパーサーはどれでもいいとは思っていますが、使い慣れてるPyMuPDFを使用します。
unstructuredでもPDFをパースできるので、依存ライブラリを減らしたい人はそちらを使ってください。
import fitz # PyMuPDF
def extract_text_from_pdf_range_and_save(pdf_path):
doc = fitz.open(pdf_path)
text = ""
# 指定されたページ範囲内の各ページを反復処理
for page_num in range(doc.page_count):
# ページオブジェクトを取得
page = doc.load_page(page_num)
# ページからテキストを抽出
text += page.get_text()
# PDFドキュメントを閉じる
doc.close()
return text
2. パースしたPDFを分割する
分割はGPT3.5が出力できるトークンの最大値を設定しています。
と言うのも、LLMの役割としてパースした内容を綺麗にすることを想定しているので、入力値と同じぐらいの文字数の出力が返ってくるはずです。入力トークンが大きいと、出力が伴わないので出力トークンに合わせた方がいいでしょう。
text_splitter = CharacterTextSplitter(
separator="",
chunk_size=4096,
chunk_overlap=0,
length_function=len,
is_separator_regex=False,
)
segments = text_splitter.split_text(preprocess_text)
3. 一つ前の会話を用いて、HaikuでパースしたPDFのテキストから本文抽出
一個前までの会話だけを記憶させるようにしています。大体一個前まで覚えていればPDFの本文の抽出はうまくいっていました。
いい実装がどんなかわからなかったので、これよりいい実装があるかもしれません。
また、使うLLMによっては、RateLimitに制限がかかるかもしれないので、適宜timeなどで休ませてあげてください。
from langchain_core.prompts import ChatPromptTemplate
from langchain_core.runnables.history import RunnableWithMessageHistory
from langchain_community.chat_message_histories import ChatMessageHistory
from langchain_core.chat_history import BaseChatMessageHistory
from langchain_text_splitters import CharacterTextSplitter
def process_segments_with_langchain(segments):
store = {}
def get_session_history(session_id: str) -> BaseChatMessageHistory:
if session_id not in store:
store[session_id] = ChatMessageHistory()
return store[session_id]
system = (
"You are an excellent text extractor. You remove headers, footers and figure captions that are not relevant to the body text. Don't skip the headline.The output is always the body text only, with no explanations."
)
human = "{text}"
prompt = ChatPromptTemplate.from_messages([("system", system), MessagesPlaceholder(variable_name="chat_history"), ("human", human),])
runnable = prompt | chat
with_message_history = RunnableWithMessageHistory(
runnable,
get_session_history,
input_messages_key="text",
history_messages_key="chat_history",
)
session_id = "unique_session_id"
processed_segments = []
for i, segment in enumerate(segments):
response = with_message_history.invoke(
{"text": segment},
config={"configurable": {"session_id": session_id}},
)
processed_segments.append(response)
# 終わるたびに記憶をリセットして、さっきまでの会話の記憶を注入する。
store[session_id].clear()
prememory=ChatMessageHistory()
prememory.add_user_message(segment)
prememory.add_ai_message(response.content)
store[session_id] = prememory
return processed_segments
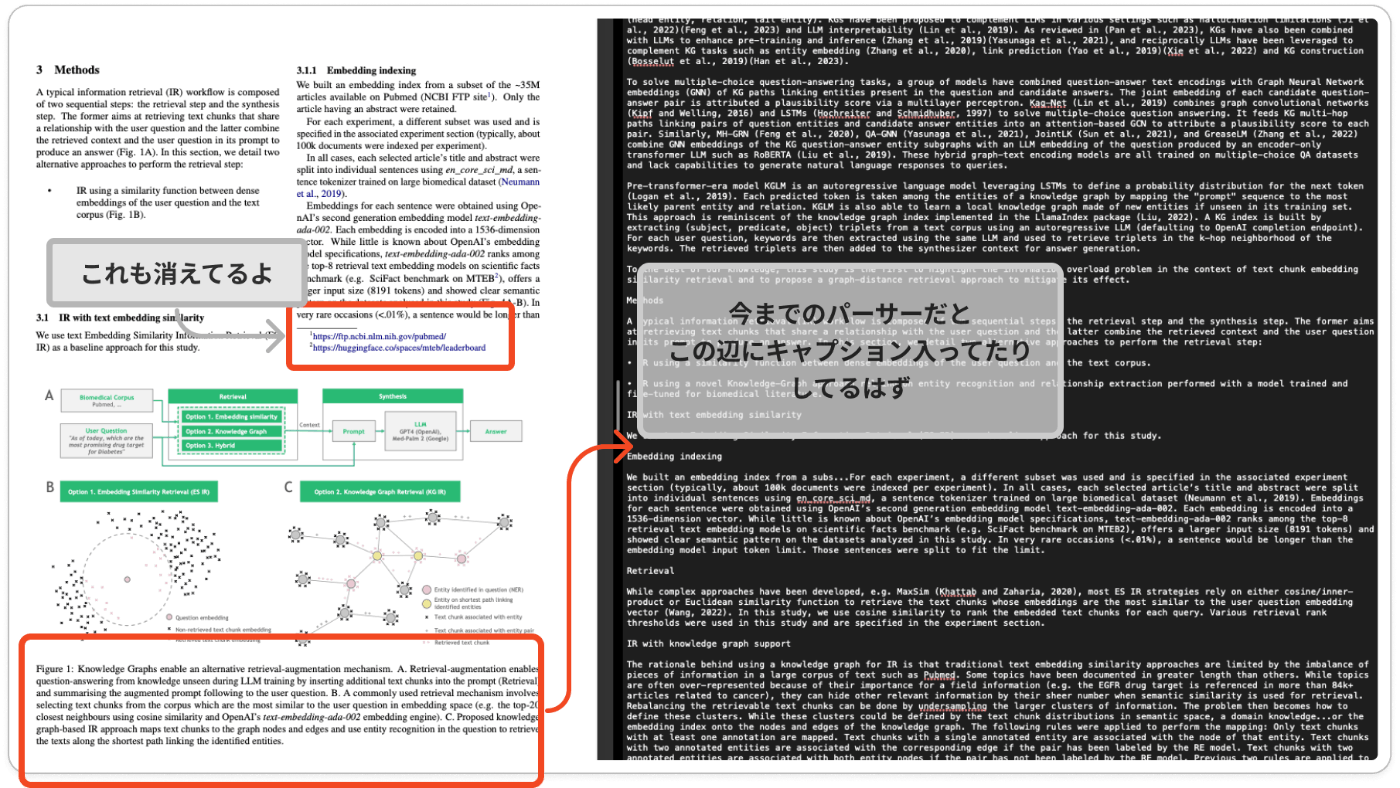
本文抽出結果(Haikuを使わなかった理由も記載)
これで本文抽出は割と綺麗にできていますが、若干まだよくないところも見られます。
-
綺麗にできているところ
いい感じです。
-
できていないところ
一部キャプションが混ざってますね。

実用性を考えるとHaikuを使用した方がいいと考えられます。
実は最初はHaikuでやっていたんですが、Free TierのAPIを使用していたので検証中にTPD(1日あたりの使用可能なトークン数)でRateLimitがきてしまってGPT3.5に途中から切り替えました。
Claude3にしっかりとお金を落とそうと思います。
Unstructuredを使った図・テーブル・キャプション抽出を実装
あとは図・テーブル・キャプションが抽出できれば、PDFを余すことなく構造化して使えますね。
以下の流れで実装します。
- Unstructuredで図、テーブル、キャプションを抽出
- 図とキャプションの紐付け
1. Unstructuredで図、テーブル、キャプションを抽出
unstructuredはPDFを画像として扱って、OCRを図やテーブルを認識して抜き出しています。(筆者調べ)
なのでよくあるPDFの「XObject」で抜き出す的なルールベースの抜き出しではありません。
ルールベースの抜き出しはかなり早いのですが、経験上うまくいくことがかなり少なかったです。
なので、時間はかかりますが、こちらを使った方がうまくいくと思います。
from unstructured.partition.pdf import partition_pdf
def extract_images_and_captions(filename):
elements = partition_pdf(filename=filename, strategy="hi_res",
extract_images_in_pdf=True,
extract_image_block_types=["Image","Table"],
extract_image_block_to_payload=False,
extract_image_block_output_dir="./images")
tables = [el for el in elements if el.category == "Image"]
caption = [el for el in elements if el.category == "FigureCaption" or el.text.lower().startswith(("figure", "fig"))]
return tables, caption
うまく切り抜けています。

ただ一つ困ったことがあります。それは、1つのFigureが分割されて取り出される可能性があります。
これの何が問題かと言うとキャプションを単にFigureの順番で上から紐づけることができないのです。
2. 図とキャプションの紐付け
今回はルールベースで紐付けを行います。
Unstructuredでの抜き出しではありがたいことに、何ページの、どの位置にあったかがわかるのです。
なので以下のルールで紐付けを行います。
同じページで、図よりy軸が下になる、一番近いキャプションを紐づける
このルールが全てのPDFでうまくいくかは正直微妙です。実際に試したPDFは2つほどなので、コーナーケースは取れていないと思います。
「図とキャプションが別ページ」とか、「キャプションが右にある」とかの場合はうまくいきません。その時はまた考えましょう。
実装は長くなるので省略します。気になる方はGitHubを確認してください。
結果
[
{
"id": "195ca66879fc18743909ca4c1ad3d286",
"category": "Image",
"name": "Figure 1",
"caption": "Figure 1: Knowledge Graphs enable an alternative retrieval-augmentation mechanism. A. Retrieval-augmentation enables question-answering from knowledge unseen during LLM training by inserting additional text chunks into the prompt (Retrieval) and summarising the augmented prompt following to the user question. B. A commonly used retrieval mechanism involves selecting text chunks from the corpus which are the most similar to the user question in embedding space (e.g. the top-20 closest neighbours using cosine similarity and OpenAI’s text-embedding-ada-002 embedding engine). C. Proposed knowledge graph-based IR approach maps text chunks to the graph nodes and edges and use entity recognition in the question to retrieve the texts along the shortest path linking the identified entities.",
"image_path": "./images/figure-3-1.jpg",
"image_text": "A Biomedical Corpus Rte Pubmed, ... User Question “As of today, which are the most promising drug target for Diabetes” HI Option 2. Knowledge Graph HI Option 3. Hybrid SCT LLM GPT4 (OpenAl), Med-Palm 2 (Google) Answer"
},
{
"id": "81986483fcb74beabf062b9078ce1769",
"category": "Image",
"name": "Figure 1",
"caption": "Figure 1: Knowledge Graphs enable an alternative retrieval-augmentation mechanism. A. Retrieval-augmentation enables question-answering from knowledge unseen during LLM training by inserting additional text chunks into the prompt (Retrieval) and summarising the augmented prompt following to the user question. B. A commonly used retrieval mechanism involves selecting text chunks from the corpus which are the most similar to the user question in embedding space (e.g. the top-20 closest neighbours using cosine similarity and OpenAI’s text-embedding-ada-002 embedding engine). C. Proposed knowledge graph-based IR approach maps text chunks to the graph nodes and edges and use entity recognition in the question to retrieve the texts along the shortest path linking the identified entities.",
"image_path": "./images/figure-3-2.jpg",
"image_text": "x x gt EES ¢~ © Question embedding x x XSL Lee ye * Non-retrieved text chunk embedding x x * Retrieved text chunk embedding QO Entity identified in question (NER) Entity on shortest path linking identified entities x Text chunk associated with entity Text chunk associated with entity Retrieved text chunk"
},
{
"id": "a21ebc9eb313848ae00d9c01317c37d7",
"category": "Image",
"name": "Figure 2",
"caption": "Figure 2: Annotations from scientific articles structure the biomedical knowledge graph used for retrieval. A. A scientific article is annotated by extracting entities (NER) and the type of relationships linking entities (RE). B. Each sentence containing 1+ entity is mapped onto the knowledge graph, either on a link related two entities if it contains two entities that are linked semantically (+ sign) or to its individual entities otherwise (× sign).",
"image_path": "./images/figure-5-3.jpg",
"image_text": "> J Virol. 2020 Jun 16:94(13):e02161-19. doi: 10.1128/JV1.02161-19. Print 2020 Jun 16. Neutrophil-Airway Epithelial Interactions Result in Increased Epithelial Damage and Viral Clearance during Respiratory Syncytial Virus Infection Yu Deng * 2, Jenny A Herbert \", Elisabeth Robinson ', Luo Ren ' 2, Rosalind LSmyth * 7, Claire M Smith * 3 PMID: 32295918 PMCID: PMC7307165 DOK: 10.1128/JV1.02161-19 Abstract Respiratory syncytial virus (RSV) is a major cause of pediatric respiratory disease, Large numbers of neutrophils are recruited into the airways of children with severe RSV disease. Itis not clear whether or how neutrophils enhance recovery from disease or contribute to its pathology. Using an in vitro model of the differentiated airway epithelium, we found that the addition of physiological concentrations of neutrophils to RSV-infected nasal cultures was associated with greater epithelial damage with lower ciliary activity, cilium loss, less tight junction expression/(ZO-1), and more detachment of epithelial cells than is seen with RSV infection alone. This was also associated with a decrease in infectious virus and fewer RSV-positive cells in cultures efter neutrophil exposure than in preexposure cultures. Epithelial damage in response to RSV infection was associated with neutrophil activation (within 1h) and neutrophil degranulation, with significantly greater cellular expression of CD11b and myeloperoxidase and higher levels of neutrophil elastase and myeloperoxidase activity in apical surface media than in media with mock-infected airway epithelial cells (AECs). We also recovered more apoptotic neutrophils from RSV-infected cultures (>40%) than from mock-infected cultures (<5%) after 4h. The results of this study could provide important insights into the role of neutrophils in host response in the airway. This study shows that the RSV-infected human airway drives changes in the behavior of human neutrophils, including increasing activation markers and Q *O: Gene Name: ITGAM Alt. Name: CD11B NCBI ID: 3684 O: + x + Disease Name: Respiratory *O) Syncytial Virus Infection MesH ID: 0018357 Compound Name: Myeloperoxidase ChEMBL ID: CHEMBL2107858 Gene Name: ELANE Alt. Name: elastase, neutrophil expressed NCBI ID: 1991 Gene Name: TJP1 Alt. Name: ZO-1 NCBI ID: 7082 delaying epoptosis, that result in greater airway damage and viral clearance. © cere © disease @ vue"
},
{
"id": "7ae74c5b2bee207b49f16406d19ac9d0",
"category": "Image",
"name": "Figure 3",
"caption": "Figure 3: Retrieval performance comparison between embedding similarity IR (blue), knowledge graph IR (orange) and hybrid method (green). The metrics compare each method’s retrieval performance for the same task: retrieving biomedical text chunks which are relevant to the question “What are the known drug targets for treating <disease>?” over 8 diseases: asthma, pulmonary arterial hypertension, heart failure, hypertension, Parkinson’s disease, Alzheimer’s disease, liver cirrhosis, inflammatory bowel disease. Solid lines indicate the metric averages and transparent ribbon 95% confidence intervals. A-B. Recall@K and Precision@K. C. Number of clusters containing at least one retrieved text chunk, among the 200 clusters defined in 1536-dimension embedding space.",
"image_path": "./images/figure-6-4.jpg",
"image_text": "A Precision@K B Recall@k 0.8 0.4 — Embedding Similarity IR — Knowledge Graph IR 03 — Hybrid IR 0.6 _ 0 2 = 2 & 0.4 GS 0.2 o @ & a 01 0.2 0.0 £ 0.0 0 250 500 750 1000 0 250 K C Cluster Coverage © 3S a oS Number of clusters N > oO o 500 750 1000 0 250 500 K K 750 1000"
},
{
"id": "a82a195a09c9150f7411e6615cc677fd",
"category": "Image",
"name": "Figure 4",
"caption": "Figure 4: Characterization of differences between the ES IR and KG IR methods over the text embedding landscape. Each plot represents the ~731k 1536-dimensional text chunk embeddings in two dimensions via UMAP transformation. A. The biomedical entity landscape illustrates the entity(ies) present in each text chunk. Text chunks containing a pair of genes (resp. drugs) are represented with same colour as text chunks containing a single gene (resp. drug). B. Disease area landscape. Only text chunks containing 1+ disease entity are represented. C. Question similarity landscape, colours indicate the cosine similarity of the text chunk embedding with the question embedding (“What are the known drug targets for treating Asthma?”, also in D-E). Arrows indicate remote spots of text chunks that are most similar to the question embedding. D. High-density retrieval regions indicates the parts of the landscape where both methods are retrieving most of their chunks for K=200. Arrow indicates a secondary cluster of high-density retrieval for KG IR. Black dots represent the 355 text chunks that are part of the gold-standard dataset. E. Granular comparison of the retrieved documents for K=200. All dots represent the 442 curated text chunks.",
"image_path": "./images/figure-9-5.jpg",
"image_text": "A UMAP 1 . B Text chunk entity Disease Drug Disease areas Gene lm Nervous System _ Mental Disorders | Cardiovascular Ml Respiratory lll Digestive System Disease-Gene Disease-Drug Gene-Drug No entity Sines Question Similarity Landscape High Text chunk r “ similarity with Question embedding x question embedding | ° Curated chunks Low Ee ©) Knowledge Graph IR - ©) Embedding Similarity IR X Question embedding Curated chunk retrieved by KG IR Curated chunk retrieved by ES IR Retrieved by both models Unretrieved curated chunk : e e e Curated chunk e x Question embedding"
}
]
最後に
これで前もってドキュメントを準備しておくRAGを作る場合はある程度綺麗なドキュメントが使えますね!
画像もナレッジグラフでドキュメントと図やテーブルを紐づけておくとRAGで引っ張ってこれるのでかなり有用ですよ。
次回は、Haiku APIでの検証+表の扱いを調整+コーナーケースの対応
ぐらいはやりたいと思います!
Xやってるのでぜひフォローお願いします。
@hudebakonosoto
Discussion