Kaggle Titanic で AWS を使った ML パイプライン構築 1
機械学習パイプラインの構築は、機械学習モデルの運用工数削減や、統一された学習・テスト環境を組み込むことで、学習の精度を評価し、不具合のある機械学習モデルの誤ったデプロイを防止できるため、MLOps の第一歩として取り組むことが多いのではないでしょうか。特にクラウドでサーバーレスな構成にすることは、運用工数少なく機械学習パイプラインを構築できることから、事例も増えてきているようです。今回はワークフローエンジンとして馴染みの方も多いであろう、AWS Step Functions を使って、機械学習パイプラインを構築する方法について触れてみたいと思います。
今回は、Kaggle にある Titanic のチュートリアルに沿って AWS で機械学習パイプラインを構築してみようと思います。前提として、S3 に今回使うデータが格納されているところから始めます。特徴量の作成や欠損の処理といった前処理部分や、学習、推論といったそれぞれのステップにおいて計算リソースとして、Amazon SageMaker の機能を使います。それぞれのステップはジョブの開始時に Docker コンテナが立ち上がり、終わったら消えるため、いちいち計算リソースの管理をする必要がなく便利です。さらに、それぞれのステップをワークフローとして束ねる部分にサーバーレスなワークフローエンジンである AWS Step Functions を使います。
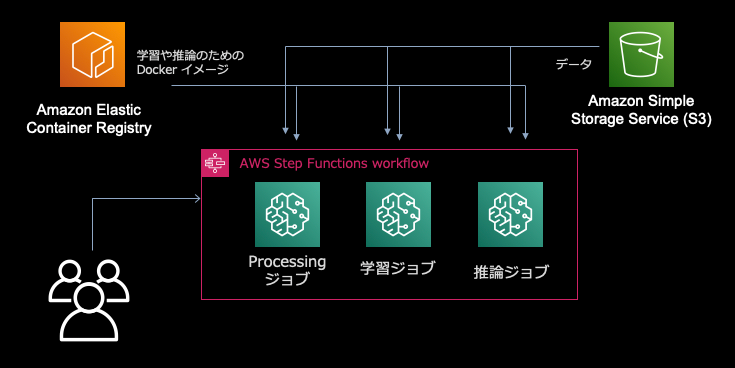
前処理、学習、推論といったそれぞれのステップで計算リソースが異なり、かつ変更頻度も違うので、分割することによって、計算リソースの最適化や、柔軟な改変が可能になります。
Amazon SageMaker での前処理、学習、推論
SageMaker ではデータの前処理や学習、バッチ推論とそれぞれに対応したジョブを発行する機能があります。例えば学習ジョブではハイパラの調整を並列化して効率的に行いたい、推論は学習と違って、処理そのものを並列化できる、など、それぞれの過程においてやりたいことや、求められる機能が違うため、異なるジョブを発行する形になっています。また推論では、リアルタイム推論を実現する推論APIサーバーを簡単に作成する機能も備わっています。今回は下記の3つを使います。
- SageMaker Processing: シンプルに Docker コンテナを実行し、処理が終われば仮想環境ごとコンテナは消えます。保存内容は S3 に自動的に退避させることができます。
- SageMaker 学習ジョブ: Processing ジョブのようにコンテナの実行に加えて、ハイパラの調整や実験管理など、様々な機能をアドオンできます。こちらも仮想環境が起動し、処理が終わると消えるため、S3を経由してファイルのやり取りを行います。その際に、S3 とコンテナ内でのファイルの受け渡しパスが環境変数に既に指定された形で保存されているため、それを使う必要がります。
- SageMaker バッチ変換ジョブ: バッチ推論を行うためのジョブ。学習ジョブと同様に、S3 とコンテナ内でのファイルの受け渡しパスに指定されたものを使う必要があります。
今回の Titanic のタスクにどう使うか簡単に見てみましょう。特徴量作成や学習などについては、Titanic: random forest using sklearn and pandasを参考にさせて頂きました。
データの前処理ジョブの実行
今回は、下記のような Dockerfile を定義し、ECR へ登録してつかいました。
FROM python:3.7-slim-buster
RUN pip3 install pandas scikit-learn
ENV PYTHONUNBUFFERED=TRUE
ENTRYPOINT ["python3"]
そして、特徴量作成を行うスクリプトは下記のようにだとします。スクリプトの記述で注意する点は、スクリプトへのデータの入力と出力です。
import joblib
import os
import argparse
import pandas as pd
def deriveChildren(age: int, parch: int) -> int:
""" 連れている両親の数だけを抽出。18歳以上は両親がいないと仮定。
Args:
age (int): 搭乗者の年齢
parch (int): 両親、子供の数
Returns:
両親の数 (int)
"""
if(age < 18):
return parch
else:
return 0
if __name__ == '__main__':
parser = argparse.ArgumentParser()
parser.add_argument('--data_type', type=str)
parser.add_argument('--input_dir', type=str, default=None)
parser.add_argument('--output_dir', type=str, default=None)
args, _ = parser.parse_known_args()
input_data_path = os.path.join(args.input_dir, data_type+'.csv')
output_data_path = os.path.join(args.output_dir, data_type+'.csv')
data = pd.read_csv(input_data_path)
data = data.drop(['Cabin'], axis=1)
data = data.dropna(how="any")
data = data.assign(children=data.apply(lambda row: deriveChildren(row['Age'], row['Parch']), axis = 1))
data = data.drop(['Name', 'Parch', 'SibSp', 'title', 'Ticket', 'PassengerId'], axis=1)
これらの Docker イメージと、processing.py をジョブに投げる際には、SageMaker Python SDK で下記のようにジョブ定義し、SageMaker の AIP を叩きます。特にファイルの受け渡しでは、Processing ジョブの発行時に、どこでファイルを受け渡すのかを指定し、処理用スクリプト内で、args.input_dir と args.output_dir で受け取っています。
processing_input_dir = '/opt/ml/processing/input'
processing_output_dir = '/opt/ml/processing/output'
output_s3_path = 's3://' + sagemaker_session.default_bucket() + '/kaggle-ml-pipeline'
output_s3_path_preprocess = output_s3_path + '/preprocessed'
processor = ScriptProcessor(
base_job_name=job_name,
image_uri=image_uri,
command=['python3'],
role=role,
instance_count=1,
instance_type='ml.c5.xlarge'
)
processor.run(
code='./scripts/preprocess/preprocess_script/preprocess.py', # S3 の URI でも可
inputs=[ProcessingInput(source=input_train, destination=processing_input_dir)],
outputs=[ProcessingOutput(source=processing_output_dir, destination=output_s3_path_preprocess)],
arguments=[
'--data_type', 'train',
'--input_dir',processing_input_dir,
'--output_dir',processing_output_dir
]
)
学習ジョブの実行
学習でも、Docker イメージと学習用スクリプトを準備するのに変わりはありません。Processing ジョブと異なるのは、入出力のパスが SageMaker で既に指定されているものを使う点のみです。詳細はドキュメントを確認頂くと早いですが、下記のように、環境変数経由で指定されたパスを読み込み、そこに保存されたデータを読み込んだり、学習済モデルを保存したりしています。
import argparse
import os
import pandas as pd
import joblib
from sklearn.ensemble import RandomForestClassifier
if __name__ == '__main__':
parser = argparse.ArgumentParser()
parser.add_argument('--output-data-dir', type=str, default=os.environ['SM_OUTPUT_DATA_DIR'])
parser.add_argument('--model-dir', type=str, default=os.environ['SM_MODEL_DIR'])
parser.add_argument('--train', type=str, default=os.environ['SM_CHANNEL_TRAIN'])
args = parser.parse_args()
train_path = os.path.join(args.train, 'train.csv')
train = pd.read_csv(train_path)
X_train, y_train = train.drop(['Survived'], axis=1), train['Survived']
y_train = y_train.astype({"Survived": int})
rf = RandomForestClassifier(n_estimators=100, n_jobs=-1)
joblib.dump(rf, os.path.join(args.model_dir, "model.joblib"))
今回は、学習ジョブには AWS で用意されている scikit-learn や pandas などが既にインストールされている Docker コンテナを使用しました。from sagemaker.sklearn.estimator import SKLearn することでそのコンテナを使用することができます。
from sagemaker.sklearn.estimator import SKLearn
output_s3_path_train = output_s3_path + '/train'
sklearn = SKLearn(
entry_point='scripts/train/train.py',
framework_version="0.23-1",
instance_type="ml.m5.xlarge",
output_path=output_s3_path_train,
role=role)
train_input = output_s3_path_preprocess + '/train.csv'
sklearn.fit({'train': train_input})
保存されているモデルは、SageMaker 特有のものではなく、どの環境でも動く、一般的なモデルファイルとなっています。
推論ジョブの実行
SageMaker のバッチ推論では、学習ジョブで出力されたモデルファイルと、その際に使われた環境(つまり Docker イメージ)、推論スクリプトを使って推論ジョブを実行することができます。このモデルファイルと Docker イメージ、実行ファイルをひとまとめにしたものを SageMaker の概念として Model と呼びます。AWS が準備している Docker コンテナには、推論実行のためのスクリプトが既に準備されているので、train.py の中に model_fn として学習済モデルを読み込みさえすれば、推論を実行してくれます。詳細はこちらから確認できます。
fit() 後の sklearn インスタンスはこの Model クラスとなっていますので、そのまま推論ジョブを実行できます。
output_s3_path_inference = output_s3_path + '/batch_inference'
transformer = sklearn.transformer(
instance_count=1,
instance_type='ml.m5.xlarge',
output_path=output_s3_path_inference
)
test_input = output_s3_path_preprocess + '/test.csv'
transformer.transform(
data=test_input,
content_type='text/csv')
transformer.wait()
まずは、それぞれのジョブを実行する流れを確認しました。次回 、これらのジョブを AWS Step Functions でパイプライン化します。
次回記事
Discussion