状態空間モデル 論文解説⑤「 Mamba-2 」
論文
前回 は Mamba を解説しました。今回はその正当進化モデルである Mamba-2 です。
タイトル
Transformers are SSMs: Generalized Models and Efficient Algorithms Through Structured State Space Duality
論文: https://arxiv.org/pdf/2405.21060
Github: https://github.com/state-spaces/mamba
解説ステップ
概要
- 行列Aを対角からスカラーに変更した
- それ(とSSD※後述※)によりメモリ効率化を実現し、内部状態次元数が拡張された
- アーキテクチャーを一部変更し、Multi-head の概念を取り入れた
- 結果、性能アップとスピードアップを実現した
以上が、Mamba-2のモデルに対する概要になりますが、論文では多くの部分を以下について言及しています。
- Attention と SSM の比較
- SSD アルゴリズムの一般化
実は、SSDなどの高速計算部分などの理解を無視する場合、Mamba からの変更点は軽微です。しかし、その無視できる箇所が難解で、論文の理解を難しくしています。
本記事では、あくまで Mamba-2 のモデル構造に主眼を置きそれを軸に解説し、さらに後半で私が理解したところまでの「その他」の解説を試みます。
Mamba からの変更点
Mamba からのアップデートは、内部状態次元数を増やす 事が主眼だと思われます。なので、基本的な構造は似ています。
パラメータA(とD)をスカラーに変更
モデル構造 の章に詳細を書きますが、パラメータ
The structure on
is further simplified from diagonal to scalar times identity structure. Each 𝐴 can also be identified with just a scalar in this case. A_t
※デフォルト設定では、パラメータ
Multi-head 化
パラメータ
アーキテクチャーを変更
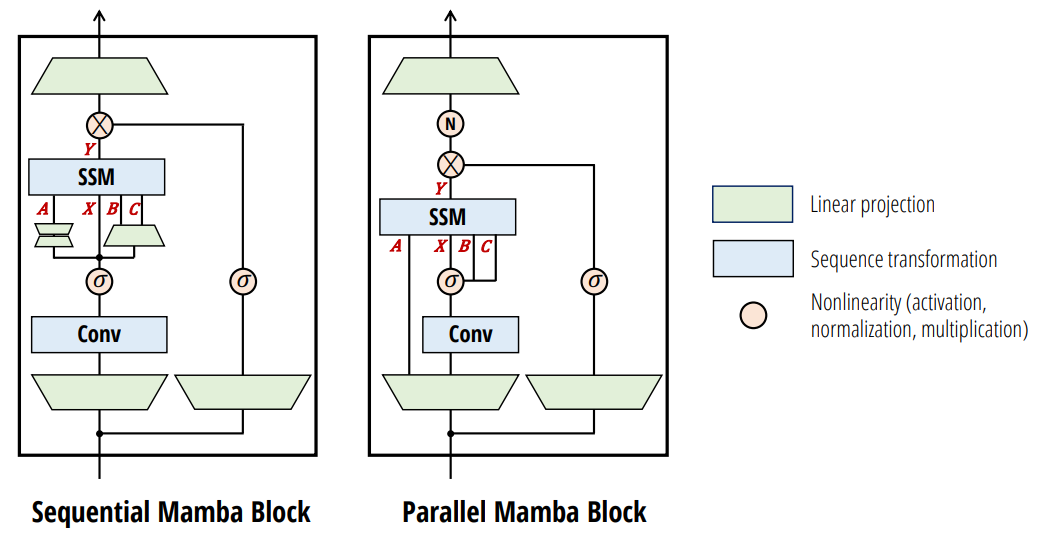
上図で、左:Mamba 右:Mamba-2 です。計算方法や、Aのパラメータの違いはありますが、構造レベルでいえばこれ以外は Mamba と同じです。
モデル構造 の章に詳細を書きます。
モデル構造
Pythonの簡易環境構築は前回資料を参考にしてください。
>>> model = MixerModel(128, 4, 128, 50277, ssm_cfg={"layer": "Mamba2", "d_ssm": 64, "headdim": 32})
>>> model
MixerModel(
(embedding): Embedding(50277, 128)
(layers): ModuleList(
(0-3): 4 x Block(
(norm): LayerNorm((128,), eps=1e-05, elementwise_affine=True)
(mixer): Mamba2(
(in_proj): Linear(in_features=128, out_features=770, bias=False)
(conv1d): Conv1d(320, 320, kernel_size=(4,), stride=(1,), padding=(3,), groups=320)
(act): SiLU()
(norm): RMSNorm()
(out_proj): Linear(in_features=256, out_features=128, bias=False)
)
(norm2): LayerNorm((128,), eps=1e-05, elementwise_affine=True)
(mlp): GatedMLP(
(fc1): Linear(in_features=128, out_features=256, bias=False)
(fc2): Linear(in_features=128, out_features=128, bias=False)
)
)
)
(norm_f): LayerNorm((128,), eps=1e-05, elementwise_affine=True)
)
Mamba-2 Layer
コードを図に書き起こすと以下になります。
B # バッチ数
T # 時系列長. sequence length
D # 入力次元. 言語モデルにおいてはトークンの embedding 次元
H = 2 # Head の数
P = 32 # 各 Head の次元数
N = 128 # SSMの内部状態(h)の次元数
G = 1 # B,Cのグループの数
Dssm = Nh x P # SSMに入力するxの次元数
Dmlp = 192 # Mamba Layer内部の MLP の次元数
Din = Dssm + Dmlp
K = 4 # 1次元畳み込みのカーネルサイズの初期値
各変数のshapeなど
>>> from mamba_ssm.modules.mamba2 import *
>>> self = model.layers[0].mixer
>>> self
Mamba2(
(in_proj): Linear(in_features=128, out_features=770, bias=False)
(conv1d): Conv1d(320, 320, kernel_size=(4,), stride=(1,), padding=(3,), groups=320)
(act): SiLU()
(norm): RMSNorm()
(out_proj): Linear(in_features=256, out_features=128, bias=False)
)
>>> u = torch.rand(1, 16, 128)
>>> batch, seqlen, dim = u.shape
>>> conv_state, ssm_state = None, None
>>> zxbcdt = self.in_proj(u) # (B, L, d_in_proj) or (B * L, d_in_proj)
>>> A = -torch.exp(self.A_log.float()) # (nheads) or (d_inner, d_state)
>>> d_mlp = (zxbcdt.shape[-1] - 2 * self.d_ssm - 2 * self.ngroups * self.d_state - self.nheads) // 2
>>> z0, x0, z, xBC, dt = torch.split(
zxbcdt,
[d_mlp, d_mlp, self.d_ssm, self.d_ssm + 2 * self.ngroups * self.d_state, self.nheads],
dim=-1
)
>>> for x in [z0, x0, z, xBC, dt]: print(x.shape)
torch.Size([1, 16, 192])
torch.Size([1, 16, 192])
torch.Size([1, 16, 64])
torch.Size([1, 16, 320])
torch.Size([1, 16, 2])
>>> self.conv1d(xBC.transpose(1, 2)).shape
torch.Size([1, 320, 19])
>>> self.conv1d(xBC.transpose(1, 2)).transpose(1, 2).shape
torch.Size([1, 19, 320])
>>> xBC = self.act(
self.conv1d(xBC.transpose(1, 2)).transpose(1, 2)[:, :-(self.d_conv - 1)]
)
>>> xBC.shape
torch.Size([1, 16, 320])
>>> x, B, C = torch.split(xBC, [self.d_ssm, self.ngroups * self.d_state, self.ngroups * self.d_state], dim=-1)
>>> for x in [x, B, C]: print(x.shape)
torch.Size([1, 16, 64])
torch.Size([1, 16, 128])
torch.Size([1, 16, 128])
>>> dt = F.softplus(dt + self.dt_bias.to(dtype=dt.dtype))
>>> dA = torch.exp(dt * A)
>>> x = rearrange(x, "b l (h p) -> b l h p", p=self.headdim)
>>> dB = torch.einsum("blh,bln->blhn", dt, B)
>>> dBx = torch.einsum("blhn,blhp->blhpn", dB, x)
>>> ssm_state = torch.rand(dBx.shape)
>>> ssm_state = ssm_state * rearrange(dA, "b l h -> b l h 1 1") + dBx
>>> ssm_state.shape
torch.Size([1, 16, 2, 32, 128])
>>> y = torch.einsum("blhpn,bln->blhp", ssm_state.to(dtype), C)
>>> y = y + rearrange(self.D.to(dtype), "h -> h 1") * x

補足① SSD アルゴリズム
敢えて、補足と書きます。Mamba の Selective Scan もそうですが、これらは計算とメモリ効率化の話であり、モデル構造そのものの本質ではないからです。
論文では当然重要と捉えていますが、あえて本記事では、このスタンスで解説を進めたいと思います。
SSM を行列Mで書き直す
SSMは次のように書けます。
これを一般化すると以下のようになります。
つまり以下のように一般化できます。
N-SSS (N-sequentially semiseparable) 行列
上述の行列
要素別にまとめると以下です。
N-semiseparable 行列 ※本記事ではほとんど関係ありません
次のようなベクトル列とスカラー
これらを使って以下のように行列Sが書けるとします。
するとこの行列Sは次のように分解でき、簡略化できます。
※tril や triu は下三角、上三角へのマスク処理です。
N-SSS の一般系
N-SSのさらに特殊な形を表します。次のような
スカラー列
ベクトル列
行列 列
があります。これらを使って以下のように行列Sが書けるとします。
これを要素別にまとめると以下のようになります。
1-SS 行列
まず、1-SSS を記述してみましょう。
これをさらに分解すると、次のようになります。
この真ん中の
N-SSSをチャンクに分けて構造化
さて、Mamba の Selective Scan にあたる箇所を、より高速化した計算方法を解説します。行列
実はチャンクに分ける事で、

この N-SSS 行列は適当なチャンクに分割した際、さらに同じような構造が現れます。上図ではチャンクサイズ

上図の左側は、計算結果を連続している絵ですが、これは疑似コードの解説まで触れた時に理解できるかと思います。
さて、行列
この時、対角成分は以下のようになります。
それ以外の成分(
さて、改めて
するとどうでしょう。再度、N-SSS 行列が現れました。このように、N-SSSは別のN-SSSの構造で書き表すことができます。
同様に
こちらをさらに整理すると
Mamba と同様、この構造を並列化を駆使して計算します。
SSDの具体的な計算
以下は「Listing 1 Full PyTorch example of the state space dual (SSD) model」の疑似コードをそのまま使えるように少し修正したプログラムです。
import torch
from einops import rearrange
import torch.nn.functional as F
def segsum(x):
"""Naive segment sum calculation. exp(segsum(A)) produces a 1-SS matrix,
which is equivalent to a scalar SSM."""
T = x.size(-1)
x_cumsum = torch.cumsum(x, dim=-1)
x_segsum = x_cumsum[..., :, None] - x_cumsum[..., None, :]
mask = torch.tril(torch.ones(T, T, device=x.device, dtype=bool), diagonal=0)
x_segsum = x_segsum.masked_fill(~mask, -torch.inf)
return x_segsum
def ssd(X, A, B, C, block_len=64, initial_states=None):
"""
Arguments:
X: (batch, length, n_heads, d_head)
A: (batch, length, n_heads)
B: (batch, length, n_heads, d_state)
C: (batch, length, n_heads, d_state)
Return:
Y: (batch, length, n_heads, d_head)
"""
assert X.dtype == A.dtype == B.dtype == C.dtype
assert X.shape[1] % block_len == 0
# Rearrange into blocks/chunks
X, A, B, C = [rearrange(x, "b (c l) ... -> b c l ...", l=block_len) for x in (X, A, B, C)]
A = rearrange(A, "b c l h -> b h c l") # b: B, h: H, c: T/Q, l: Q
A_cumsum = torch.cumsum(A, dim=-1)
# 1. Compute the output for each intra-chunk (diagonal blocks)
L = torch.exp(segsum(A))
Y_diag = torch.einsum("bclhn,bcshn,bhcls,bcshp->bclhp", C, B, L, X) # n: N, s: Q, p: P
# 2. Compute the state for each intra-chunk
# (right term of low-rank factorization of off-diagonal blocks; B terms)
decay_states = torch.exp((A_cumsum[:, :, :, -1:] - A_cumsum))
states = torch.einsum("bclhn,bhcl,bclhp->bchpn", B, decay_states, X)
# 3. Compute the inter-chunk SSM recurrence; produces correct SSM states at chunk boundaries
# (middle term of factorization of off-diag blocks; A terms)
if initial_states is None:
initial_states = torch.zeros_like(states[:, :1])
states = torch.cat([initial_states, states], dim=1)
decay_chunk = torch.exp(segsum(F.pad(A_cumsum[:, :, :, -1], (1, 0))))
new_states = torch.einsum("bhzc,bchpn->bzhpn", decay_chunk, states)
states, final_state = new_states[:, :-1], new_states[:, -1]
# 4. Compute state -> output conversion per chunk
# (left term of low-rank factorization of off-diagonal blocks; C terms)
state_decay_out = torch.exp(A_cumsum)
Y_off = torch.einsum('bclhn,bchpn,bhcl->bclhp', C, states, state_decay_out)
# Add output of intra-chunk and inter-chunk terms (diagonal and off-diagonal blocks)
Y = rearrange(Y_diag+Y_off, "b c l h p -> b (c l) h p")
return Y, final_state
if __name__ == "__main__":
batch = 2 # B
length = 72 # T
n_heads = 4 # H
dim_din = 512 # Din
d_head = dim_din // n_heads # P
d_state = 32 # N
block_len = 8 # Q
X = torch.randn(batch, length, n_heads, d_head)
A = -F.softplus(torch.randn(batch, length, n_heads))
B = torch.randn(batch, length, n_heads, d_state)
C = torch.randn(batch, length, n_heads, d_state)
Y, final_state = ssd(X, A, B, C, block_len=block_len)
print(Y.shape)
print(final_state.shape)
segsum は次のような操作になります。
>>> segsum(torch.arange(1, 5, dtype=float).reshape(1, 1, -1))
tensor([[[[0., -inf, -inf, -inf],
[2., 0., -inf, -inf],
[5., 3., 0., -inf],
[9., 7., 4., 0.]]]], dtype=torch.float64)
これは次のようなマトリクスを作っています。(
そしてこれを以下のように exp を操作する事で、1-SS行列を作成します。(
X, A, B, C = [rearrange(x, "b (c l) ... -> b c l ...", l=block_len) for x in (X, A, B, C)]
A = rearrange(A, "b c l h -> b h c l")
L = torch.exp(segsum(A))
Y_diag = torch.einsum("bclhn,bcshn,bhcls,bcshp->bclhp", C, B, L, X)
これは
Y = A_cumsum[:, :, :, -1:] - A_cumsum の操作は
となり、よって
>>> decay_states = torch.exp((A_cumsum[:, :, :, -1:] - A_cumsum))
>>> states = torch.einsum("bclhn,bhcl,bclhp->bchpn", B, decay_states, X)
>>> states.shape
torch.Size([2, 9, 4, 128, 32])
この操作は
A_cumsum[:, :, :, -1] について考えてみます。これは、各チャンクの最終配列です。batch と head 次元を無視すると、
つまり、Y=F.pad(A_cumsum[:, :, :, -1], (1, 0)) は
プログラムで書いてみると以下です。
>>> A = torch.arange(1, 10).to(float).reshape(-1, 3)
>>> A
tensor([[1., 2., 3.],
[4., 5., 6.],
[7., 8., 9.]], dtype=torch.float64)
>>> A_cumsum
tensor([[ 1., 3., 6.],
[ 4., 9., 15.],
[ 7., 15., 24.]], dtype=torch.float64)
>>> F.pad(A_cumsum[:, -1], (1, 0))
tensor([ 0., 6., 15., 24.], dtype=torch.float64)
>>> segsum(F.pad(A_cumsum[:, -1], (1, 0)))
tensor([[ 0., -inf, -inf, -inf],
[ 6., 0., -inf, -inf],
[21., 15., 0., -inf],
[45., 39., 24., 0.]], dtype=torch.float64)
です。もうお分かりかと思いますが、
>>> decay_chunk = torch.exp(segsum(F.pad(A_cumsum[:, :, :, -1], (1, 0))))
この操作は、Center Factor x Right Factor を表します。そのまま Left の流れも同様です。以上が、SSDの操作です。
このように、N-SSS の中に N-SSS が現れる事を利用して、Qのサイズでチャンク化する事で、計算量を減らせる仕組みとなっています。
疑問点
ただ、上記の計算ですが、少し間違っているかもしれません。というのも Center Factor で
となっている以上、例えば
私の式展開か疑似コードのどこかが間違っている可能性があります。ただ、ここでは計算の雰囲気がつかめれば問題ないとも思っています。
補足② SSMとAttention の比較
こちらも敢えて、補足と書きます。論文では当然重要な内容となっていますが、Mamba-2 の構造を単に理解するだけなら、避けて通れる内容にはなります。
申し訳ありませんが、こちらは機会があれば解説を試みたいと思います。これまで解説でだいぶ体力を使ってしまいました...。
Discussion