polarsが劇遅だった件
今流行りのpolarsを触ってみたらある条件を満たすと劇遅になった件について書きます。しかも一度遅くなるとセッションを再起動するまでずっと遅いです。なかなか気が付きにくいので記事にしておきます。
環境
幣計算環境は以下の通りです。他の条件で同じ現象が起こるかは未確認です。コードは置いておくので、polarsの利用を考えている方は事前に同様の問題が起きないか確かめておいた方が良いかもしれません。
OS: Windows 11 Pro (Version 22H2, OS build 22621.1265)
CPU: AMD Ryzen 7 5800X 8-Core Processor 3.80 GB
RAM: 16.0 GB
使用ライブラリのバージョン
python = "3.9.6"
pandas = "^1.4.3"
numpy = "^1.23.1"
polars = "^0.16.7"
pyarrow = "^11.0.0"
jupyter = "^1.0.0"
matplotlib = "^3.5.3"
seaborn = "^0.11.2"
データ準備
検証用のデータフレームを作成します。比較対象としてpandasのデータフレームも用意します。
import numpy as np
import pandas as pd
import polars as pl
arr = np.random.rand(10000, 5)
df_pl = pl.DataFrame(arr)
df_pd = pd.DataFrame(arr)
polarsは基本速い。
そもそもですが、polarsは大抵の場合pandasより高速に動作します。pandasの要素へのアクセス速度をベースラインとして、polarsで要素へアクセスするスピードを見てみましょう。
pandas(ベースライン)
%%timeit
df_pd.iloc[0, 0]
10.9 µs ± 194 ns per loop (mean ± std. dev. of 7 runs, 100,000 loops each)
polars
%%timeit
df_pl[0, 0]
1.79 µs ± 33.2 ns per loop (mean ± std. dev. of 7 runs, 1,000,000 loops each)
おー、約5倍くらいpolarsの方が速いです。これは嬉しい。
%%timeit
df_pl.select("column_1")[0]
39.8 µs ± 1.32 µs per loop (mean ± std. dev. of 7 runs, 10,000 loops each)
あれ、pandasより遅くなってしまいました。selectは積極的に使うのは避けた方が良さそうです。
%%timeit
df_pl.get_column("column_1")[0]
1.49 µs ± 19.2 ns per loop (mean ± std. dev. of 7 runs, 1,000,000 loops each)
get_columnでSeriesを取ってきてからスライスすると、要素へ直接アクセスするのと同等になりました。要素へ直接アクセスするのでも良いですが、可読性を考慮するとget_columnで列を指定してから目的の要素へアクセスするのが良いかもしれません。
# s = df_pl.get_column("column_1") を実行した後で実行
%%timeit
s[0]
693 ns ± 9.71 ns per loop (mean ± std. dev. of 7 runs, 1,000,000 loops each)
はっや。一旦Seriesを生成しておくことでより速くなりました。
polarsはある条件を満たすと劇遅になる。
いかがでしたか?polarsは基本速いです。pandasよりコーディング量は多少増えるものの、得られる高速化の恩恵は大きそうです。
ところが、一定サイズを超えるデータフレームから要素を1つずつ取り出してデータフレームを再構築すると劇遅になります(バグか仕様かは分かっていませんが、おそらくバグでしょう)。以下が劇遅polarsの再現実験コードです。
from timeit import timeit
from datetime import datetime, timedelta
import polars as pl
import pandas as pd
import numpy as np
import seaborn as sns
ns = [10000, 30000, 100000, 300000]
results = []
for n in ns:
print(f"n={n}")
data = dict(
time=[datetime.now() - (timedelta(minutes=15) * i) for i in range(n)],
value=np.random.rand(n),
)
df_pandas = pd.DataFrame(data)
df_polars = pl.DataFrame(data)
result = {}
result[f"pandas, before"] = timeit("df_pandas.iloc[0, 0]", globals=globals(), number=10000)
result[f"polars, before"] = timeit("df_polars[0, 0]", globals=globals(), number=10000)
# ただ値を取り出して新規にDataFrameを作るだけ
# これのせいで遅くなる
data2 = []
for i in range(df_polars.shape[0]):
time = df_polars[i, "time"]
value = df_polars[i, "value"]
data2.append(dict(time=time, value=value))
df_polars2 = pl.DataFrame(data2)
# 上段の"*before"と全く同じ処理
result[f"pandas, after"] = timeit("df_pandas.iloc[0, 0]", globals=globals(), number=10000)
result[f"polars, after"] = timeit("df_polars[0, 0]", globals=globals(), number=10000)
results.append(result)
上記コードは以下の処理を行っています。
- pandasとpolarsそれぞれでデータフレームを作成
- それぞれのデータフレームの要素
(0, 0) - polarsが劇遅になる処理を実行
- それぞれのデータフレームの要素
(0, 0)
用いたデータフレームはdatetime型とfloat64型のカラムを持ち、サイズは
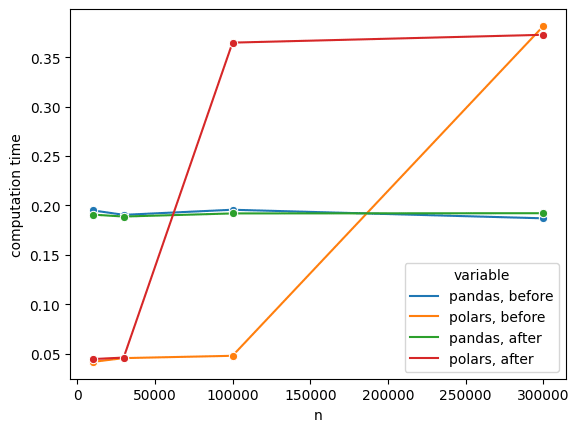
なんということでしょう。行数が polars, afterが劇遅になってしまいました。そればかりか、そのまま polars, before も劇遅になってしまっています。もしやと思い上記実験コードを2回連続で回すと2回目の結果が以下のようになりました。
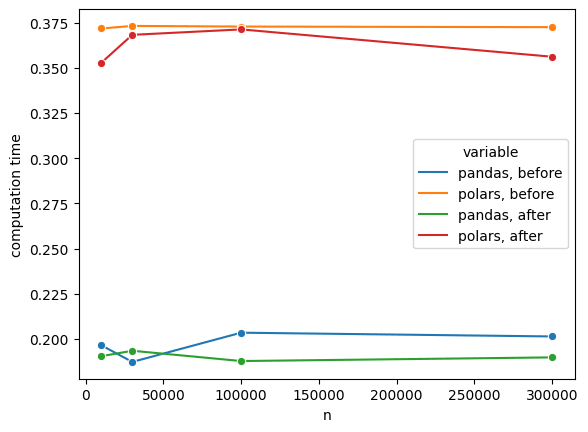
なんということでしょう。なぜか要素数に関係なくpolarsが劇遅になってしまいました。これでは到底使えたものではありません。仕様なのかバグなのかは不明ですが、挙動から考えるとバグでしょう。issueが上がっていなければpolars公式に報告する予定です。
対策
twitterにて、識者の方々からコメントを頂きました。御礼申し上げます。
一回遅くなるとオブジェクトを再生成しようがずっと遅いという謎挙動で、自分でもまだもやもやしていたので、コメントにも基づいて追加検証しました。
頂いたコメントで共通していたのは
- 並列化ができていないのではないか
- 順序が保てない(ソートフラグが外れた)のではないか
とのことでした。今回の検証で行っていたのは、forループで要素
from timeit import timeit
from datetime import datetime, timedelta
import polars as pl
import pandas as pd
import numpy as np
import seaborn as sns
ns = [10000, 30000, 100000, 300000, 1000000]
results = []
for n in ns:
print(f"n={n}")
data = dict(
time=[datetime.now() - (timedelta(minutes=15) * i) for i in range(n)],
value=np.random.rand(n),
)
df_pandas = pd.DataFrame(data)
df_polars = pl.DataFrame(data)
arr = df_polars.to_numpy()
result = {}
result[f"pandas, before"] = timeit("df_pandas.iloc[0, 0]", globals=globals(), number=10000)
result[f"polars, before"] = timeit("df_polars[0, 0]", globals=globals(), number=10000)
# 変更ここから=======================================================================
# numpyで先にメモリを確保しておく
dt_np = np.empty(df_polars.shape[0], dtype=datetime)
value = np.empty(df_polars.shape[0], dtype=np.float64)
for i in range(df_polars.shape[0]):
dt_np[i] = df_polars[i, "time"]
value[i] = df_polars[i, "value"]
df_polars2 = pl.DataFrame([pl.Series("time", dt_np), pl.Series("value", value)])
# 変更ここまで=======================================================================
# 上段の"*before"と全く同じ処理
result[f"pandas, after"] = timeit("df_pandas.iloc[0, 0]", globals=globals(), number=10000)
result[f"polars, after"] = timeit("df_polars[0, 0]", globals=globals(), number=10000)
results.append(result)
念のため、生成するデータフレームの行数を
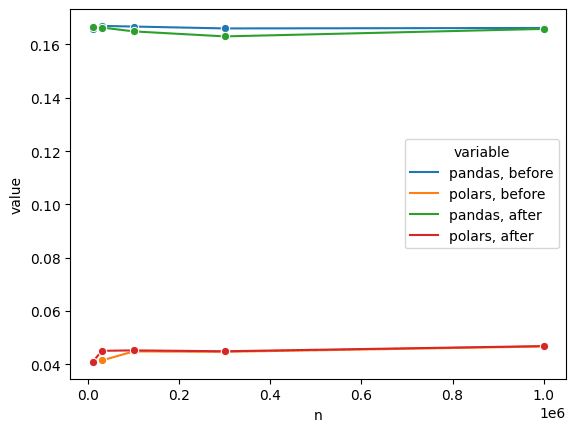
polarsの圧勝です。最初に検証した1要素へのアクセス速度は4,5倍くらいpolarsの方が速かったので、大体期待通りです。これで安心です。
おまけ
ループで要素に逐次アクセスする場合、実はnumpyが最速です。以下の結果をご覧ください。
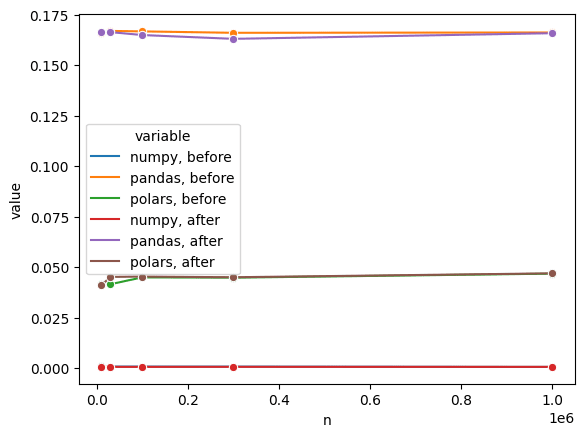
numpyが圧倒的ですね(polarsのさらに100倍程度)。要素へ次々とアクセスする必要がある時はnumpyにしてから計算した方が良いでしょう。
まとめ
- polarsを使ってデータフレームを再構築するときは気をつけましょう。
- 結局numpyが速い。
Discussion
df_polars2 を作ると df_polars に対するアクセスが遅くなる件は、最近のバージョンでは直っているようです。(polars 0.18.7 で確認)
そうなのですね!
今週末には記事に追記させて頂きます。
お知らせありがとうございます!