NASAの時系列データtelemanomの異常値を見てみる(少しだけVAEで異常検知)
本記事の目的と背景
前回は、シンプルな時系列データにVAE(Variational AutoEncoder)を適用し、異常検知と生成がうまくいくことを確認しました。
今回はさらに一歩進めるために、異常検知の候補としてNASAの実際の宇宙機テレメトリデータを選びVAEで異常検知にチャレンジしました!
前回のおさらい
前回の記事では、VAEを使った異常検知の練習をしました。モデルに学習させたのは、自作したシンプルな時系列データセットで、その正常データにスパイクのようなノイズを載せたデータの検知をさせました。結果、異常検知をすることが出来ました。
その際、学習させたモデルは特にチューニングせずに、ドメイン知識も入出力のサイズ程度しか気にせずに異常検知ができました。そのため、入出力のサイズさえ変えれば実データに対しても異常検知が出来るのではないかと思い、挑戦してみることにしました。
Telemanom データセットとは?
時系列のデータセットとして、NASA提供のTelemanomを選びました。
選定理由は次の通りです:
- 実際の宇宙機(SMAP、Curiosity Rover)のリアルなセンサーデータ
- 異常区間のラベルが明示されている
- データが正規化済みで前処理が不要
データセットはKaggleのNASA Anomaly Detection Dataset SMAP & MSLからダウンロードします。
ダウンロードしてきた dataフォルダ と labeled_anomalies.csv を以下のような構成にします。
.
├───data
├───labeled_anomalies.csv
└───main.ipynb
まずは異常データについての情報がある labeled_anomalies.csv を見てみます。
import pandas as pd
label = pd.read_csv('labeled_anomalies.csv')
label
| chan_id | spacecraft | anomaly_sequences | class | num_values |
|---|---|---|---|---|
| P-1 | SMAP | [[2149, 2349], [4536, 4844], [3539, 3779]] | [contextual, contextual, contextual] | 8505 |
| S-1 | SMAP | [[5300, 5747]] | [point] | 7331 |
| E-1 | SMAP | [[5000, 5030], [5610, 6086]] | [contextual, contextual] | 8516 |
| E-2 | SMAP | [[5598, 6995]] | [point] | 8532 |
| E-3 | SMAP | [[5094, 8306]] | [point] | 8307 |
上記のカラム名は以下のような意味だと思われます。
| カラム名 | 説明 |
|---|---|
| chan_id | チャンネルID。各センサーデータ系列の識別子。先頭文字で種類が分かる(例:P = 電源系) |
| spacecraft | データを収集した宇宙機の名前(例:SMAP や MSL) |
| anomaly_sequences | 異常が発生した区間のリスト。各要素は [開始インデックス, 終了インデックス] 形式 |
| class | 各異常区間の種類。point(瞬間的な異常)または contextual(文脈的異常) |
| num_values | そのチャンネルのテストデータに含まれる総データ数(サンプル数) |
まずは一行目の P-1 をプロットして異常値の範囲を赤く強調します。
import matplotlib.pyplot as plt
import numpy as np
import ast
# 対象のID
chan_id: str = 'P-1'
# 対象データの読み込み
data = np.load(f'data/test/{chan_id}.npy')
# 異常値の範囲を取得
anomaly_sequences: list = ast.literal_eval(label[label['chan_id'] == chan_id]['anomaly_sequences'].values[0])
# データをプロット
fig = plt.figure(figsize=(12, 8))
ax = fig.add_subplot(111)
start: int = 0
end: int = data.shape[0]
for seq in anomaly_sequences:
start_seq: int = seq[0]
end_seq: int = seq[1]
ax.axvspan(start_seq, end_seq, color="red", alpha=0.2)
x = np.linspace(start, end, end - start)
ax.plot(x,data[start:end, 0])
plt.show()
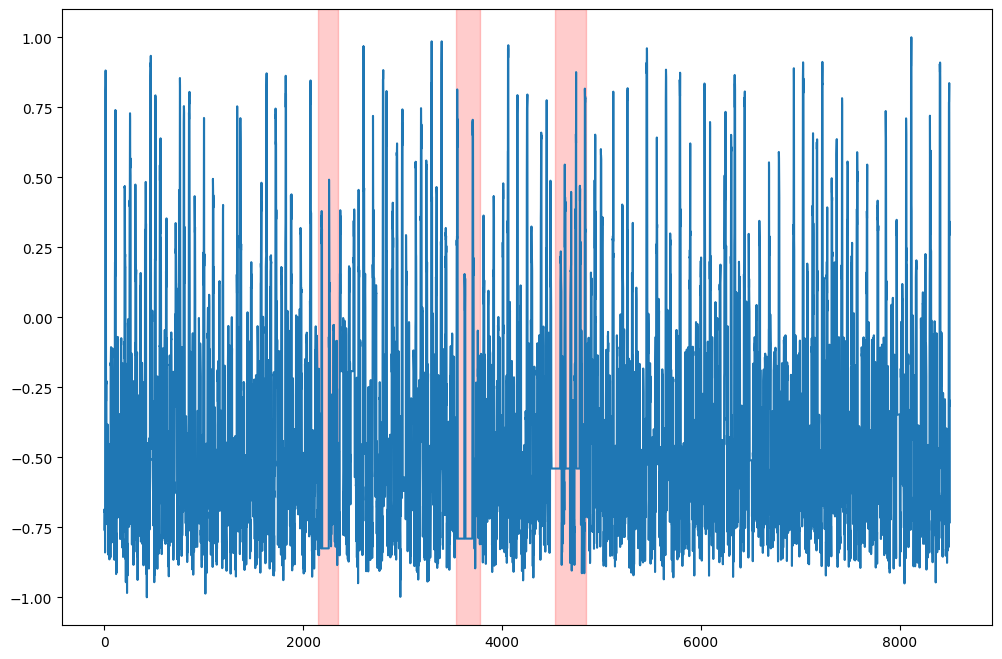
ざっくり見た印象では、異常値の範囲は他と比べて振動してなさそうですね。全体では分かりづらいので異常値に注目してみます。
# データをプロット
fig = plt.figure(figsize=(12, 8))
ax_len: int = len(anomaly_sequences)
for i, seq in enumerate(anomaly_sequences):
start_seq: int = seq[0]
end_seq: int = seq[1]
start = start_seq - 200
end = end_seq + 200
ax = fig.add_subplot(ax_len, 1, i + 1)
ax.set_ylim(-1, 1)
ax.axvspan(start_seq, end_seq, color="red", alpha=0.2)
x = np.linspace(start, end, end - start)
ax.plot(x,data[start:end, 0])

注目すると確かに平たんになっていますね。いきなり電源が一定になるのは考えづらいのでデータが飛んだんでしょうか?
コラム:SMAPとは?
少し休憩もかねて、このデータを提供している SMAP について調べてみます。RESTECというサイトが日本語で概要をまとめてくれているのでわかりやすかったです。
そもそもSMAPは Soil Moisture Active/Passive Mission の略称で地球の土壌水分を調査しているのですね。
実際に得られたデータをプロットしたものを引用させていただきます。この図は地球の土壌の水分を表しており、各色は以下のような構成になっています。
- 青色:湿っている場所
- 黄色:乾燥している場所
- 白色:凍っている場所
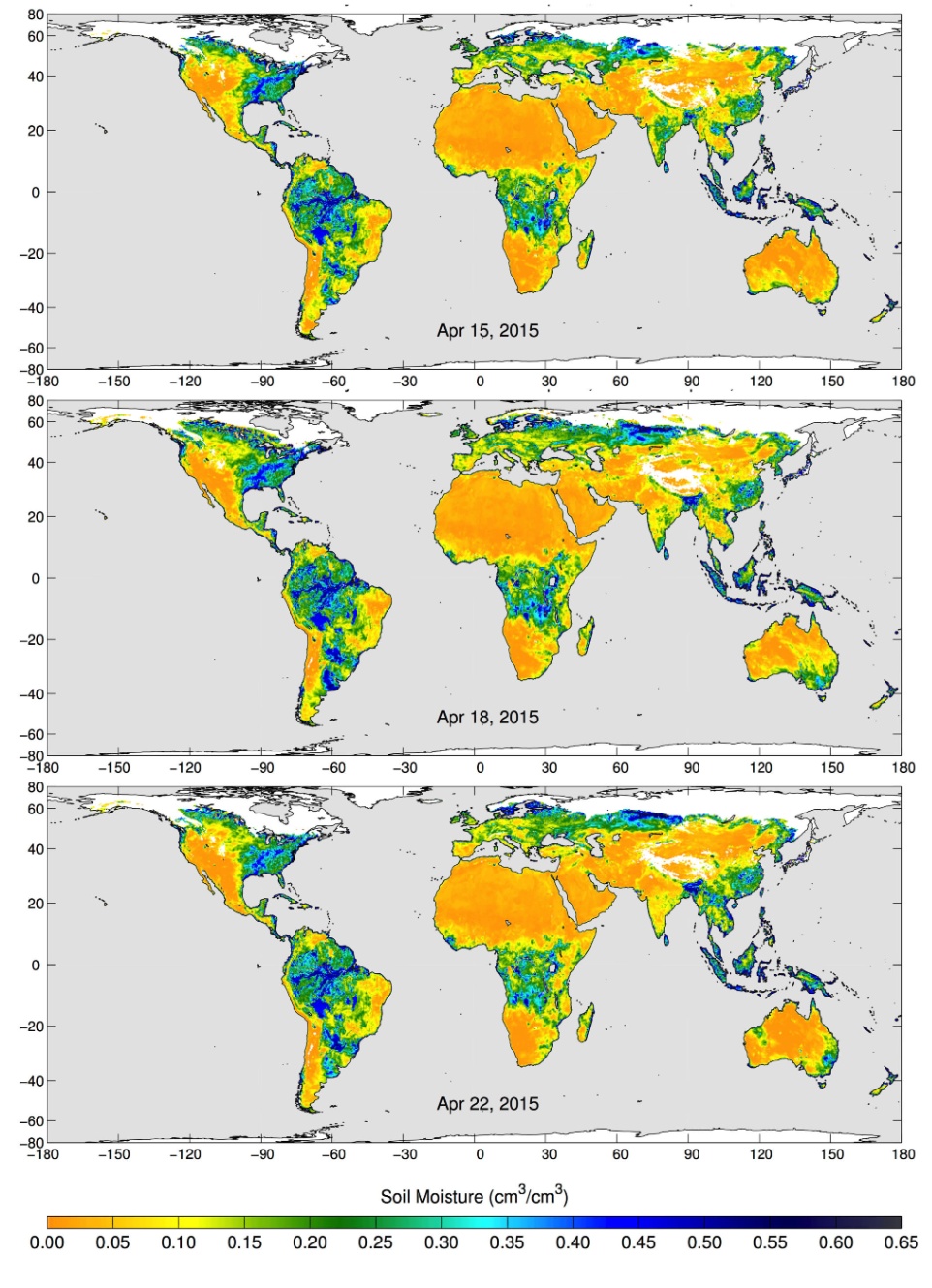
SMAP's Radiometer Captures Views of Global Soil Moisture 引用元:NASA/JPL-Caltech/GSFC
異常値の可視化
さあ、いよいよtelemanomの異常検知をしていきましょう!モデルに学習させるデータは dataフォルダ の中にある trainフォルダ で正常データが入っています。と異常値が入っている testフォルダ の2つがあります。
data
├───2018-05-19_15.00.10
├───test
└───train
今回はP系のチャネルのみtrainとtestをプロットしてみます。グラフは、青線がtrain、赤線がtest、赤い領域がcontextual、緑の領域がpointです。
def get_datas(folder_path: str) -> dict:
datas = {}
for file_name in os.listdir(folder_path):
if file_name.startswith("P") and file_name.endswith(".npy"):
file_path = os.path.join(folder_path, file_name)
data = np.load(file_path)
datas[file_name] = data
return datas
# データ読み込み
train = get_datas("data/train/")
test = get_datas("data/test/")
# データをひとまとめにする
plot_data: pd.DataFrame = pd.DataFrame(columns=["value", "train_end", "anomalys"])
for file_name in train.keys():
chan_id: str = file_name.replace(".npy", "")
# 値を1次元化して連結
train_values = train[file_name][:, 0].tolist()
test_values = test[file_name][:, 0].tolist()
all_values = train_values + test_values
# 境界インデックス
train_end = len(train_values) - 1
# ラベルから異常区間と種別を抽出
row = label[label['chan_id'] == chan_id]
sequences = ast.literal_eval(row['anomaly_sequences'].values[0])
raw_classes = row['class'].values[0] # -> "[contextual, contextual, contextual]"
classes = [s.strip() for s in raw_classes.strip("[]").split(",")]
anomaly_df = pd.DataFrame({
"start": [s[0] for s in sequences],
"end": [s[1] for s in sequences],
"class": classes
})
# オフセット追加:start, end に train_end + 1 を加算
anomaly_df["start"] = anomaly_df["start"].astype(int) + (train_end + 1)
anomaly_df["end"] = anomaly_df["end"].astype(int) + (train_end + 1)
# plot_data に登録
plot_data.loc[chan_id] = {
"value": all_values,
"train_end": train_end,
"anomalys": anomaly_df
}
fig, axes = plt.subplots(len(plot_data), 1, figsize=(12, 3 * len(plot_data)), sharex=False)
if len(plot_data) == 1:
axes = [axes] # 1つだけのときはリスト化
for i, (chan_id, row) in enumerate(plot_data.iterrows()):
values = row["value"]
train_end = row["train_end"]
anomaly_df = row["anomalys"]
x = range(len(values))
axes[i].plot(x[:train_end + 1], values[:train_end + 1], color='blue', label='train')
axes[i].plot(x[train_end + 1:], values[train_end + 1:], color='red', label='test')
# 異常区間のハイライト
for _, anomaly in anomaly_df.iterrows():
color = 'green' if anomaly["class"].strip() == "point" else 'red'
alpha = 0.3 if anomaly["class"].strip() == "contextual" else 0.6
axes[i].axvspan(anomaly["start"], anomaly["end"], color=color, alpha=alpha)
axes[i].set_title(chan_id)
axes[i].set_ylabel("Telemetry Value")
axes[i].set_ylim(-1.2, 1.2)
axes[i].grid(True)
axes[i].legend()
plt.xlabel("Time Step")
plt.tight_layout()
plt.show()
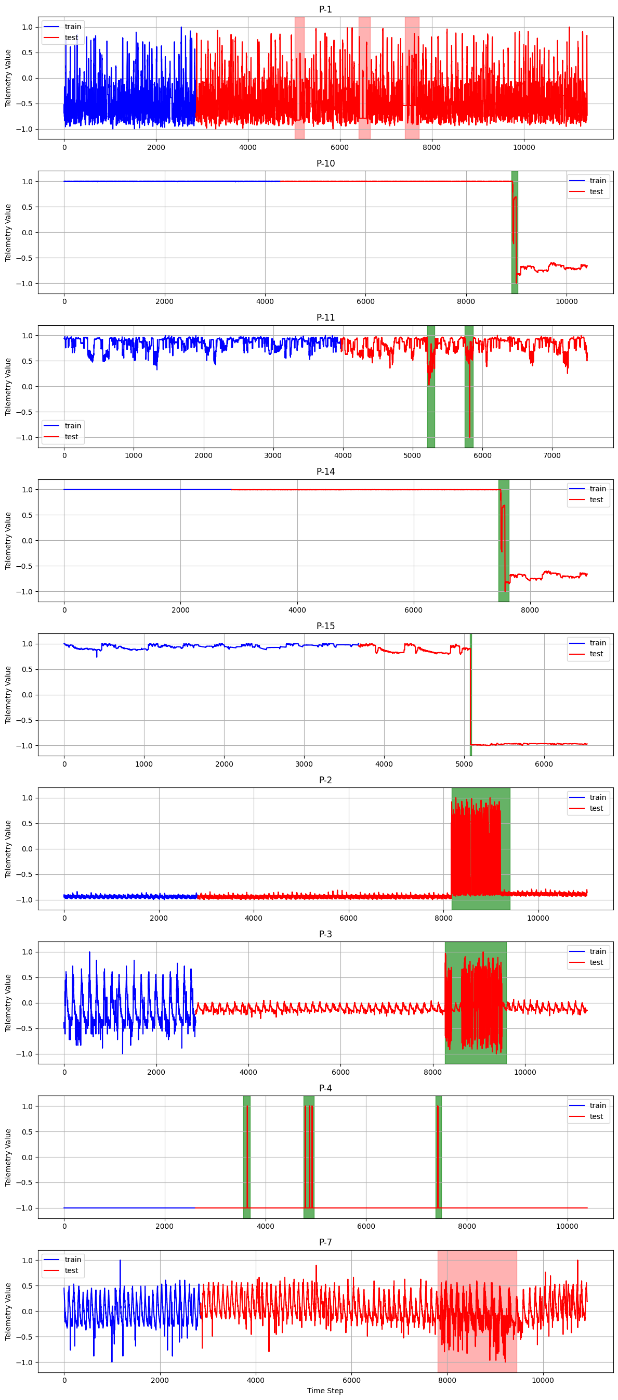
グラフをみるとチャネルによってだいぶ波形が違いますね。また、 P-1, P-7 以外はVAEを使わずとも閾値設定で異常検知が容易そうなので、対象をこの2チャネルに絞ります(ラベルにも文脈的な異常であると示唆されてますしね)。
VAEモデル設計と実装
いよいよVAEに学習させて異常検知していきます!入出力の次元は前回と同じ50にするので、データの末尾は切り捨てます。
class ChunkedDataset(Dataset):
def __init__(self, data, chunk_size=50):
self._chunk_size = chunk_size
# 切り捨てる(例:2872 → 2850まで使用)
usable_len = (len(data) // chunk_size) * chunk_size
self._data = data[:usable_len]
def __len__(self):
return len(self._data) // self._chunk_size
def __getitem__(self, idx):
start = idx * self._chunk_size
end = start + self._chunk_size
chunk = self._data[start:end]
return torch.tensor(chunk, dtype=torch.float32)
p1_train = ChunkedDataset(train['P-1.npy'])
p1_test = ChunkedDataset(test['P-1.npy'])
p7_train = ChunkedDataset(train['P-7.npy'])
p7_test = ChunkedDataset(test['P-7.npy'])
データを分割出来たら前回のVAEを引用して学習させます。
class Encoder(nn.Module):
def __init__(self, input_dim, hidden_dim, latent_dim):
super().__init__()
self._linear = nn.Linear(input_dim, hidden_dim)
self._mu = nn.Linear(hidden_dim, latent_dim)
self._logvar = nn.Linear(hidden_dim, latent_dim)
def forward(self, x):
h = self._linear(x)
h = F.relu(h)
mu = self._mu(h)
logvar = self._logvar(h)
return mu, logvar
class Decoder(nn.Module):
def __init__(self, latent_dim, hidden_dim, output_dim):
super().__init__()
self._linear = nn.Linear(latent_dim, hidden_dim)
self._output = nn.Linear(hidden_dim, output_dim)
def forward(self, z):
h = self._linear(z)
h = F.relu(h)
x_hat = self._output(h)
return x_hat
def reparameterize(mu, logvar):
sigma = torch.exp(0.5 * logvar)
epsilon = torch.randn_like(sigma)
z = mu + epsilon * sigma
return z
class VAE(nn.Module):
def __init__(self, input_dim, hidden_dim, latent_dim):
super().__init__()
self._encoder = Encoder(input_dim, hidden_dim, latent_dim)
self._decoder = Decoder(latent_dim, hidden_dim, input_dim)
def forward(self, x):
mu, logvar = self._encoder(x)
z = reparameterize(mu, logvar)
x_hat = self._decoder(z)
return x_hat, mu, logvar
def get_loss(self, x):
x_hat, mu, logvar = self.forward(x)
recon_loss = F.mse_loss(x_hat, x, reduction='sum')
kl_loss = - torch.sum(1 + logvar - mu ** 2 - logvar.exp())
total_loss = recon_loss + kl_loss
return total_loss, recon_loss, kl_loss
vae = VAE(input_dim=50, hidden_dim=32, latent_dim=10)
optimizer = torch.optim.Adam(vae.parameters(), lr=1e-4)
epochs = 1024
batch_size = 32
train_loader = DataLoader(
p1_train,
batch_size=batch_size,
shuffle=False)
train_losses = []
for epoch in range(epochs):
vae.train()
total_loss = 0
for batch in train_loader:
x = batch[0]
loss, recon_loss, kl_loss = vae.get_loss(x)
optimizer.zero_grad()
loss.backward()
optimizer.step()
total_loss += loss.item()
train_losses.append(total_loss)
if epoch % 10 == 0:
print(f"Epoch {epoch} | Loss: {total_loss:.2f}")
# プロット
plt.plot(train_losses)
plt.title("Training Loss")
plt.xlabel("Epoch")
plt.ylabel("Loss")
plt.grid(True)
plt.show()
学習と評価
プロットされた損失関数をみるとしては減少傾向ではありますが、振動していますね、、、

最後に、VAEに訓練データとテストデータをモデルに入力し生成してみます。
vae.eval()
train_originals = []
train_reconstructions = []
test_originals = []
test_reconstructions = []
train_loader = DataLoader(p1_train, batch_size=1, shuffle=False)
test_loader = DataLoader(p1_test, batch_size=1, shuffle=False)
with torch.no_grad():
for x in train_loader:
x = x.squeeze(0) # shape: (50,)
x_hat, _, _ = vae(x)
train_originals.append(x.numpy())
train_reconstructions.append(x_hat.numpy())
for x in test_loader:
x = x.squeeze(0) # shape: (50,)
x_hat, _, _ = vae(x)
test_originals.append(x.numpy())
test_reconstructions.append(x_hat.numpy())
# 1. 全チャンクを縦に結合(flatten)
plot_data: pd.DataFrame = pd.DataFrame({
'id': [1, 7],
'original': [
np.concatenate(train_originals),
np.concatenate(test_originals)],
'reconstruction': [
np.concatenate(train_reconstructions),
np.concatenate(test_reconstructions)]
})
# 2. 描画
fig, axes = plt.subplots(nrows=2, ncols=2, figsize=(14, 6), sharex=False)
for i, row in plot_data.iterrows():
original = row['original']
recon = row['reconstruction']
error = np.abs(original - recon) # 絶対誤差
# --- Original vs Reconstruction ---
axes[0, i].plot(original, label='Original', color='blue', alpha=0.5)
axes[0, i].plot(recon, label='Reconstruction', color='red', alpha=0.5)
axes[0, i].set_title(f"id={row['id']} Original vs Reconstruction")
axes[0, i].legend()
axes[0, i].set_ylim(-1.2, 1.2)
axes[0, i].grid(True)
# --- Reconstruction Error ---
axes[1, i].plot(error, label='Abs Error', color='purple', alpha=0.5)
axes[1, i].set_title(f"id={row['id']} Reconstruction Error")
axes[1, i].set_ylim(-0.2, 2.2)
axes[1, i].grid(True)
plt.tight_layout()
plt.show()
for i, row in plot_data.iterrows():
error = np.abs(row['original'] - row['reconstruction'])
print(f"▼ id = {row['id']}")
print(f" Max Error : {error.max():.6f}")
print(f" Min Error : {error.min():.6f}")
print(f" Mean Error : {error.mean():.6f}")
print(f" Std (σ) : {error.std():.6f}")
print("-" * 40)
プロットされた波形を見ると訓練データのピークをうまく再現できていないですね。閾値をきめるなら最大誤差でしょうか、、、
異常検知をするならもう少しデータを見る必要がありそうですね。
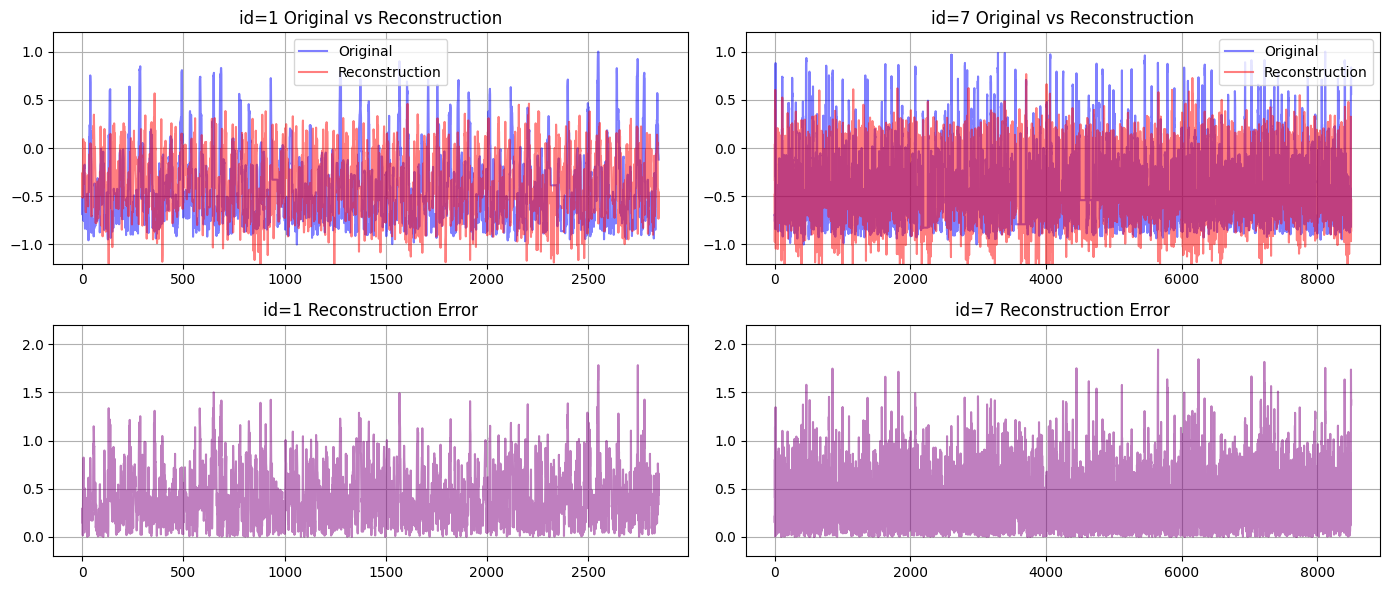
▼ id = 1
Max Error : 1.781561
Min Error : 0.000207
Mean Error : 0.411541
Std (σ) : 0.299734
----------------------------------------
▼ id = 7
Max Error : 1.943274
Min Error : 0.000011
Mean Error : 0.407376
Std (σ) : 0.300571
----------------------------------------
まとめと今後の展望
今回は、VAEを用いた異常検知の応用編として、NASAの実データ(SMAP衛星の電源系)に取り組みました。時系列に対してシンプルなVAEを適用し、再構成誤差を可視化することで異常区間の兆候を捉えるアプローチを試しました。
今後は、異常検知、異常検知個所の特定まで出来るようにデータの分析とモデルのチューニングをしていこうと考えています!
Discussion