Rustを使って機械学習アルゴリズムを実装しPythonから呼び出す: k-meansクラスタリング
概要
最近Rustの勉強を始めたのですが、以下の2点について実際どうなんだろうと思ったので、Rustを使って機械学習アルゴリズムを実装してPythonから呼び出すということを試してみました。
- Pythonで実装すると遅い処理をRustで高速化できるか
- 実装したアルゴリズムを簡単に並列化することが出来るか
この記事では、どのように実装したかについて説明した後、Python実装とRust実装の速度を実験により比較します。
ちなみに、前の記事の続きになりますが、この記事だけで完結するように書いてはいます。
興味がありましたら前の記事も読んでみてください。
記事を読むにあたり注意してもらいたいこと
Rustに関しては勉強中なので、内容の正確性についてはあまり自信がありません。
あくまでも「こうやったら出来ました」という事例紹介だと思って読んで頂けるとありがたいです。
k-means クラスタリング
機械学習アルゴリズムとして何を実装するかですが、今回はk-meansクラスタリングを実装することにしました。
k-meansを選んだ理由は、端的に言うと自分が知っている機械学習アルゴリズムの中では最も実装が簡単な部類だと思うからです。
Rustでまともにプログラミングすること自体が初めてなので、実装が難しくて挫折するのは避けたいという意図で簡単なものを選びました。
NumPy を使った k-means の実装
Rust の実装と比較する必要もありますし、ひとまず Python で k-means クラスタリングを実装してみましょう。
"""
[ k-means アルゴリズム ]
1. 各データ点をランダムにk個のクラスタに割り当てて、その平均ベクトルを中心点とする
2. 以下のステップを繰り返す
- 各データ点がどの中心点に近いかを計算して、最も近いクラスタに割り当てる
- 新しいクラスタの割り当てを用いて、各クラスタの中心点を計算しなおす
"""
import numpy as np
def kmeans(X, k, num_steps=100):
def calc_means(X, k, cluster_indices):
return np.array([X[cluster_indices == i].mean(axis=0) for i in range(k)])
# init clusters
N, _ = X.shape
cluster_indices = np.random.randint(0, k, size=(N,))
means = calc_means(X, k, cluster_indices)
# training
for _ in range(num_steps):
cluster_indices = ((np.expand_dims(X, 1) - means) ** 2).sum(axis=2).argmin(axis=1)
means = calc_means(X, k, cluster_indices)
return cluster_indices, means
# 使い方
X = np.random.random(size=(1000, 10))
cluster_indices, means = kmeans(X, 5)
簡単な手法なのであまり説明することもないですが、個人的なこだわりポイントは「各データ点がどの中心点に近いかを計算して、最も近いクラスタに割り当てる」のところです。
コードで言うと
cluster_indices = ((np.expand_dims(X, 1) - means) ** 2).sum(axis=2).argmin(axis=1)
の部分ですね。
素朴に実装すると (データ点数 x クラスタ数) の二重ループを書きたくなるところですが、NumPyのブロードキャストを上手く使ってこの処理が一発で終わるようになっています。[1]
scikit-learn の kmeans
そもそも k-means はかなりメジャーな手法なので、自分で実装しなくても使えるライブラリが幾らでもあります。
例えば、scikit-learn を使えば以下のように簡単に k-means クラスタリングを行うことができます。
from sklearn.cluster import KMeans
def sklearn_kmeans(X, k, num_steps=100):
# 自分で作ったものと同じような処理になるようにパラメータを設定
clf = KMeans(n_clusters=k, max_iter=num_steps, init='random', n_init=1, tol=0.0)
cluster_indices = clf.fit_predict(X)
means = clf.cluster_centers_
return cluster_indices, means
準備: 実験用Crateの作成
Rustでk-meansを実装するために、まずは実験用のCrateを作成します。
Crate作成の手順は以下の通りです。
- kmeansという名前でライブラリクレートを作成します。
cargo new --lib kmeans
- 次に、Cargo.tomlを以下のように編集します。
[package]
name = "kmeans"
version = "0.1.0"
edition = "2021"
[lib]
crate-type = ["cdylib"]
[dependencies]
rand = "0.8.5"
rayon = "1.6"
Rust で k-means を実装する
比較対象の Python 実装が出来たところで、早速 Rust でも k-means を実装してみます。
以下のコードが完成した Rust 版の実装です。基本的には素直に実装しただけという感じで注目すべき点は多くありませんが、苦労した点もあるので簡単に説明します。
use rand::Rng;
/// 各クラスタの平均ベクトルを求める
fn calc_means(data: &Vec<Vec<f64>>, k: usize, cluster_indices: &[usize]) -> Vec<Vec<f64>> {
let n = data.len();
let d = data[0].len();
let mut means = vec![vec![0.0; d]; k];
let mut counts = vec![0; k];
for i in 0..n {
let j = cluster_indices[i];
for l in 0..d {
means[j][l] += data[i][l];
}
counts[j] += 1;
}
for j in 0..k {
if counts[j] == 0 {
continue;
}
for l in 0..d {
means[j][l] /= counts[j] as f64;
}
}
means
}
/// 2つのベクトル間のユークリッド距離の2乗を計算する
fn calc_dist(x: &[f64], y: &[f64]) -> f64 {
x.iter()
.zip(y.iter())
.map(|(xi, yi)| (xi - yi).powi(2))
.sum()
}
/// ベクトルxに最も近い平均ベクトルのインデックスを取得する
fn get_min_index(x: &[f64], means: &[Vec<f64>]) -> usize {
let mut min_dist = std::f64::MAX;
let mut min_index = 0;
for (j, m) in means.iter().enumerate() {
let dist = calc_dist(x, m);
if dist < min_dist {
min_dist = dist;
min_index = j;
}
}
min_index
}
/// kmeans algorithm
fn _kmeans(data: Vec<Vec<f64>>, k: usize, num_steps: usize) -> (Vec<usize>, Vec<Vec<f64>>) {
// init clusters
let n = data.len();
let mut rng = rand::thread_rng();
let mut cluster_indices: Vec<usize> = (0..n).map(|_| rng.gen_range(0..k)).collect();
let mut means = calc_means(&data, k, &cluster_indices);
// traininig
for _ in 0..num_steps {
cluster_indices = data.iter().map(|x| get_min_index(x, &means)).collect();
means = calc_means(&data, k, &cluster_indices);
}
(cluster_indices, means)
}
/// kmeansの結果をPythonに返すための構造体
#[repr(C)]
pub struct KmeansResult {
pub cluster_indices: *const usize,
pub means: *const f64,
}
/// # Safety
/// Pythonからデータを受け取ってkmeansを実行する
#[no_mangle]
pub unsafe extern "C" fn kmeans(
array: *const f64,
rows: usize,
cols: usize,
k: usize,
num_steps: usize,
) -> *const KmeansResult {
let a = unsafe { std::slice::from_raw_parts(array, rows * cols) };
let data: Vec<Vec<f64>> = a.chunks(cols).map(|x| x.to_vec()).collect();
let (cluster_indices, means) = _kmeans(data, k, num_steps);
let means = means.concat();
let kmeans_result = KmeansResult {
cluster_indices: cluster_indices.as_ptr(),
means: means.as_ptr(),
};
std::mem::forget(cluster_indices);
std::mem::forget(means);
Box::into_raw(Box::new(kmeans_result))
}
/// # Safety
/// KmeansResultのメモリを解放する
#[no_mangle]
pub unsafe extern "C" fn free_kmeans_result(kmeans_result: *mut KmeansResult) {
let _ = Box::from_raw(kmeans_result);
}
/// # Safety
/// *const f64 のメモリを解放する
#[no_mangle]
pub unsafe extern "C" fn free_f64_array(array: *mut f64, len: usize) {
let _ = std::slice::from_raw_parts(array, len);
}
/// # Safety
/// *const usize のメモリを解放する
#[no_mangle]
pub unsafe extern "C" fn free_usize_array(array: *mut usize, len: usize) {
let _ = std::slice::from_raw_parts(array, len);
}
まず肝心の kmeans の本体(?)の部分ですが、下のように Python 版とほぼ同じような見た目の実装になっています。なので、Python版と比べて特別に実装が難しいと感じることもありませんでした。
途中で使われている calc_means や get_min_index なども自分で実装したものなのですが、基本的には for とかを使って愚直に実装しただけという感じなので、そんなに苦労もしていません。
/// kmeans algorithm
fn _kmeans(data: Vec<Vec<f64>>, k: usize, num_steps: usize) -> (Vec<usize>, Vec<Vec<f64>>) {
// init clusters
let n = data.len();
let mut rng = rand::thread_rng();
let mut cluster_indices: Vec<usize> = (0..n).map(|_| rng.gen_range(0..k)).collect();
let mut means = calc_means(&data, k, &cluster_indices);
// traininig
for _ in 0..num_steps {
cluster_indices = data.iter().map(|x| get_min_index(x, &means)).collect();
means = calc_means(&data, k, &cluster_indices);
}
(cluster_indices, means)
}
逆に、苦労した点はどこかと言うと、以下に示す Pythonとのデータの受け渡しの部分です。
とりあえずどのような方針でデータの受け渡しを行うかを決めなければいけないのですが、この記事では下のような方針で実装しました。
- Pythonとのデータの受け渡しはctypesを使って行う。
- PythonからRustにデータを渡すとき: NumPyの配列はdouble型のポインタとして渡す。
- RustからPythonにデータを渡すとき:
cluster_indicesとmeansという2つのVecをKmeansResult構造体に詰めて Python 側に返す。
ちゃんと動かすために色々と学ばなければいけないことがあったので苦労しました。
- Pythonから呼び出したい関数の頭には
#[no_mangle]とpub unsafe extern "C"を付ける。 - 引数や返り値を配列や構造体にする場合は
*const Tや*mut Tのような生ポインタを使う。 - 生ポインタから値を取り出すのはRust的にはunsafeな処理なので、
unsafeブロックで囲む必要がある。 - 構造体の定義のところに
#[repr(C)]を付けると、構造体のメモリレイアウトが C言語の構造体と同じになる。 -
Vecをas_ptrで生ポインタに変換した上で、std::mem::forgetで所有権を放棄する。 - 構造体を出力する前に
Box::newでヒープ領域にデータを置いてから、Box::into_rawで生ポインタに変換しつつ所有権を放棄する。 - メモリを解放する処理 (
free_kmeans_result,free_f64_array,free_usize_array) を用意して、使い終わったデータはPythonから再度戻してもらいメモリを解放する。[2]
/// kmeansの結果をPythonに返すための構造体
#[repr(C)]
pub struct KmeansResult {
pub cluster_indices: *const usize,
pub means: *const f64,
}
/// # Safety
/// Pythonからデータを受け取ってkmeansを実行する
#[no_mangle]
pub unsafe extern "C" fn kmeans(
array: *const f64,
rows: usize,
cols: usize,
k: usize,
num_steps: usize,
) -> *const KmeansResult {
let a = unsafe { std::slice::from_raw_parts(array, rows * cols) };
let data: Vec<Vec<f64>> = a.chunks(cols).map(|x| x.to_vec()).collect();
let (cluster_indices, means) = _kmeans(data, k, num_steps);
let means = means.concat();
let kmeans_result = KmeansResult {
cluster_indices: cluster_indices.as_ptr(),
means: means.as_ptr(),
};
std::mem::forget(cluster_indices);
std::mem::forget(means);
Box::into_raw(Box::new(kmeans_result))
}
/// # Safety
/// KmeansResultのメモリを解放する
#[no_mangle]
pub unsafe extern "C" fn free_kmeans_result(kmeans_result: *mut KmeansResult) {
let _ = Box::from_raw(kmeans_result);
}
/// # Safety
/// *const f64 のメモリを解放する
#[no_mangle]
pub unsafe extern "C" fn free_f64_array(array: *mut f64, len: usize) {
let _ = std::slice::from_raw_parts(array, len);
}
/// # Safety
/// *const usize のメモリを解放する
#[no_mangle]
pub unsafe extern "C" fn free_usize_array(array: *mut usize, len: usize) {
let _ = std::slice::from_raw_parts(array, len);
}
...若干の不安はありますが、とりあえず Rust 側での k-means の実装は完了しました。
コードが書けたらリリースモードでビルドして、共有ライブラリを作成することでPythonからも呼び出せるようになります。
(今回は速度の比較をするのが目的なのでリリースモードでビルドする必要があります。)
cargo build --release
OSによって出力されるファイル名が異なるので、環境に応じて以下のように読み替えてください。
- Windowsの場合
target/release/kmeans.dll - MacOSの場合
target/release/libkmeans.dylib - Linuxの場合
target/release/libkmeans.so
ちなみに今回は Windows で実装しているので、kmeans.dll というファイル名になります。
Rustで実装した k-means を Python から呼び出す
以下のようなコードを書くとRustで実装したk-meansをPythonから呼び出すことができます。
正直に言ってしまうとctypesについてもそんなに詳しくないのでかなり手探りで実装していますが、以下のような点がポイントです。
- doubleのポインタを表すには、
ctypes.POINTER(ctypes.c_double)とする。 - 構造体を定義するには
ctypes.Structureを継承したクラスを定義して、その中に_fields_というクラス変数を定義する。 - NumPyの配列
Xをdoubleのポインタに変換するにはX.ctypes.data_as(ctypes.POINTER(ctypes.c_double))とする。 - Rust側から返ってきた構造体の中身のデータには、
contentsという属性を使ってアクセスできる。(ret.contenst.cluster_indicesなど) - Rust側から返ってきたポインタをNumPyの配列に変換するには
np.ctypeslib.as_arrayを使う。(np.ctypeslib.as_array(ret.contents.means, shape=(k, d))など) - メモリリークを防ぐために
.copy()でコピーした配列を使い、Rustから受け取ったメモリはすぐに解放してしまう。[3]
import ctypes
import numpy as np
lib = ctypes.cdll.LoadLibrary('kmeans/target/release/kmeans.dll')
class KmeansResult(ctypes.Structure):
_fields_ = [
('cluster_indices', ctypes.POINTER(ctypes.c_size_t)),
('means', ctypes.POINTER(ctypes.c_double)),
]
lib.kmeans.argtypes = [
ctypes.POINTER(ctypes.c_double),
ctypes.c_size_t,
ctypes.c_size_t,
ctypes.c_size_t,
ctypes.c_size_t,
]
lib.kmeans.restype = ctypes.POINTER(KmeansResult)
lib.free_kmeans_result.argtypes = [ctypes.POINTER(KmeansResult)]
lib.free_f64_array.argtypes = [ctypes.POINTER(ctypes.c_double), ctypes.c_size_t]
lib.free_usize_array.argtypes = [ctypes.POINTER(ctypes.c_size_t), ctypes.c_size_t]
def rust_kmeans(X, k, num_steps=100):
n, d = X.shape
ret = lib.kmeans(X.ctypes.data_as(ctypes.POINTER(ctypes.c_double)), n, d, k, num_steps)
cluster_indices = np.ctypeslib.as_array(ret.contents.cluster_indices, shape=(n,)).copy()
means = np.ctypeslib.as_array(ret.contents.means, shape=(k, d)).copy()
lib.free_kmeans_result(ret)
lib.free_usize_array(ret.contents.cluster_indices, cluster_indices.size)
lib.free_f64_array(ret.contents.means, means.size)
return cluster_indices, means
これでPython側からRust実装を呼び出すことも出来るようになりました。
次はいよいよ実験と行きたいところですが、その前にもうひとつ試してみたいことがあります。
Rust で実装した k-means を並列化する
ネットとかでRustについて調べていると、Rustは並列処理が得意といような内容がしばしばみられる[4]ので、今回実装した kmeans を並列化するとどんな感じになるのかちょっと試してみたいと思います。
Rustで並列処理を簡単にやる方法について調べてみると、どうやらrayonというクレートを使うとイテレータで実装している部分をかなり簡単に並列化出来るようなので試してみました。rayonを使って並列化された実装は以下の通りです。
先ほどのコードと見比べてみると、.iter となっていた部分が .par_iter となっていたりと、微妙に違う部分が所々にあるのが分かると思います。
use rand::Rng;
use rayon::prelude::*;
/// 各クラスタの平均ベクトルを求める (並列版)
fn par_calc_means(data: &[Vec<f64>], k: usize, cluster_indices: &[usize]) -> Vec<Vec<f64>> {
let d = data[0].len();
(0..k)
.into_par_iter()
.map(|j| {
let mut mean = vec![0.0; d];
let mut count = 0;
for (&c, x) in cluster_indices.iter().zip(data.iter()) {
if c == j {
for l in 0..d {
mean[l] += x[l];
}
count += 1;
}
}
if count == 0 {
return mean;
}
for m in mean.iter_mut() {
*m /= count as f64;
}
mean
})
.collect()
}
/// 2つのベクトル間のユークリッド距離の2乗を計算する
fn calc_dist(x: &[f64], y: &[f64]) -> f64 {
x.iter()
.zip(y.iter())
.map(|(xi, yi)| (xi - yi).powi(2))
.sum()
}
/// ベクトルxに最も近い平均ベクトルのインデックスを取得する
fn get_min_index(x: &[f64], means: &[Vec<f64>]) -> usize {
let mut min_dist = std::f64::MAX;
let mut min_index = 0;
for (j, m) in means.iter().enumerate() {
let dist = calc_dist(x, m);
if dist < min_dist {
min_dist = dist;
min_index = j;
}
}
min_index
}
/// kmeans algorithm (並列版)
fn _par_kmeans(data: Vec<Vec<f64>>, k: usize, num_steps: usize) -> (Vec<usize>, Vec<Vec<f64>>) {
// init clusters
let n = data.len();
let mut rng = rand::thread_rng();
let mut cluster_indices: Vec<usize> = (0..n).map(|_| rng.gen_range(0..k)).collect();
let mut means = par_calc_means(&data, k, &cluster_indices);
// traininig
for _ in 0..num_steps {
cluster_indices = data.par_iter().map(|x| get_min_index(x, &means)).collect();
means = par_calc_means(&data, k, &cluster_indices);
}
(cluster_indices, means)
}
/// kmeansの結果をPythonに返すための構造体
#[repr(C)]
pub struct KmeansResult {
pub cluster_indices: *const usize,
pub means: *const f64,
}
/// # Safety
/// Pythonからデータを受け取ってkmeansを実行する (並列版)
#[no_mangle]
pub unsafe extern "C" fn par_kmeans(
array: *const f64,
rows: usize,
cols: usize,
k: usize,
num_steps: usize,
) -> *const KmeansResult {
let a = unsafe { std::slice::from_raw_parts(array, rows * cols) };
let data: Vec<Vec<f64>> = a.par_chunks(cols).map(|x| x.to_vec()).collect();
let (cluster_indices, means) = _par_kmeans(data, k, num_steps);
let means = means.into_par_iter().flatten().collect::<Vec<f64>>();
let kmeans_result = KmeansResult {
cluster_indices: cluster_indices.as_ptr(),
means: means.as_ptr(),
};
std::mem::forget(cluster_indices);
std::mem::forget(means);
Box::into_raw(Box::new(kmeans_result))
}
別に驚くべきことでも何でもないですが、目につく繰り返し処理を全部イテレータにして、イテレータは全部 par_iter とか into_par_iter で並列化して、とすれば早くなる訳ではなく、むしろ並列化のためのオーバーヘッドで逆に遅くなってしまうことも多いです。
細かい話は省きますが、上の実装は色々と試した中で、「明らかに高速化の効果があったもの」と「効果は微妙だが少なくともマイナスの効果ではなさそうなもの」だけを残した実装になっています。
なお、rayonで並列化した中で効果が特に大きかったと思われる部分は以下の2つです。
-
calc_meansの実装の、データ点にわたるforでやっていた処理を、クラスタインデックスにわたるイテレータを使った実装に変えて、into_par_iterでクラスタ毎に並列に中心点を求めるようにした。
(元々の実装と比べると冗長な処理も発生してしまう[5]が、実験で試したところ並列化による高速化の効果の方が遥かに大きかった。) -
cluster_indicesを更新するところのiterをpar_iterに変更
とりあえず、これで並列化したk-meansの実装も完了しました。
Pythonから呼び出す方法については、並列化していない実装の時と同じなので省略します。
実験
実験環境
- OS: Windows11
- CPU: Core i7-12700
- rustc 1.65.0
- Python 3.10.8
- Numpy 1.23.5
各実装の速度比較
以下のようなスクリプトを作成して、データ点数を様々に変えながら各実装の所要時間がどれくらい変わるかを試していきました。
今回比較したのは以下の4つです。
-
kmeans: Numpyで実装した k-means -
sklearn_kmeans: scikit-learn の k-means -
rust_kmeans: Rustで実装した k-means -
rust_par_kmeans: Rustで実装した k-means (並列化あり)
import ctypes
import time
import numpy as np
import pandas as pd
from sklearn.cluster import KMeans
lib = ctypes.cdll.LoadLibrary('kmeans/target/release/kmeans.dll')
class KmeansResult(ctypes.Structure):
_fields_ = [
('cluster_indices', ctypes.POINTER(ctypes.c_size_t)),
('means', ctypes.POINTER(ctypes.c_double)),
]
lib.kmeans.argtypes = [
ctypes.POINTER(ctypes.c_double),
ctypes.c_size_t,
ctypes.c_size_t,
ctypes.c_size_t,
ctypes.c_size_t,
]
lib.kmeans.restype = ctypes.POINTER(KmeansResult)
lib.par_kmeans.argtypes = [
ctypes.POINTER(ctypes.c_double),
ctypes.c_size_t,
ctypes.c_size_t,
ctypes.c_size_t,
ctypes.c_size_t,
]
lib.par_kmeans.restype = ctypes.POINTER(KmeansResult)
lib.free_kmeans_result.argtypes = [ctypes.POINTER(KmeansResult)]
lib.free_f64_array.argtypes = [ctypes.POINTER(ctypes.c_double), ctypes.c_size_t]
lib.free_usize_array.argtypes = [ctypes.POINTER(ctypes.c_size_t), ctypes.c_size_t]
num_samples = [1000, 10000, 100000, 1000000]
def kmeans(X, k, num_steps=100):
def calc_means(X, k, cluster_indices):
return np.array([X[cluster_indices == i].mean(axis=0) for i in range(k)])
# init clusters
N, _ = X.shape
cluster_indices = np.random.randint(0, k, size=(N,))
means = calc_means(X, k, cluster_indices)
# training
for _ in range(num_steps):
cluster_indices = ((np.expand_dims(X, 1) - means) ** 2).sum(axis=2).argmin(axis=1)
means = calc_means(X, k, cluster_indices)
return cluster_indices, means
def sklearn_kmeans(X, k, num_steps=100):
clf = KMeans(n_clusters=k, max_iter=num_steps, init='random', n_init=1, tol=0.0)
cluster_indices = clf.fit_predict(X)
means = clf.cluster_centers_
return cluster_indices, means
def rust_kmeans(X, k, num_steps=100):
n, d = X.shape
ret = lib.kmeans(X.ctypes.data_as(ctypes.POINTER(ctypes.c_double)), n, d, k, num_steps)
cluster_indices = np.ctypeslib.as_array(ret.contents.cluster_indices, shape=(n,)).copy()
means = np.ctypeslib.as_array(ret.contents.means, shape=(k, d)).copy()
lib.free_kmeans_result(ret)
lib.free_usize_array(ret.contents.cluster_indices, cluster_indices.size)
lib.free_f64_array(ret.contents.means, means.size)
return cluster_indices, means
def rust_par_kmeans(X, k, num_steps=100):
n, d = X.shape
ret = lib.par_kmeans(X.ctypes.data_as(ctypes.POINTER(ctypes.c_double)), n, d, k, num_steps)
cluster_indices = np.ctypeslib.as_array(ret.contents.cluster_indices, shape=(n,)).copy()
means = np.ctypeslib.as_array(ret.contents.means, shape=(k, d)).copy()
lib.free_kmeans_result(ret)
lib.free_usize_array(ret.contents.cluster_indices, cluster_indices.size)
lib.free_f64_array(ret.contents.means, means.size)
return cluster_indices, means
def benchmark(f):
n_trials = 10
d = 10
times = []
for n in num_samples:
tmp = []
for _ in range(n_trials):
X = np.random.random(size=(n, d))
start = time.time()
f(X, 5, 100)
end = time.time()
tmp.append(end - start)
times.append(tmp)
return times
if __name__ == '__main__':
results = {}
results['kmeans'] = np.array(benchmark(kmeans)).mean(axis=1)
results['sklearn_kmeans'] = np.array(benchmark(sklearn_kmeans)).mean(axis=1)
results['rust_kmeans'] = np.array(benchmark(rust_kmeans)).mean(axis=1)
results['rust_par_kmeans'] = np.array(benchmark(rust_par_kmeans)).mean(axis=1)
df = pd.DataFrame(data=results, index=num_samples)
print(df)
実験の結果は以下のようになりました。
kmeans sklearn_kmeans rust_kmeans rust_par_kmeans
1000 0.019617 0.035934 0.004695 0.004682
10000 0.320295 0.039109 0.024943 0.015625
100000 3.431728 0.228175 0.273535 0.076563
1000000 35.251610 2.027506 4.198329 1.093956
各行はデータサイズ、各列はそれぞれの手法に対応しています。書かれている数字は平均実行時間 (秒)です。
簡単にまとめると以下のような結果だったと言えそうです。
並列化ありRust > scikit-learn > 並列化なしRust > NumPy の順に早い [6]
驚くべきことに、Cythonとかを使って高速化されているはずの scikit-learnの実装よりも、並列化した Rust 実装の方が倍くらい早いという結果になっています。
今回実装した k-means は、scikit-learnの実装している k-means とは完全に同じではないと思うのであくまでも参考記録としておきたいところですが、この結果は予想外でした。
あと、どうでもいい話かもしれませんが、自分なりにテクニックを駆使して作ったNumPy実装だけが圧倒的に遅いのが残念でした。まだ高速化の余地はありますが、ここまで差が大きいとRust実装に勝つのは流石に無理ですね。
おまけ
k-meansクラスタリングの記事を書いておきながら、実際にクラスタリングした結果どうなるかという話が一切なかったので、最後にRust実装のクラスタリング結果を視覚化してみたいと思います。
以下のようなコードを書いて...
import ctypes
import matplotlib.pyplot as plt
import numpy as np
lib = ctypes.cdll.LoadLibrary('kmeans/target/release/kmeans.dll')
class KmeansResult(ctypes.Structure):
_fields_ = [
('cluster_indices', ctypes.POINTER(ctypes.c_size_t)),
('means', ctypes.POINTER(ctypes.c_double)),
]
rust_kmeans = lib.par_kmeans
rust_kmeans.argtypes = [
ctypes.POINTER(ctypes.c_double),
ctypes.c_size_t,
ctypes.c_size_t,
ctypes.c_size_t,
ctypes.c_size_t,
]
rust_kmeans.restype = ctypes.POINTER(KmeansResult)
lib.free_kmeans_result.argtypes = [ctypes.POINTER(KmeansResult)]
lib.free_f64_array.argtypes = [ctypes.POINTER(ctypes.c_double), ctypes.c_size_t]
lib.free_usize_array.argtypes = [ctypes.POINTER(ctypes.c_size_t), ctypes.c_size_t]
def rust_par_kmeans(X, k, num_steps=100):
n, d = X.shape
ret = rust_kmeans(X.ctypes.data_as(ctypes.POINTER(ctypes.c_double)), n, d, k, num_steps)
cluster_indices = np.ctypeslib.as_array(ret.contents.cluster_indices, shape=(n,)).copy()
means = np.ctypeslib.as_array(ret.contents.means, shape=(k, d)).copy()
lib.free_kmeans_result(ret)
lib.free_usize_array(ret.contents.cluster_indices, cluster_indices.size)
lib.free_f64_array(ret.contents.means, means.size)
return cluster_indices, means
def make_data():
means = np.array([
[1, 1],
[-1, 1],
[-1, -1],
[1, -1],
])
X = np.vstack([np.random.normal(m, 0.3, size=(100, 2)) for m in means])
return X
if __name__ == '__main__':
X = make_data()
cluster_indices, means = rust_par_kmeans(X, 4)
plt.scatter(X[:, 0], X[:, 1], c=cluster_indices)
plt.scatter(means[:, 0], means[:, 1], c='red', marker='x')
plt.savefig('kmeans_results.png')
出来上がったグラフがこちらです。
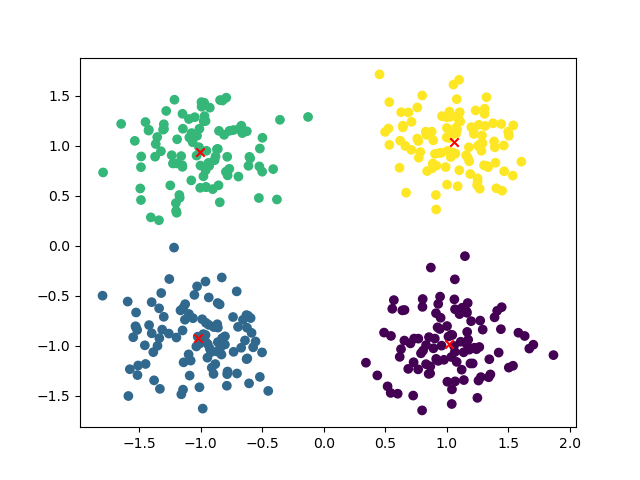
ちゃんと狙い通りにクラスタリング出来ているようです。
-
(N x D) と (K x D) の行列はそのままでは引き算 (ブロードキャスト) 出来ないが、
expand_dimsでXの方を (N x 1 x D) にしてから引き算を行うことで、(N x D)の方はK個に、(K x D)の方はN個にそれぞれ複製され (N x K x D) 同士の引き算が行われることになる。あとは二乗してsumをとって、argminを取るだけで、各データ点がどの中心点に近いかを計算することができる。 ↩︎ -
この処理を入れないとメモリリークが起きてしまうということを前の記事のコメント欄で検証しました。 ↩︎
-
これにより余計なコピーが発生してしまうわけですが、単純さと安全性重視でこうすることにしました。 ↩︎
-
例: https://doc.rust-jp.rs/book-ja/ch16-00-concurrency.html ↩︎
-
同じデータ点に対して、どのクラスタに属するかを確認する処理がk回発生してしまう(並列化する前は1回のみ)。 ↩︎
-
ただし、データサイズが小さい場合はその限りではない。 ↩︎
Discussion