医薬品副作用データセットを使ってローカルにデータ分析基盤を作る
コンテキスト:
- オープンデータを 1 次クリーニングしつつ DWH にロードしたい
- ここで扱うオープンデータは独立行政法人医薬品医療機器総合機構の下記サイトから得られるデータ https://www.info.pmda.go.jp/fukusayoudb/CsvDownload
- 四種類のデータがそれぞれ分割されて配布されている
- ローカルでやりたい
- 理由: 初めからクラウドでやってやってられなくなったため https://qiita.com/Rindrics/items/035d2a709ef198d2dbfe
アーキテクチャ
要件
- ローカルで完結できる
- できれば無料ツール
- データラングリングに強い言語を使える
ツール
- DuckDB: https://duckdb.org/
- dlt: https://dlthub.com/
- Rill Data: https://www.rilldata.com/
とりあえず一つのデータセットを Load してみる
とりあえず hist データは下記のコードで Load できた
import dlt
from filesystem import filesystem, read_csv
table_name = "hist"
files = filesystem(file_glob=f"{table_name}*.csv")
data = files | read_csv(encoding="cp932", names=["id", "times_reported", "disease_id", "disease"], header=0)
pipeline = dlt.pipeline(
pipeline_name="csv_pipeline",
destination="duckdb",
dataset_name="jader",
)
load_info = pipeline.run(
data,
table_name=table_name,
write_disposition="replace",
)
print(load_info)
[runtime]
log_level="INFO" # the system log level of dlt
dlthub_telemetry = true
[sources.filesystem]
bucket_url = "file://../sources/pmdacasereport202402/" # 相対パスで書く場合は呼び出し元(not toml dir)からの相対パスで書く必要がある
$ poetry run python jader_csv_pipeline.py
2024-02-27 01:34:41,028|[INFO ]|74852|140704591857216|dlt|pipeline.py|_restore_state_from_destination:1380|The state was restored from the destination duckdb(dlt.destinations.duckdb):jader
2024-02-27 01:34:55,160|[INFO ]|74852|140704591857216|dlt|pool_runner.py|run_pool:74|Created none pool with 1 workers
2024-02-27 01:34:55,160|[INFO ]|74852|140704591857216|dlt|normalize.py|run:355|Running file normalizing
2024-02-27 01:34:55,161|[INFO ]|74852|140704591857216|dlt|normalize.py|run:358|Found 1 load packages
2024-02-27 01:34:55,169|[INFO ]|74852|140704591857216|dlt|normalize.py|run:366|Found 1 files in schema csv load_id 1708965281.071473
2024-02-27 01:34:55,174|[INFO ]|74852|140704591857216|dlt|normalize.py|spool_schema_files:334|Created new load package 1708965281.071473 on loading volume
(余談)分割されたデータセットを処理する
https://zenn.dev/link/comments/a2f4843510befb の時点では、JADER データセットダウンロードページの「一括ダウンロード」から得られる全データセットを処理していたことに気付いた。
dlt ではせっかく file_glob でパターンマッチできるので、分割データを読み込むように変更してみる。
そうすることで、なにか問題があったときにデバッグしやすい可能性がある
分割版ファイルのダウンロードは下記のようにすることでミスなく実行できる:
import os
from datetime import datetime
from selenium import webdriver
from selenium.webdriver.common.by import By
from selenium.webdriver.chrome.options import Options
import time
import zipfile
from tqdm import tqdm
# Set up download destination
folder_name = datetime.now().strftime('%Y-%m-%d_%H-%M-%S')
downloads_path = os.path.expanduser(f"~/Downloads/{folder_name}")
os.makedirs(downloads_path, exist_ok=True)
# Set up Chrome options
chrome_options = Options()
prefs = {"download.default_directory" : downloads_path}
chrome_options.add_experimental_option("prefs", prefs)
# Open Chrome and go to the page
url = "https://www.info.pmda.go.jp/fukusayoudb/CsvDownload"
driver = webdriver.Chrome(options=chrome_options)
driver.get(url)
# To pass image auth
captcha_text = input("画像認証の文字列を入力してください: ")
captcha_input = driver.find_element(By.ID, "captchaText")
captcha_input.send_keys(captcha_text)
# Get partial download buttons and click them
buttons = driver.find_elements(By.XPATH, "//input[contains(@value, '分割ファイル') and contains(@value, 'ダウンロード')]")
print("ファイルのダウンロード中...")
for button in buttons:
button.click()
time.sleep(1)
# Check if all files are downloaded
pbar = tqdm(total=len(buttons), desc="ダウンロード完了待ち", unit="file")
downloaded_files_count = 0
while downloaded_files_count < len(buttons):
time.sleep(1) # 毎秒チェック
downloaded_files = [name for name in os.listdir(downloads_path) if name.endswith('.zip')]
new_count = len(downloaded_files)
if new_count > downloaded_files_count:
pbar.update(new_count - downloaded_files_count)
downloaded_files_count = new_count
pbar.close()
# Extract all files
downloaded_files = [name for name in os.listdir(downloads_path) if name.endswith('.zip')]
print("zip ファイルの解凍中")
for file_name in downloaded_files:
file_path = os.path.join(downloads_path, file_name)
with zipfile.ZipFile(file_path, 'r') as zip_ref:
zip_ref.extractall(downloads_path)
os.remove(file_path)
print("完了")
これをやるなら encoding 変換もやればいいのでは?となったが、これはあくまでも「手作業でのダウンロードを少し効率化するだけ」という立ち位置のスクリプトと割り切り、encoding 変換を含むデータ操作は dlt でやることにする
ここで取得した分割データセットは全て https://zenn.dev/link/comments/8a32bc73db87b3 で問題なく処理できた
Load ステップ
https://zenn.dev/link/comments/a2f4843510befb では hist データのみを処理したが、下記のようにすることで四種類すべてのデータを Load できた
import dlt
from filesystem import filesystem, read_csv
datasets = {
"demo": {
"file_glob": "demo*.csv",
"columns": ["id", "times_reported", "sex", "age", "weight", "height", "reporting_year_quarter", "reporting_status", "report_type", "reporter_qualification", "e2b"]
},
"drug": {
"file_glob": "drug*.csv",
"columns": ["id", "times_reported", "drug_id", "drug_name", "drug_generic_name", "drug_marketing_name", "route", "administration_start_date", "administration_end_date", "dose", "dose_unit", "dose_division", "dose_reason", "treatment", "is_relapse_caused_by_redose", "risk_category"]
},
"hist": {
"file_glob": "hist*.csv",
"columns": ["id", "times_reported", "disease_id", "disease"]
},
"reac": {
"file_glob": "reac*.csv",
"columns": ["id", "times_reported", "adverse_event_id", "adverse_event_name", "outcome", "onset_date"]
},
}
for name, settings in datasets.items():
files = filesystem(file_glob=settings["file_glob"])
data = files | read_csv(encoding="cp932", names=settings["columns"], header=0)
pipeline = dlt.pipeline(
pipeline_name="jader_csv",
destination="duckdb",
dataset_name="jader",
)
load_info = pipeline.run(
data,
table_name=name,
write_disposition="replace",
)
print(f"Loaded {name}: ", load_info)
処理が完了したら dlt pipeline jader_csv show を実行することで Streamlit 製のアプリが立ち上がり、Load 済データを見ることができる
地味にありがたかったのが、非正規型データへの耐性。
jader データセットは、セミコロン区切りで複数レコードが入力された列を含むが、このようなデータも下記の通りにちゃんと読み込めている:

Transform は別でやるのでとりあえず Load したいだけという今回のケースでは非常にありがたかった(以前 dlt を使わない方法では失敗したことがあった)。
もっとも、これは dlt が利用している Pandas の read_csv() の扱いやすさのおかげかもしれない。
Transform の設計
- https://zenn.dev/link/comments/8a32bc73db87b3 でとりあえずの Load に成功した
- まずどんな指標が欲しいかを考え、そこから逆算して必要な Transform 処理を洗い出す
- 理由: 非正規形データが入っているのはわかっているが、使われないカラムを操作しても時間の無駄であるため
必要な指標
藤田(2009)[1] で解説されている下記の 4 指標:
- PRR (Proportional Reporting Ratio)
- ROR (Reporting Odds Ratio)
- IC (Information Compornent)
- RR (Relative Reporting)
これらの 4 指標は、下のマトリクスで表される 4 種類の方法で集計された有害事象(副作用のこと)の報告数を使って求められる

藤田(2009)表 2
よって、Transform 処理は下記の手順で進める
-
drugとreacをid列をキーとしてジョインしてdrug_reacとしておく - n_11, n_12, n_21, n_22 をそれぞれ計算しつつ ROR を求める
-
藤田利治(2009)副作用評価におけるシグナル検出, 薬剤疫学14(1) 27--36. https://www.jstage.jst.go.jp/article/jjpe/14/1/14_1_27/_pdf ↩︎
Rill から DuckDB に接続してソースをセットアップ
https://zenn.dev/link/comments/8a32bc73db87b3 によって、4 種類のテーブルを DuckDB に load 完了したところ
(Rill を使うのが初めてなので)まず手作業でソース定義・モデル定義・可視化してみる
Rill を立ち上げてソースを追加する
'+Add source' をクリック
DuckDB を選択

ソースをロードする
dlt の streamlit 製ビューアを使って成功するクエリを確かめておくと楽
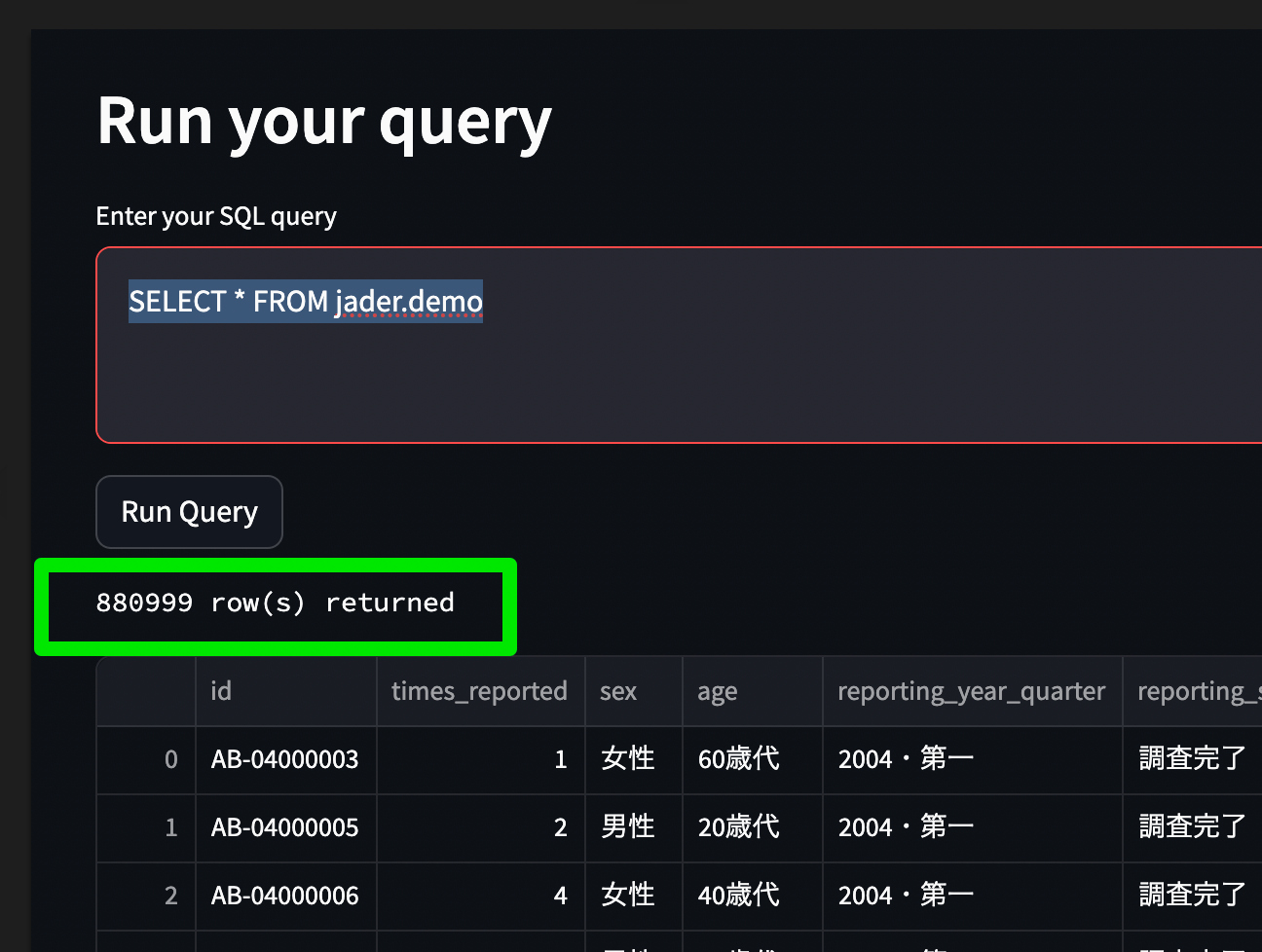
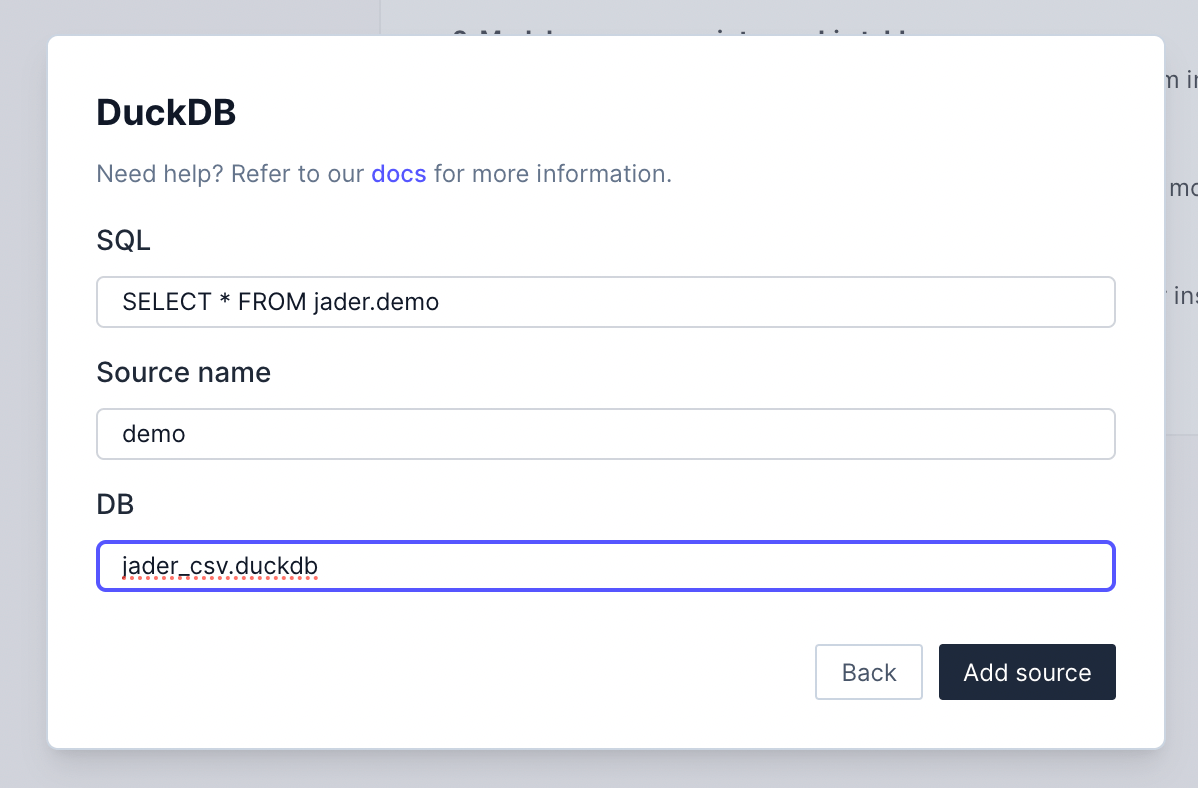
入力モーダルに下記を入力する
- SQL: 成功したロート用クエリ
- Source name: ロード後のソース名
- DB:
.duckdbのパス。相対も利用可
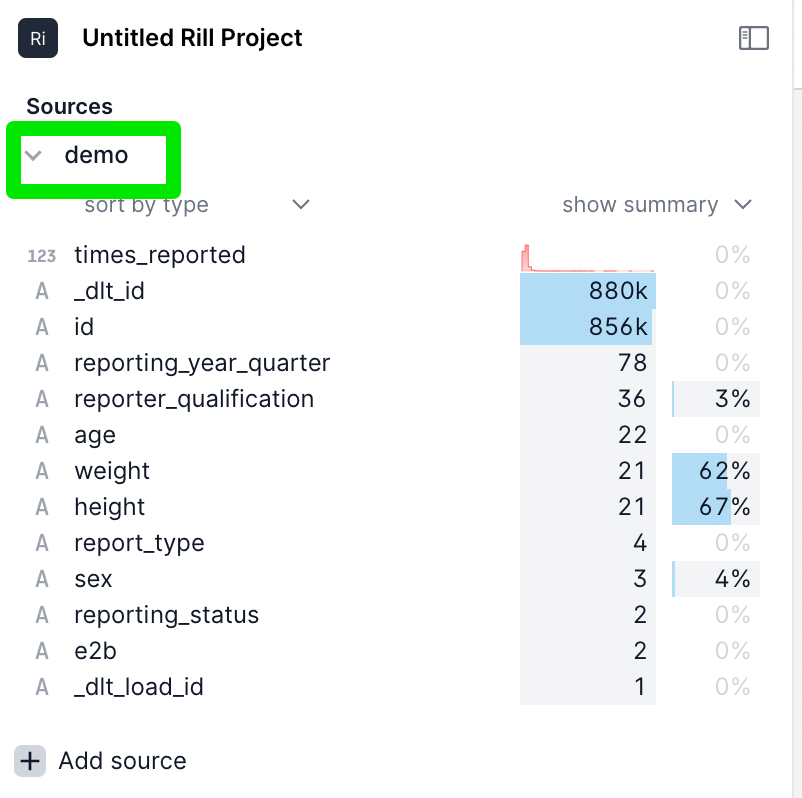
ロードに成功し、各カラムの統計情報が表示される
モデルを定義する
https://zenn.dev/link/comments/a78229ade1f684 で Transform を設計したが、Transform を別ツールでやると考えること増えるのでモデル定義もまず Rill に書いてしまう予定
drug_reac
select
d.id as id,
d.drug_id as drug_id,
d.drug_generic_name as drug_generic_name,
r.adverse_event_id as adverse_event_id,
r.adverse_event_name as adverse_event_name
from drug d
left join reac r on d.id = r.id
問題ない
ROR
drug_reac を使って ROR を計算する。
最終的には下記のクエリを書きたいのだが
SELECT
dr1.drug_id,
dr1.adverse_event_id,
(COUNT(DISTINCT dr1.id) * COUNT(DISTINCT dr4.id)) /
(COUNT(DISTINCT dr2.id) * COUNT(DISTINCT dr3.id)) AS ROR
FROM
drug_reac as dr1
LEFT JOIN
drug_reac as dr2 ON dr1.drug_id = dr2.drug_id AND dr1.adverse_event_id != dr2.adverse_event_id
LEFT JOIN
drug_reac as dr3 ON dr1.adverse_event_id = dr3.adverse_event_id AND dr1.drug_id != dr3.drug_id
LEFT JOIN
drug_reac as dr4 ON dr1.drug_id != dr4.drug_id AND dr1.adverse_event_id != dr4.adverse_event_id
GROUP BY
dr1.drug_id,
dr1.adverse_event_id
この時点ですでにクエリ速度が遅すぎて厳しい
select
d.id as id,
d.drug_id as drug_id,
d.drug_generic_name as drug_generic_name,
r.adverse_event_id as adverse_event_id,
r.adverse_event_name as adverse_event_name
from drug d
left join reac r on d.id = r.id

ちなみに Activity Monitor を見た感じ、CPU がきついらしい