Cell segmentationモデル訓練に有用なデータセット
明視野の病理画像、蛍光の病理画像、蛍光の培養細胞画像などを対象に深層学習モデルを訓練するときの正解ラベル付きの画像Datasetのメモ。
実際にダウンロードして確認していないものも多数あるので、本当に有用かは保障しない。画像として汚いものやパッチサイズが小さすぎるものなどもあるので要確認。
↓↓ 明視野の病理画像 ↓↓
【 TNBC_2018 】
- Triple Negative Breast Cancer
- HEやIHC画像
- 11症例、50枚の画像
- 512*512 pxのタイル
- 細胞の中心座標から背景までのDistance mapを使った初期の論文で使われているdataset。https://ieeexplore.ieee.org/document/8438559
- ダウンロード先
Zenodoにdepositあり。明視野画像と細胞のバイナリマスク画像のセット。https://zenodo.org/records/1175282#.WyP61xy-l5E
上記のDatasetにTCGAの脳のHE画像を追加し、さらに細胞種のラベル画像、細胞インスタンスラベル画像も追加されている。Zenodo 3552674: https://zenodo.org/records/3552674
【 PanNuke 】
- 19の組織のHE画像
- 256 × 256 pxのパッチ画像
- 7904枚
- 20万の細胞核のラベル
- arXiv (2020): https://arxiv.org/abs/2003.10778
- ダウンロード先:
HoVerNetのGitHubページ内にあるリンクでは、リンク先が変わっていてWarwick大学のTIA centerのページに飛ぶ。Datasetタブを見てもPanNukeは無し。
2024年9月時点で、このリンクは生きていた。 https://warwick.ac.uk/fac/cross_fac/tia/data/pannuke
Kaggleにもある? https://www.kaggle.com/datasets/llwlabs/pannuke
PathMLライブラリを使えば訓練/検証データなどを生成できるかも。https://pathml.readthedocs.io/en/latest/api_datasets_reference.html
【 MoNuSeg 】
- TCGAのHE画像
- 40倍対物レンズ
- 1000*1000 pxのタイル
- 細胞マスクと細胞膜マスクを予測する初期の論文で使われたdataset。
https://ieeexplore.ieee.org/abstract/document/7872382
https://ieeexplore.ieee.org/abstract/document/8880654
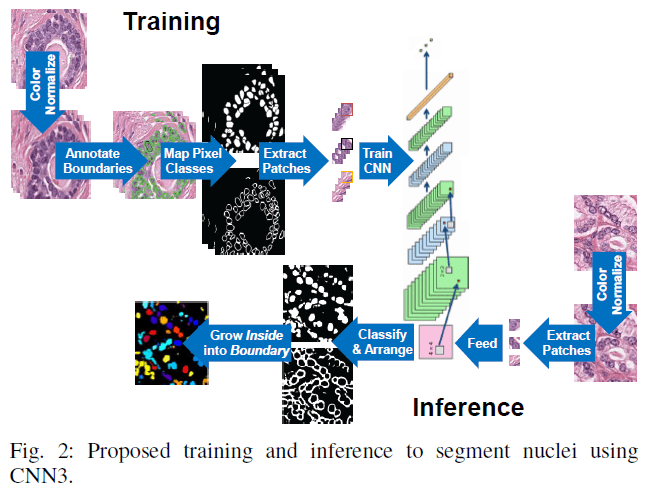
原著 Fig.2
- HoVer-Netの論文のKumar datasetはこれのことっぽい。
- ダウンロード先:
- EmbedSegのGitHubからreleaseページのリンクを辿ると「monuseg-2018.zip」のリンクがある。
中身を見ると、train 30枚、test 14枚のHE画像(tif)とそれに対応したインスタンスラベル画像(tif)が得られる。 - Grand Challengeというサイトからも取得可能。こちらはインスタンスラベルデータがxmlファイル。 https://monuseg.grand-challenge.org/Data/
【 MoNuSAC 】
- MoNuSegのCell instance segmentaionに加えて、細胞分類まで行うチャレンジで用意されたdataset
- TCGAのHE画像
- ダウンロード先: https://monusac-2020.grand-challenge.org/Data/
明視野画像(svsとtif)とインスタンスラベル情報(xml)がダウンロードできた。
【 CoNSeP 】
- Colorectal adenocarcinomaのHE画像
- 40倍対物レンズ
- 1000*1000 pxのタイル画像
HoVer-NetのGitHubページのリンクからアクセスすると、Warwick大学のサインインページに飛んでダウンロードできない。
【 CPM15 / CPM17 】
- TCGAのHE画像
- 細胞マスクと細胞膜マスクを予測するモデルの論文で使われたdataset。
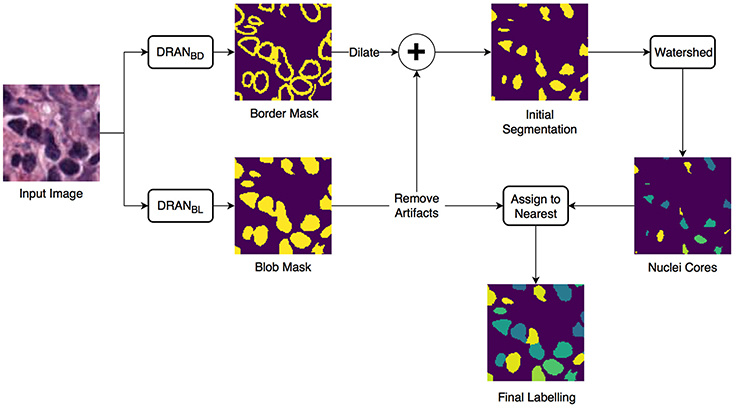
論文 2019: https://www.frontiersin.org/journals/bioengineering-and-biotechnology/articles/10.3389/fbioe.2019.00053/full - ダウンロード先: https://drive.google.com/drive/folders/1l55cv3DuY-f7-JotDN7N5nbNnjbLWchK
HoVer-Netの1st AuthorがGoogle driveで共有している。 - CPM15は画像サイズが不均一、CPM17は500*500px
- ラベルはmatlabファイル
【 Pan-Cancer-Nuclei-Seg 】
- 論文(Scientific Data, 2020): https://www.nature.com/articles/s41597-020-0528-1
- ダウンロード先: The Cancer Imaging Archive (TCIA)
https://www.cancerimagingarchive.net/analysis-result/pan-cancer-nuclei-seg/
【 NuCLS (Nucleus Classification, Localization and Segmentation) 】
- ホームページ: https://sites.google.com/view/nucls/home?authuser=0
- TCGAのHE画像
小さい領域のパッチ画像。 - DatasetのホームページのSingle-rater、Multi-raterからダウンロード可能。Singleは1名のアノテーター、Multiは32名のアノテーター。Multiはアノテーターごとのデータもあったりと冗長。
- 細胞核のセグメンテーションマスクと細胞分類ラベル

ホームページ Data Formatページ
細胞核のマスクはバウンディングボックスのものとセグメンテーションのものがある。
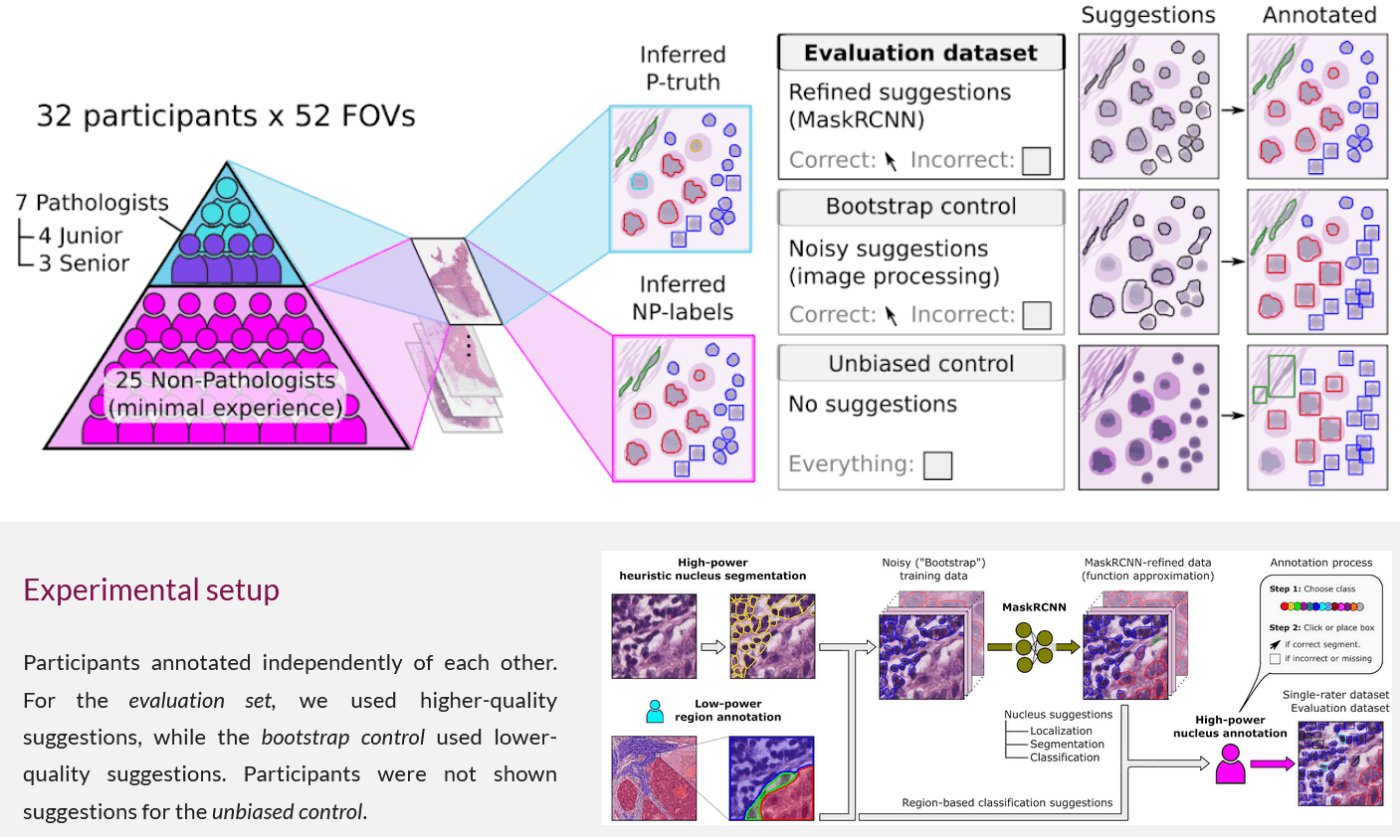
https://sites.google.com/view/nucls/multi-rater?authuser=0
schemaを見る感じでの想像だが、以下のことをやっていそう。
- 古典的な方法で核セグメンテーション
- 低解像度の広視野画像で腫瘍領域や間質領域などをアノテーション
- 細胞核が属する領域を細胞核のラベルとして採用 ➔ これがNoisy Bootstrap??
- 3.のデータを訓練データとしてMaskRCNNモデルを作る
- モデルの予測結果をアノテーターがチェック。間違っているとセグメンテーションではなく、バウンディングボックスで修正??
【 Pan-Cancer-Nuclei-Seg 】
- 論文(Scientific data, 2020): https://www.nature.com/articles/s41597-020-0528-1
- ダウンロード先:
The Cancer Imaging Archive: https://www.cancerimagingarchive.net/analysis-result/pan-cancer-nuclei-seg/
【 LyNSec 】
- びまん性大細胞型リンパ腫のHE、IHC画像
本文より「LyNSeC (lymphoma nuclear segmentation and classification) containing 73,931 annotated cell nuclei from H&E and 87,316 from IHC slides」 - 細胞のインスタンスラベルと、細胞種のラベルがある。
- 40x 対物レンズ
- 512*512 pxのタイル
- HoLy-Netというモデルの原著内で使われているDatasets。モデルはHoVerNetがベースでdropoutを追加したぐらい??
原著 (Computers in Biology and Medicine, 2024): https://www.sciencedirect.com/science/article/pii/S0010482524000623 - ダウンロード先
Zenodo: https://zenodo.org/records/8065174
【 CryoNuSeg 】
- TCGAのHE画像
- 凍結切片を対象にした??
- GitHub: https://github.com/masih4/CryoNuSeg
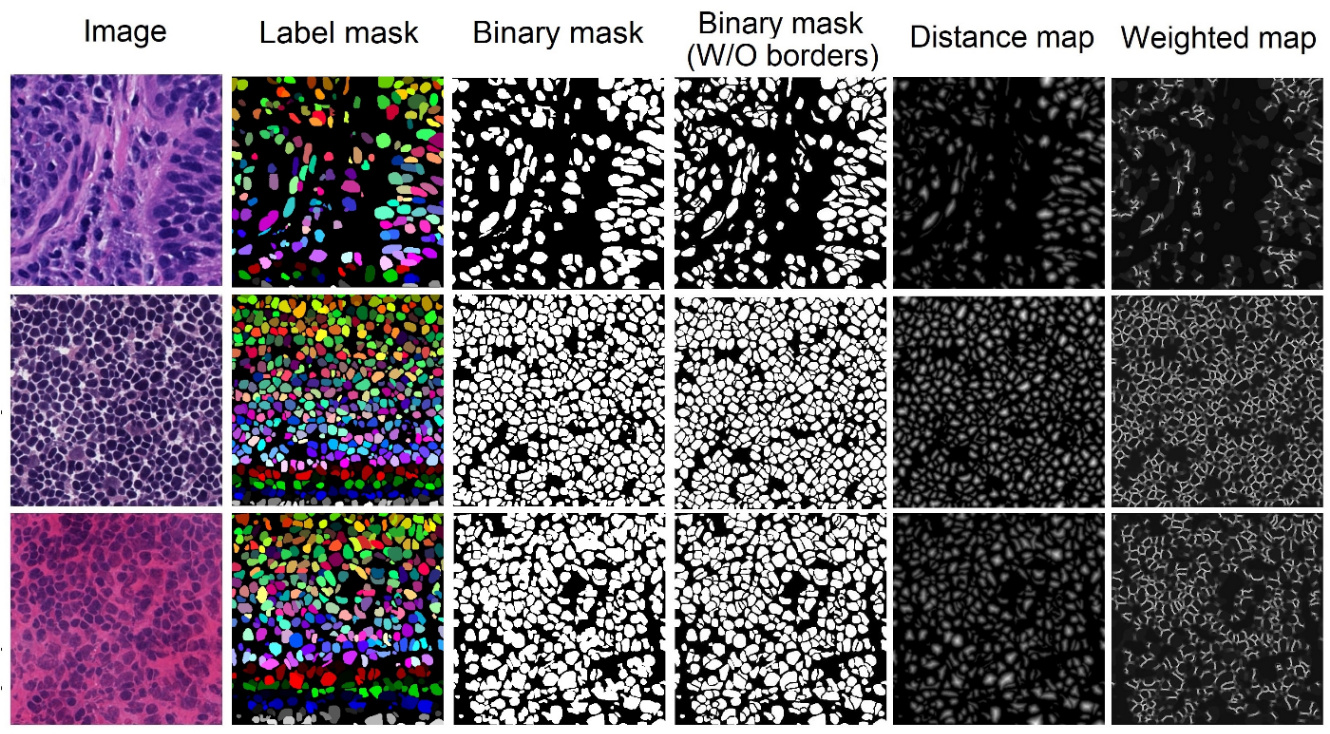
(Weighted mapは細胞がくっついているところの境界線みたい) - 原著(Computers in Biology and Medicine, 2021): https://www.sciencedirect.com/science/article/pii/S0010482521001438
- ダウンロード先
Kaggle: https://www.kaggle.com/datasets/ipateam/segmentation-of-nuclei-in-cryosectioned-he-images
明視野画像、マスク画像以外にもDistance mapなどもdepositされている。
【 NuInsSeg 】
- HE画像
- 上記CryotNuSegと同じチーム
- GitHub: https://github.com/masih4/NuInsSeg

GitHubより - 原著(Scientific Data, 2024): https://www.nature.com/articles/s41597-024-03117-2
- ダウンロード先:
Zenodo: https://zenodo.org/records/10518968
Kaggle: https://www.kaggle.com/datasets/ipateam/nuinsseg
【 CoNIC 】
- CoNIC: Colon Nuclei Identification and Counting Challenge
- 大腸のHE画像
- Grand challengeのデータセット https://conic-challenge.grand-challenge.org/
アカウント作ったら見れるかも。 - arXiv: https://arxiv.org/abs/2303.06274
【 HAPPY dataset 】
原著(Nature Communications, 2024): https://www.nature.com/articles/s41467-024-46986-2
GitHub: https://github.com/Nellaker-group/happy
-
核検出、細胞分類、組織分類の一連のモデル・ワークフローを提供
-
核検出:
RetinaNetモデルを使用。 -
細胞分類:
検出された1つの細胞核を含む200*200 pxの画像をResNet-50で分類 -
使用したdatasetの提供有り。GitHub内のリンクからGoogleドライブへ飛べる。
https://drive.google.com/drive/folders/1RvSQOxsWyUHf_SGV1Jzqa_Gc5QI4wQoy
この内の「datasets」フォルダに核検出用に使用した画像(1600*1200 px)と細胞分類に用いた画像(200*200 px)がある。
核検出の正解は「annotations/nuclei」フォルダのcsvファイルが各インスタンスのbounding box座標っぽい。
【 IHC_TMA_dataset 】
- 論文(Biomedical Signal Processing and Control, 2024): https://www.sciencedirect.com/science/article/abs/pii/S1746809424002015?via%3Dihub
このdatasetを使ったCell segmentation modelの論文 - ダウンロード先: https://zenodo.org/records/7647846
- 256*256の明視野画像pngと、maskはnpyファイル
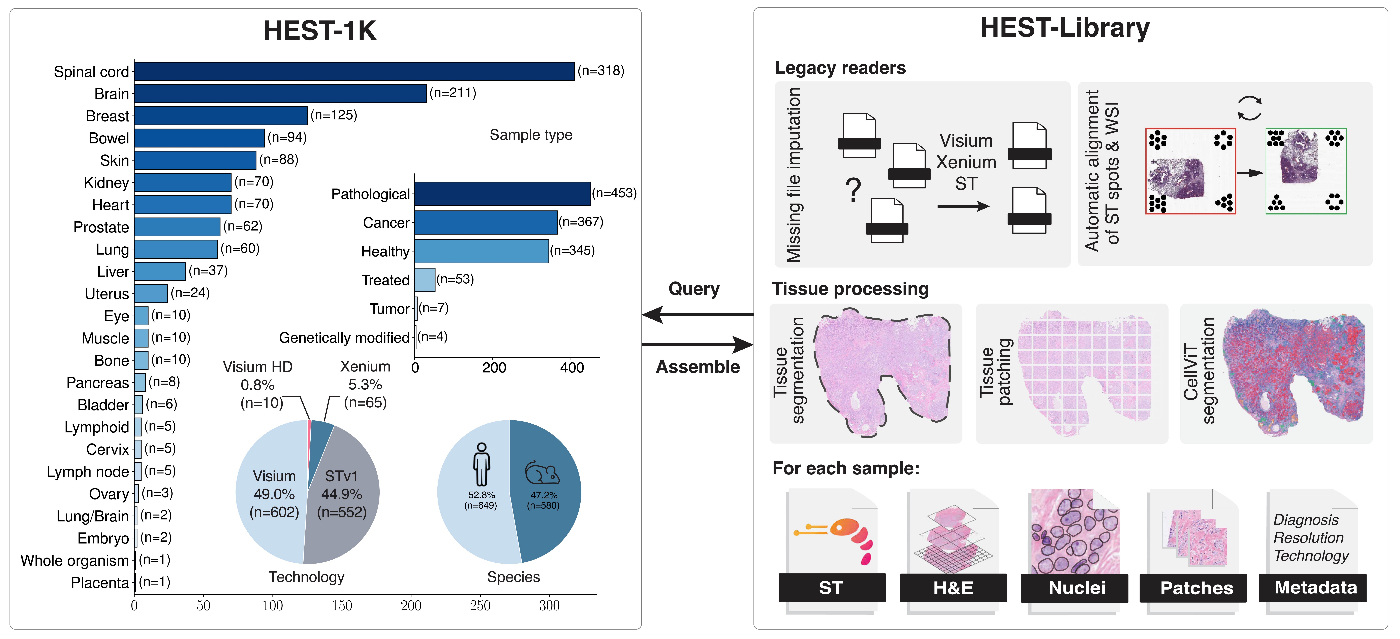
【 HESK1K 】
GitHub: https://github.com/mahmoodlab/hest
arXiv (2024): https://arxiv.org/abs/2406.16192
- 空間トランスクリプトームデータと画像データのデータセット。
高解像度画像、高解像度画像をパッチ化した画像、CellViTでsegmentationしたROIなどの情報(geojson)もある。 - 1TB以上あり
GitHubより
-
ドキュメントページもあり。HE画像と空間トランスクリプトームデータのmanipulationライブラリだってさ
https://hest.readthedocs.io/en/latest/
ダウンロード手順
HEST/tutorials/1-Downloading-HEST-1k.ipynbに従ってHuggingFaceにサインインして、HEST-1kのページへ。
「Agree and access repogitory」をクリック。審査もなくアクセス可能に。
ダウンロードにはToken発行して、Pythonスクリプトでダウンロードする。右上のアカウントメニューから「Access tokens」
次のページで「Create new token」
token名を「hest」、Writeタブを選択、「Create token」をクリック。
Token発行ページ
Tokenが表示されるのでこれを控えておく。
以下Pythonで処理。
datasets、huggingface_hubをインストール。
pip install datasets
pip install huggingface_hub
HuggingFaceにログイン。
import datasets
from huggingface_hub import login
login("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")
--> ログインは何のメッセージも無し。
ダウンロード。
local_dir='./HEST1K' # hest will be dowloaded to this folder
# Note that the full dataset is around 1TB of data
dataset = datasets.load_dataset(
'MahmoodLab/hest',
cache_dir=local_dir,
patterns='*'
)
こんな感じでダウンロードが進んでいった。

途中で止まってたけど、もう一度実行すると途中から再開してくれた。何度も止まりながら数日かかりでダウンロード。
↓↓ 蛍光の病理画像 ↓↓
【 TissueNet 】
-
蛍光染色の病理画像
-
Cell segmentation modelのMesmerの訓練使用されている。
原著(Nature Biotechnology, 2022): https://www.nature.com/articles/s41587-021-01094-0 -
ダウンロード先
本文中のData availabilityのリンクに飛ぶとDeepCellのページへ繋がる。https://datasets.deepcell.org/
アカウントを作ってログインするとダウンロード可能。
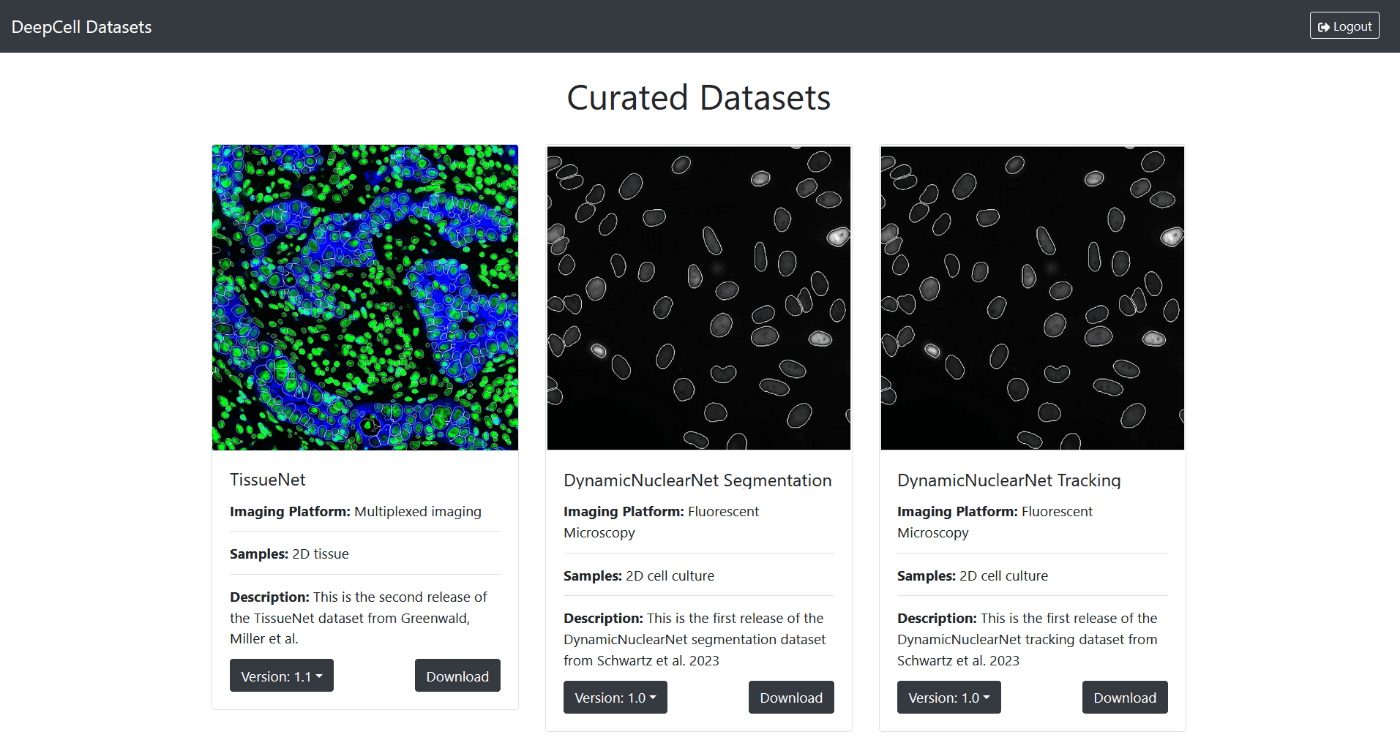
ログイン後の画面 -
Datasetの中身:
train/test/valの3つのnpzファイルがダウンロードされる。512*512 pxサイズの2 ch画像。ラベルはインスタンスラベル画像(細胞核のラベル画像と細胞のラベル画像で2 chある)。
import numpy as np
data = np.load("tissuenet_v1.1_train.npz", allow_pickle=True)
X = data["X"] # 顕微鏡画像
y = data["y"] # インスタンスラベル画像
meta = data["meta"] # ファイル名などの情報
- PythonのDeepCellライブラリを使っても訓練用/検証用データが用意できるみたい。 https://deepcell.readthedocs.io/en/master/data-gallery/tissuenet.html
【 CRC_FFPE-CODEX_CellNeighs 】
-
CODEXの蛍光マルチプレックスイメージング画像
-
原著(Cell, 2020): https://www.cell.com/cell/fulltext/S0092-8674(20)30870-9
-
ダウンロード先:
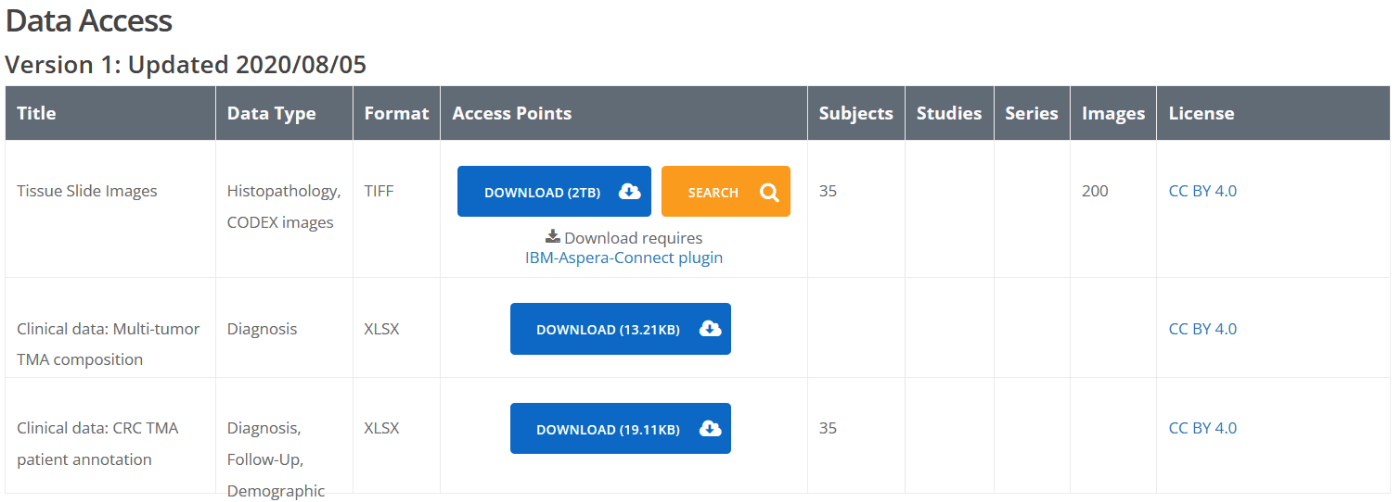
The Cancer Imaging Archive: https://www.cancerimagingarchive.net/collection/crc_ffpe-codex_cellneighs/ -
ダウンロード方法: IBM Aspera Connectを使ったダウンロード。
ダウンロード Step by step
IBM Aspera Connectのインストール
- The Cancer Imaging Archiveの上記リンクページ内の「Download」を押す。
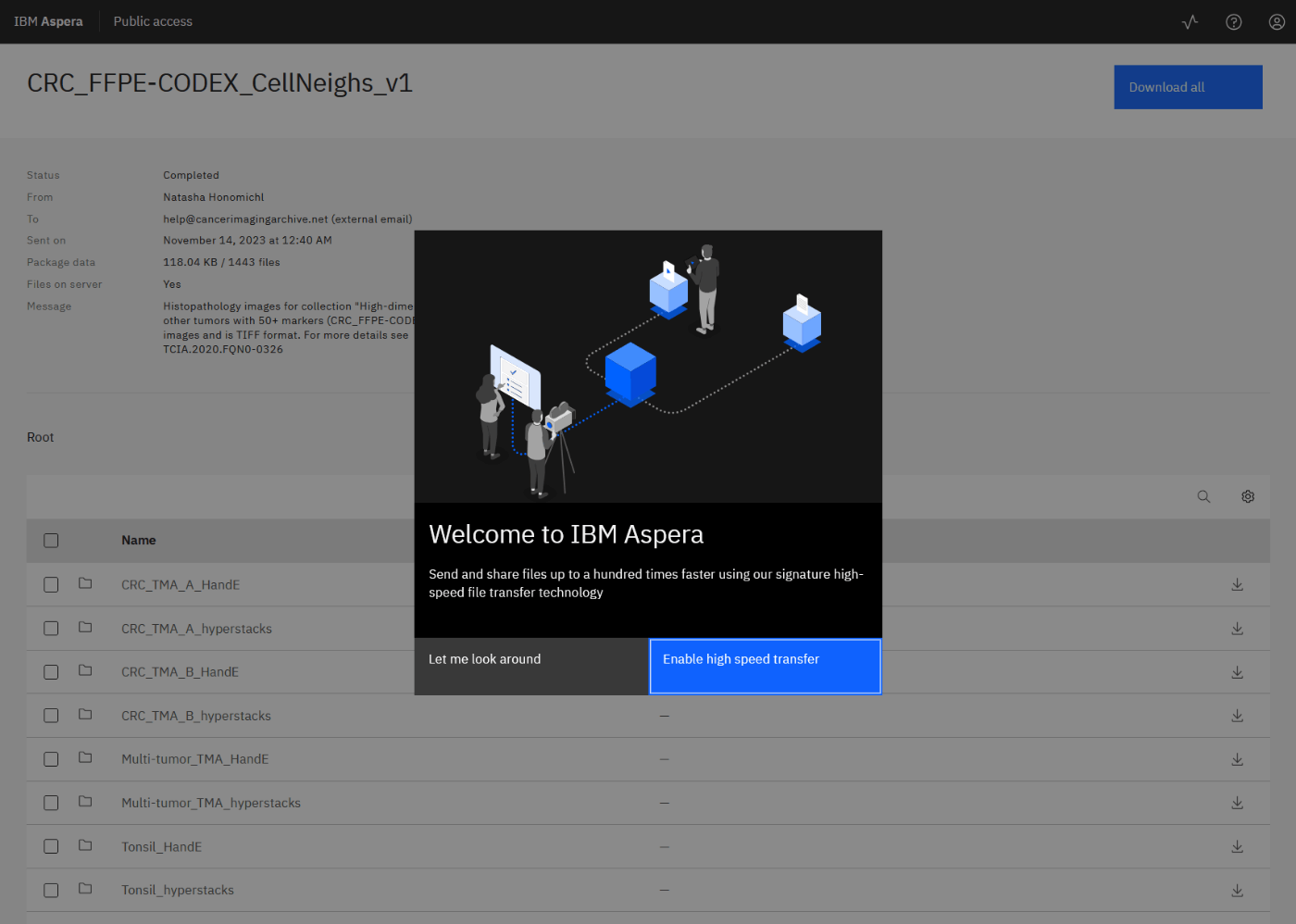
- Aspera Connectが未インストールであれば案内ページが出る。「Enable high speed transfer」をクリック。
- ブラウザの拡張機能をダウンロード。「Install extension」を押すと、ブラウザに合わせたアドオンのインストールページが立ち上がる。そこでインストールを進める。
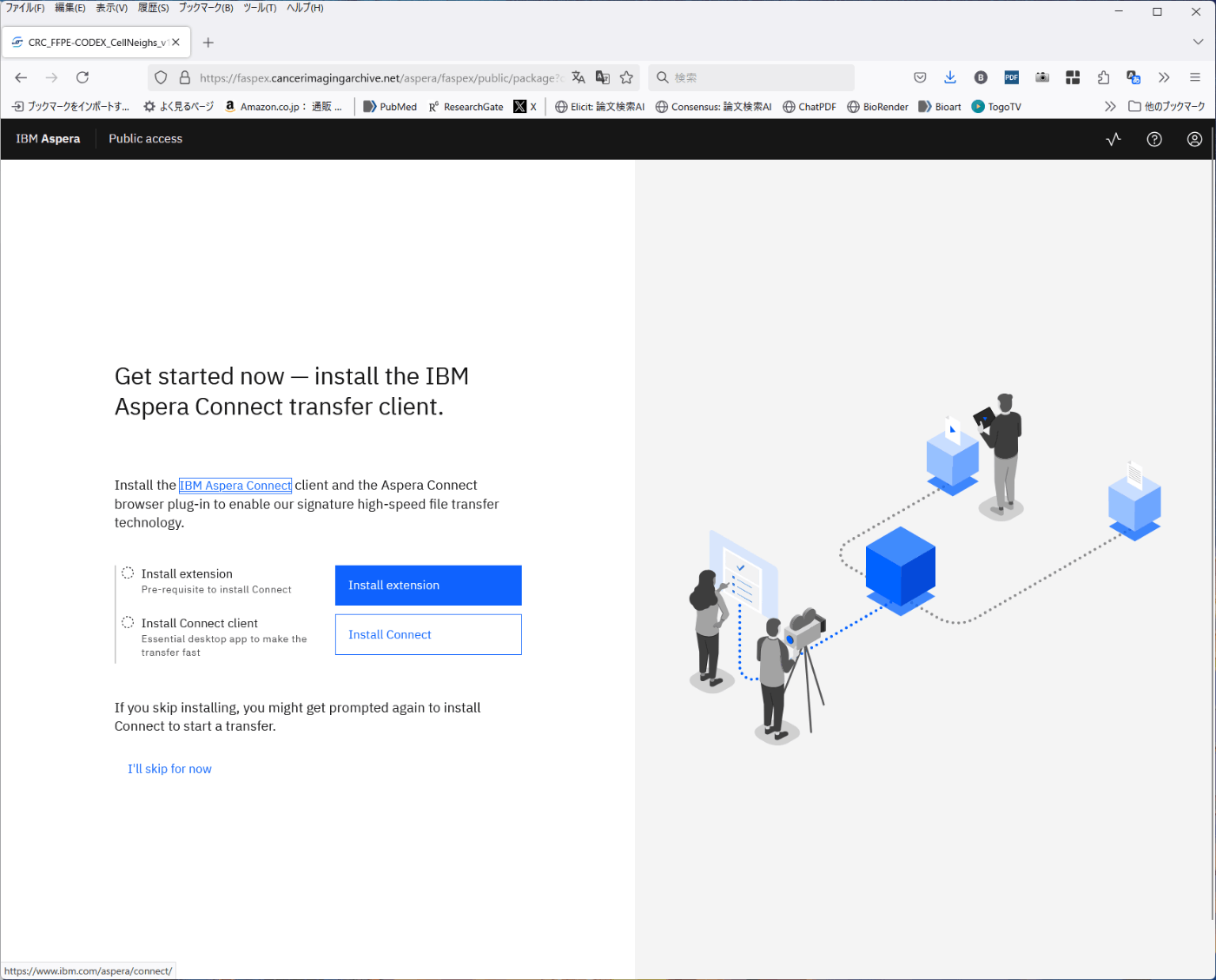
FireFoxの例
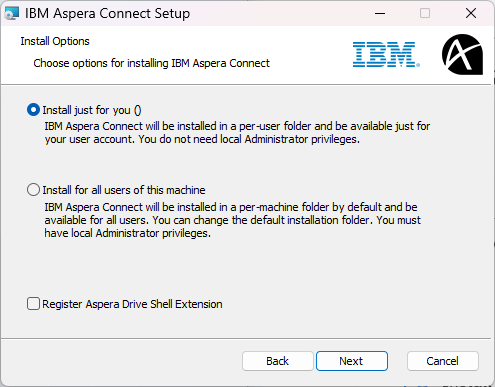
- Aspera Connectのインストール
「Install Connect」からインストーラーをダウンロード。インストーラーを立ち上げてインストールを進める。※ 「Register Aspera Drive Shell Extension」がデフォルトで選択されているが、Windowsエクスプローラーが干渉を受けて正常に動かなくなったので、このチェックは外している。
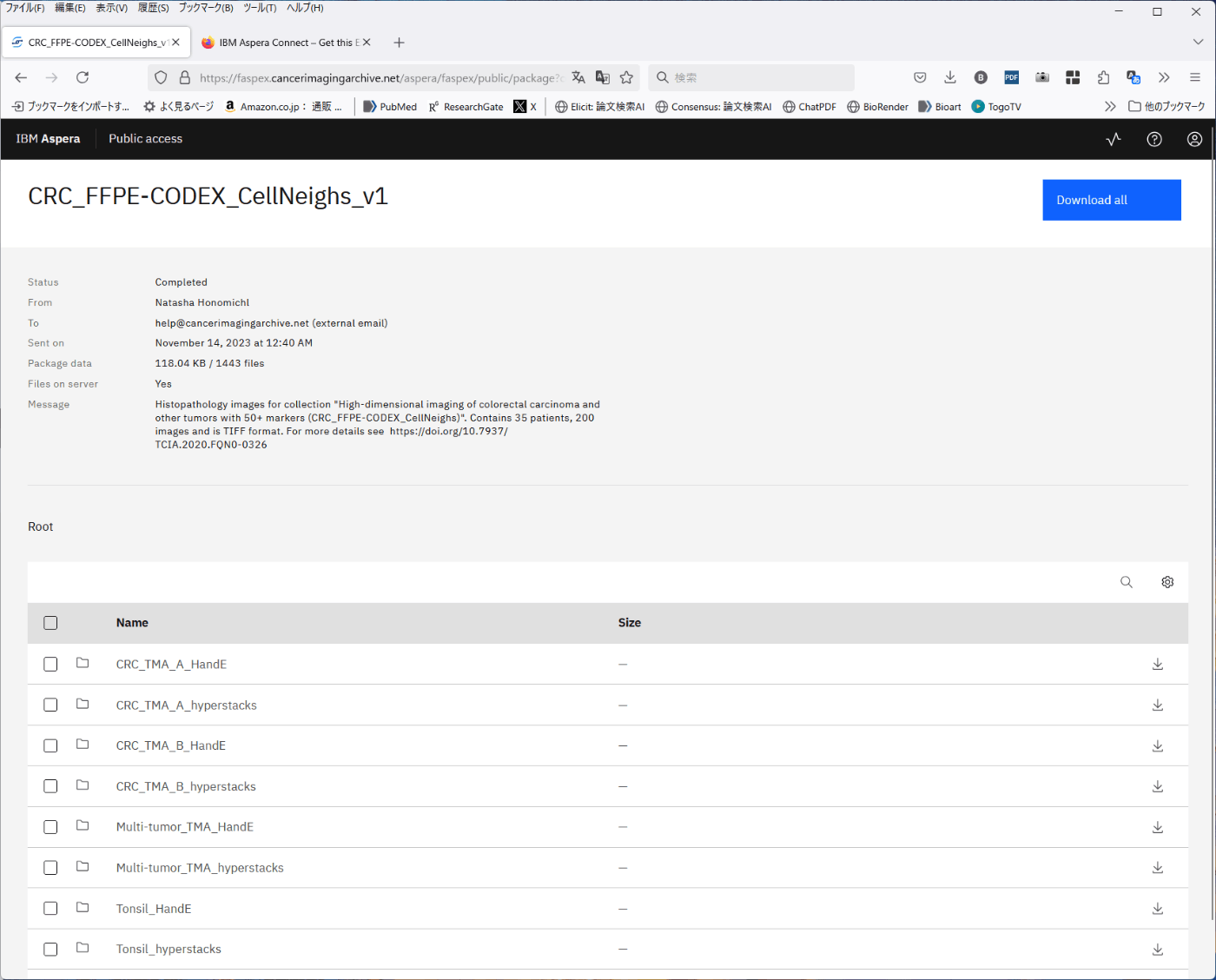
データのダウンロード
Aspera Connectの用意が終わると自動でダウンロードページが開く。「Download all」で全て一括でダウンロードするか、各フォルダのダウンロードアイコンをクリックする。(初回はセキュリティ警告が出る。)
ダウンロード設定
ページ右上のTransfer monitorをクリック。
「Change download location」をクリック。
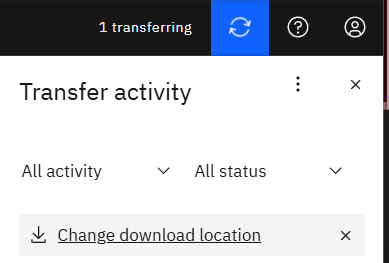
IBM Aspera Connectのウィンドウが立ち上げる。転送タブでダウンロード先を変更可能。
- 正解ラベル:
正解ラベル画像が別途用意されているわけでは無いが、ImageJで画像を開くとCell segmentationラベルがあることがわかる。画像のmetadataとして登録されているみたい。

"Tonsil_hyperstacks\bestFocus\reg001_X01_Y01_Z04.tif"をImageJで開いた画面
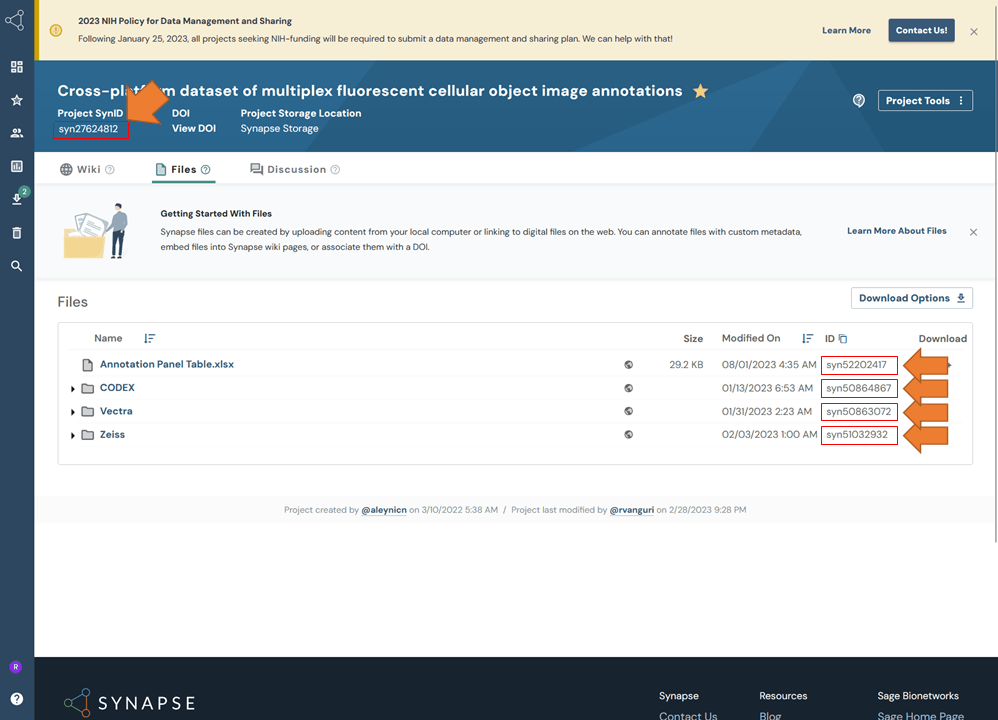
【 Cross-platform dataset of multiplex fluorescent cellular object image annotations 】
- マルチプレックス蛍光画像の大規模なDataset
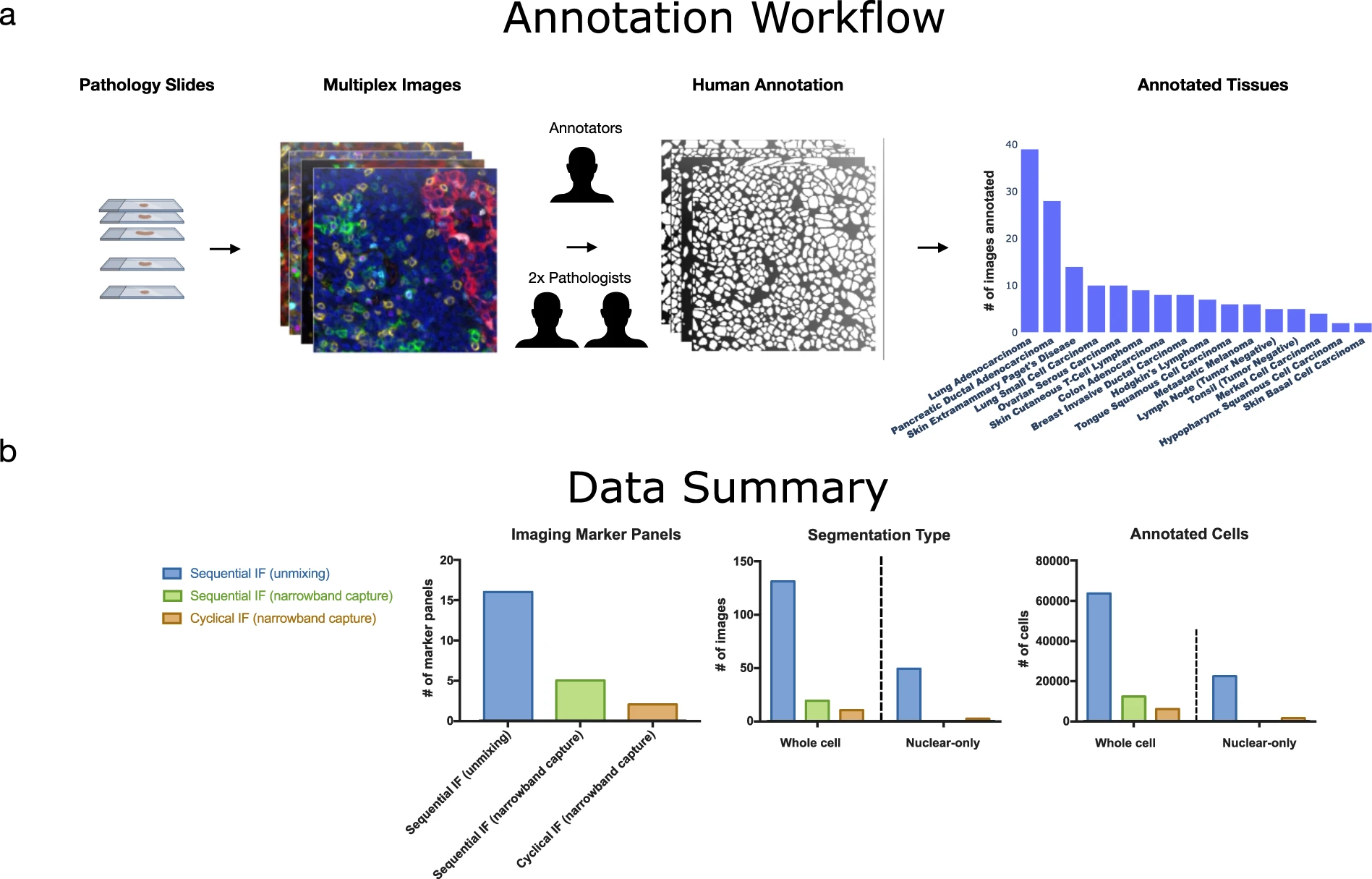
原著 Fig.1 - 原著(Scientific Data, 2023): https://www.nature.com/articles/s41597-023-02108-z
- 細胞核レベルのマスクがあるものと細胞レベルのマスクがあるものがあるとのこと。
- ダウンロード先
Synapse: https://www.synapse.org/Synapse:syn27624812/files/
まずはアカウントを作るところから必要。
Synapse API: https://help.synapse.org/docs/Installing-Synapse-API-Clients.1985249668.html
Synapseからのデータ取得
Synapseからのデータ取得
- Synapseにログイン
- Access Tokenの発行
Dashboardページの左下にアカウント設定のアイコンがある。アイコンをクリック ➔ 「Account Settings」ページを下にスクロールしていって、Personal Access Tokensの項目を探す。
「Manage Personal Access Tokens」をクリック

「Create New Token」をクリック
任意のToken名、Token Permissionを設定。(Token Permissionはよくわからないがとりあえず全部チェックを入れている。)➔ 「Create Token」
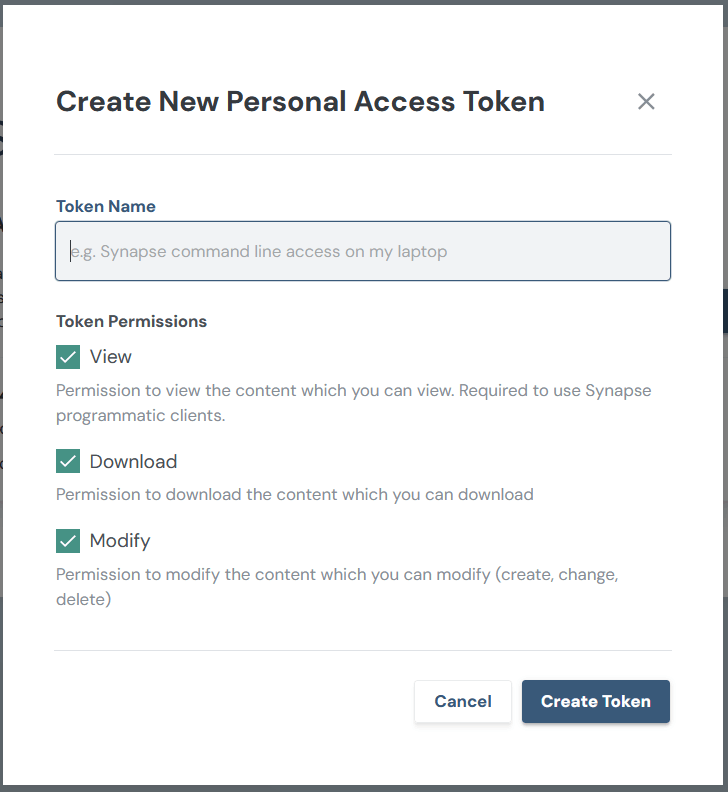
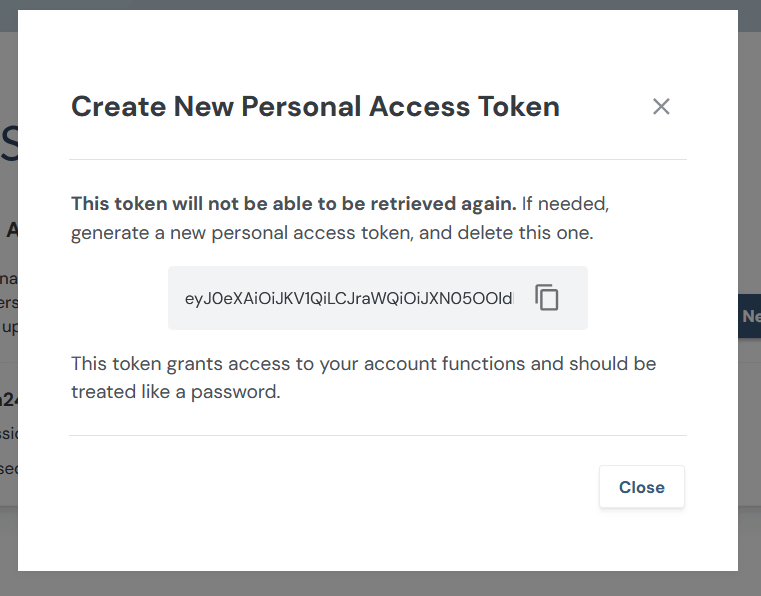
次のページでTokenが発行される。以下では一部しか表示されていないが700文字以上あった。コピーして控えておく。
- SynapseのAPIを取得
Python版、R版がある。ここではPyPiで配布されているものをインストール。
pip install synapseclient openpyxl
Installing Synapse API Clients: https://help.synapse.org/docs/Installing-Synapse-API-Clients.1985249668.html
- アカウントの接続確認
ここではコマンドラインから実行している。(コマンドプロンプトやAnaconda promptなどでsynapseコマンドが呼び出せるものを使用すればよい。)
synapse loginと打つと、usernamemとtokenを打ち込むように指示される。
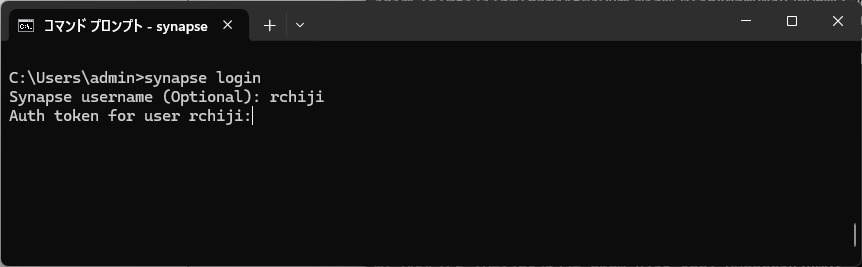
長いtokenをコピペしてEnter。Welcomと出れば接続成功。
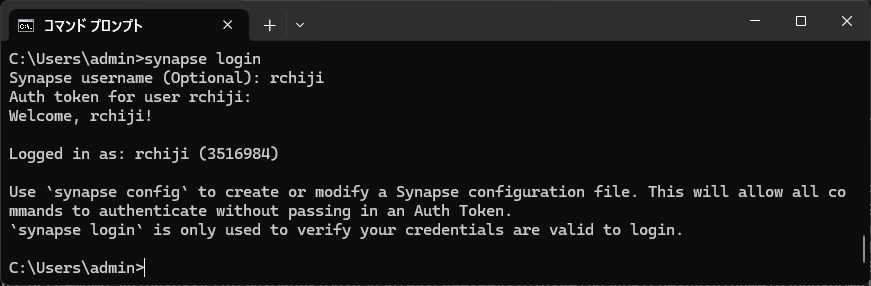
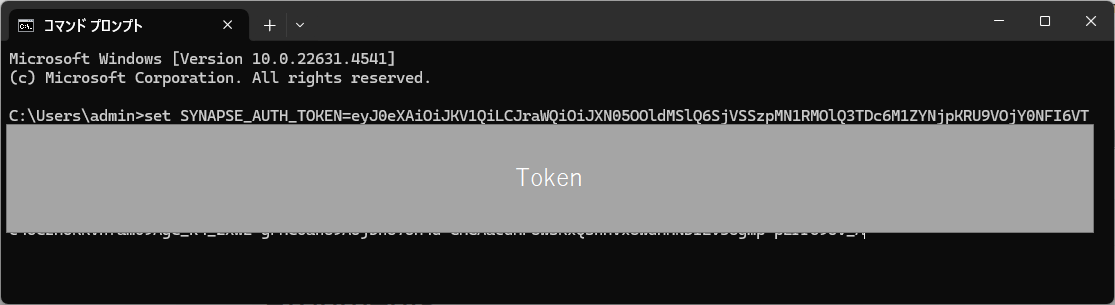
どうやら環境変数にSYNAPSE_AUTH_TOKENという名前でtokenを登録しておけば楽にログインできるみたい。
set SYNAPSE_AUTH_TOKEN=<tokenをここにコピペ>
5. データのダウンロード
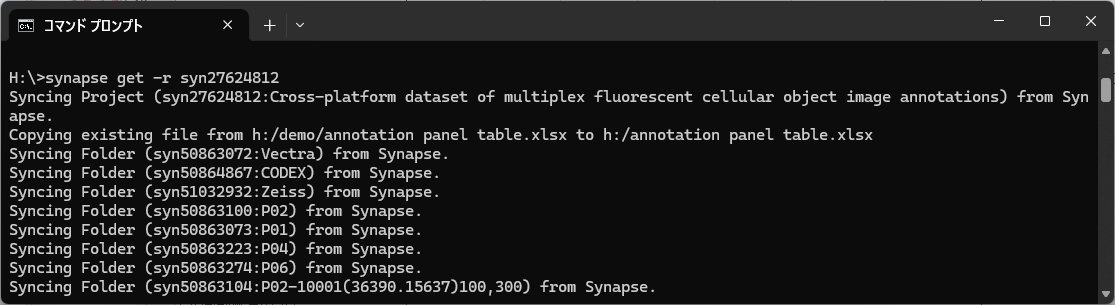
synapse get -r データIDでインストールできる。-rオプションはダウンロード対象がフォルダの場合に必要となる。カレントディレクトリにダウンロードされる。データIDはSynapseの各Projectページで確認できる。ファイル単位にもフォルダ単位にもIDが付与されている。
synapse get -r syn27624812
開始画面
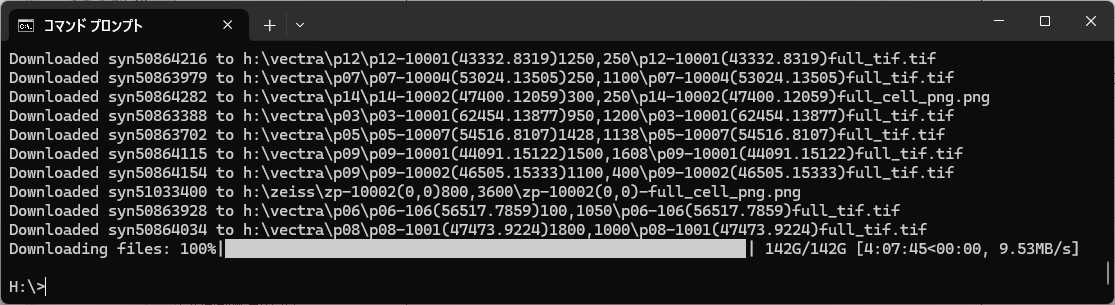
終了画面
# Vectra
synapse get -r syn50863072
# CODEX
synapse get -r syn50864867
# Zeiss
synapse get -r syn51032932
【 Multiplexed Imaging Mass Cytometry of Chemokine Milieus in Metastatic Melanoma 】
- ダウンロード先: https://zenodo.org/records/6004986
CellProfilerのセグメンテーション結果?が入っていた。 - 関連論文: https://www.science.org/doi/10.1126/sciimmunol.abk1692
【 Multiplexed Images of Melanoma Lymph Node Metastases 】
-
CellSighterで使用されているデータ。
CellSighter原著: https://www.nature.com/articles/s41467-023-40066-7
【 MAPS 】
- 40 ch以上のマルチプレックス蛍光画像
- 原著(Nature Communications, 2024.01): https://www.nature.com/articles/s41467-023-44188-w
- Zenodoにデータあり: https://zenodo.org/records/10067010
↓↓ 培養細胞の蛍光顕微鏡画像 ↓↓
【 DynamicNuclearNet 】
-
培養細胞の蛍光顕微鏡画像
-
このDatasetを使った原著: https://www.biorxiv.org/content/10.1101/803205v4.full
Cell segmentationとlive cell imagingの論文 -
ダウンロード先
DeepCellで提供されている。https://datasets.deepcell.org/data -
Datasetの中身:
train/test/valの3つのnpzファイルがダウンロードされる。512*512 pxサイズの16bitグレースケール画像。ラベルはインスタンスラベル画像。
trainデータは4950枚あった。
import numpy as np
data = np.load("train.npz", allow_pickle=True)
X = data["X"] # 顕微鏡画像
y = data["y"] # インスタンスラベル画像
meta = data["meta"] # ファイル名などの情報
- PythonのDeepCellライブラリを使っても訓練用/検証用データが用意できるみたい。 https://deepcell.readthedocs.io/en/master/data-gallery/dynamicnuclearnet.html
【 Cell Image Library 】
-
顕微鏡画像のdata portal。ラベル付き画像用ではないが、多種多様な画像がある。
https://www.cellimagelibrary.org/home -
Project: P2043の培養神経細胞の蛍光顕微鏡画像はラベル画像も用意されている。https://www.cellimagelibrary.org/images/CCDB_6843
【 HPA Cell Image Segmentation Dataset 】
-
培養細胞の蛍光顕微鏡画像。核、微小管、小胞体(+ target protein)のきれいな画像。
-
ダウンロード先:
Zenodoにdepositあり。https://zenodo.org/records/4665863
その中でDPU-Netのモデルもある。核のみのSegmentationモデルと細胞単位のSegmentationモデルがある。
Kaggleコンペでも同じDatasetが使われているっぽい。https://www.kaggle.com/c/hpa-single-cell-image-classification/data?select=train
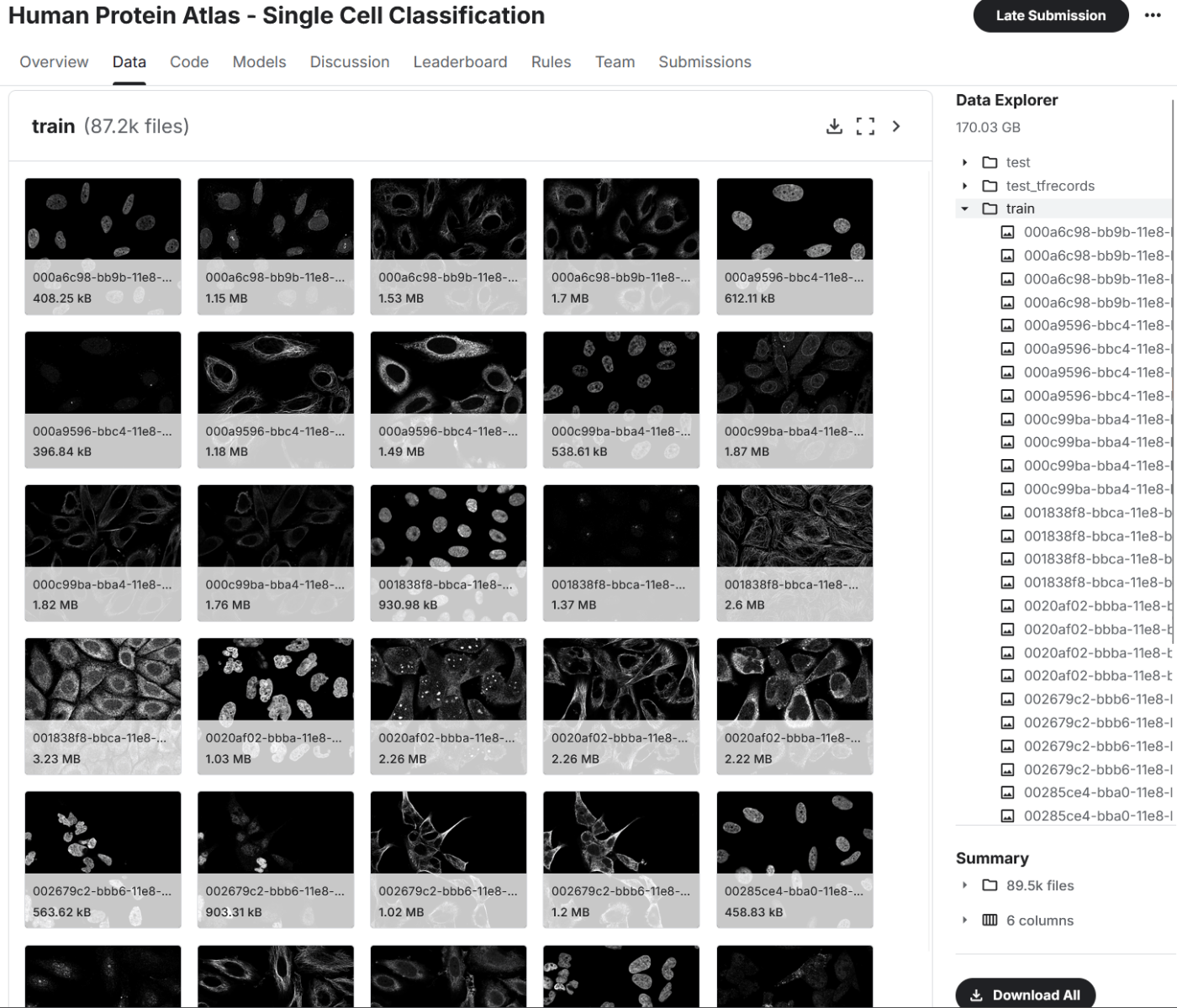
-
このモデルとDatasetを使ったセグメンテーションパッケージがある。
HPA-Cell-Segmentation: https://github.com/CellProfiling/HPA-Cell-Segmentation
紹介: https://qiita.com/Hiroaki-K4/items/298797f1621070664c25
【 BioImage Archive S-BSST265 】
- 原著(Scientific Data, 2020): https://www.nature.com/articles/s41597-020-00608-w
- ダウンロード先:https://www.ebi.ac.uk/biostudies/bioimages/studies/S-BSST265
【 LIVECell 】
Nature Methods, 2021: https://www.nature.com/articles/s41592-021-01249-6
GitHub: https://sartorius-research.github.io/LIVECell/
https://github.com/sartorius-research/LIVECell
- 複数の細胞株の顕微鏡画像
- ダウンロード先: GitHubページ内のリンク
- detectron2の訓練済みモデルや、モデルの使用例が紹介されている。
https://github.com/sartorius-research/LIVECell/tree/main/model
【 A431 】
- 原著(Communications Biology, 2023): https://www.nature.com/articles/s42003-023-04608-5
- ダウンロード先: https://data.mendeley.com/datasets/89s3ymz5wn/1
↓↓ 複数種の顕微鏡画像 ↓↓
病理画像(明視野、蛍光)、培養細胞画像(明視野、蛍光)、電顕画像など、、、複数のモダリティの画像を含むもの。
【 CellPose 】
- CellPoseモデルの訓練に使用されたdataset。
- ダウンロード先: https://www.cellpose.org/dataset
利用規約に同意するとダウンロードできる。 - 培養細胞の蛍光顕微鏡画像が主だが、電顕画像など多様な画像がある。それに対応するインスタンスラベル画像もある。
- 画像サイズはバラバラだが大体512 px辺程度。
【 EmbedSeg datasets 】
EmbedSegというセグメンテーションモデルのGitHubに、使用したdatasetの配布ページがある。明視野画像/蛍光画像とそれに対応したインスタンスラベル画像が配布されている。
2D明視野画像や2D蛍光画像のみならず、3D画像や線虫の画像なども含む。
【 NeurIPS 2022 Cell Segmentation Competition 】
Nature Methods, 2024: https://www.nature.com/articles/s41592-024-02233-6?fromPaywallRec=false#data-availability
-
Grand Challenge: https://neurips22-cellseg.grand-challenge.org/awards/
-
位相差顕微鏡画像、蛍光顕微鏡画像、塗抹標本の明視野画像など、複数モダリティの画像でCell segmentationを行うタスク用のDataset。
-
Google drive、Zenodoなどからダウンロード可能。
-
Top 10 awardのコードが見れる。
https://neurips22-cellseg.grand-challenge.org/awards/

GCのDatasetページより
【 CellSAM 】
bioRxiv (2023.11): https://www.biorxiv.org/content/10.1101/2023.11.17.567630v3
GitHub: https://github.com/vanvalenlab/cellSAM
-
GitHubの「The full dataset used to train CellSAM is available here. 」のリンクからダウンロード可能

GitHubより -
原著で書かれていたデータセット。過去のデータセットを沢山集めている。

原著より抜粋 -
ダウンロードしてみると、npyファイルで格納されていた。
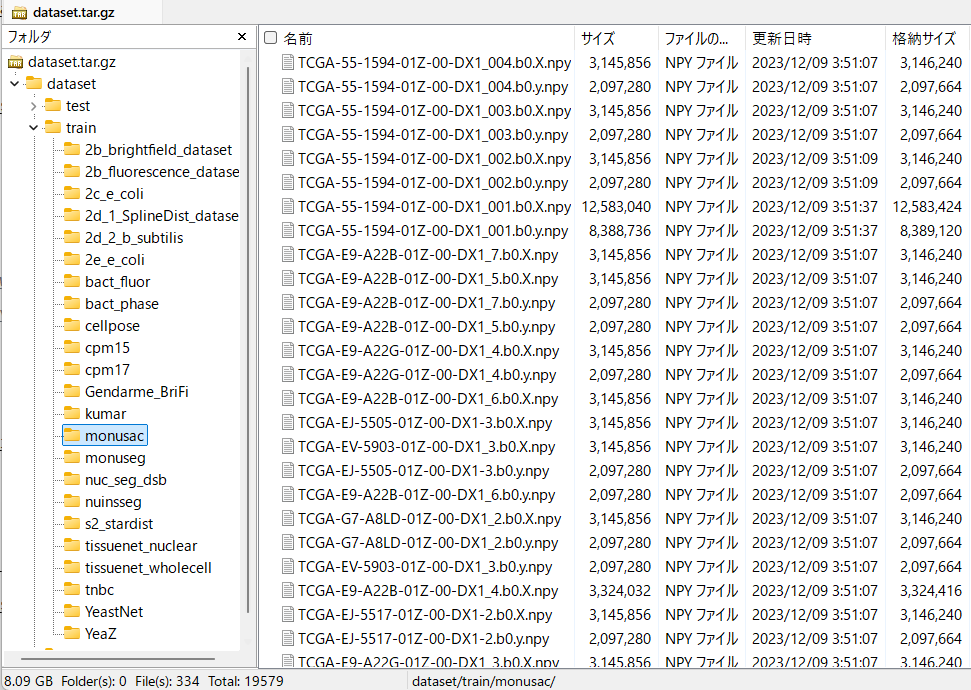
dataset.tar.gzの中身
【 CellBinDB 】
bioRxiv (2024.11): https://zenodo.org/records/14312044
-
蛍光(核染色、膜染色)、HEなど複数の画像タイプで、35の組織由来。
マウスの30組織とヒトの5つの組織。ヒト組織は10x GenomicsのXeniumやVisiumデータ。マウスはStereo-seq。
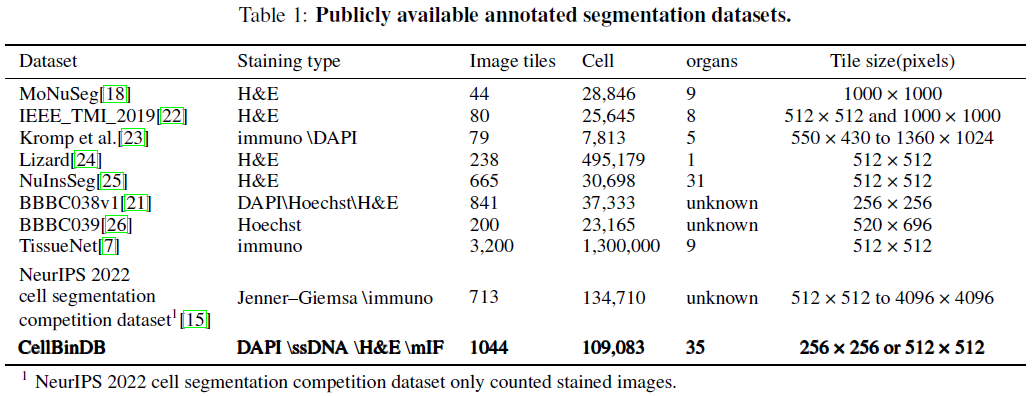
bioRxiv Table 1より -
データセット作りのワークフロー
既存モデル(CellProfiler, MEDIAR, CellPose1, CellPose3, SAM, StarDist, DeepCell)を通して、その結果を人がチェック&修正。それをエキスパートが再チェック。
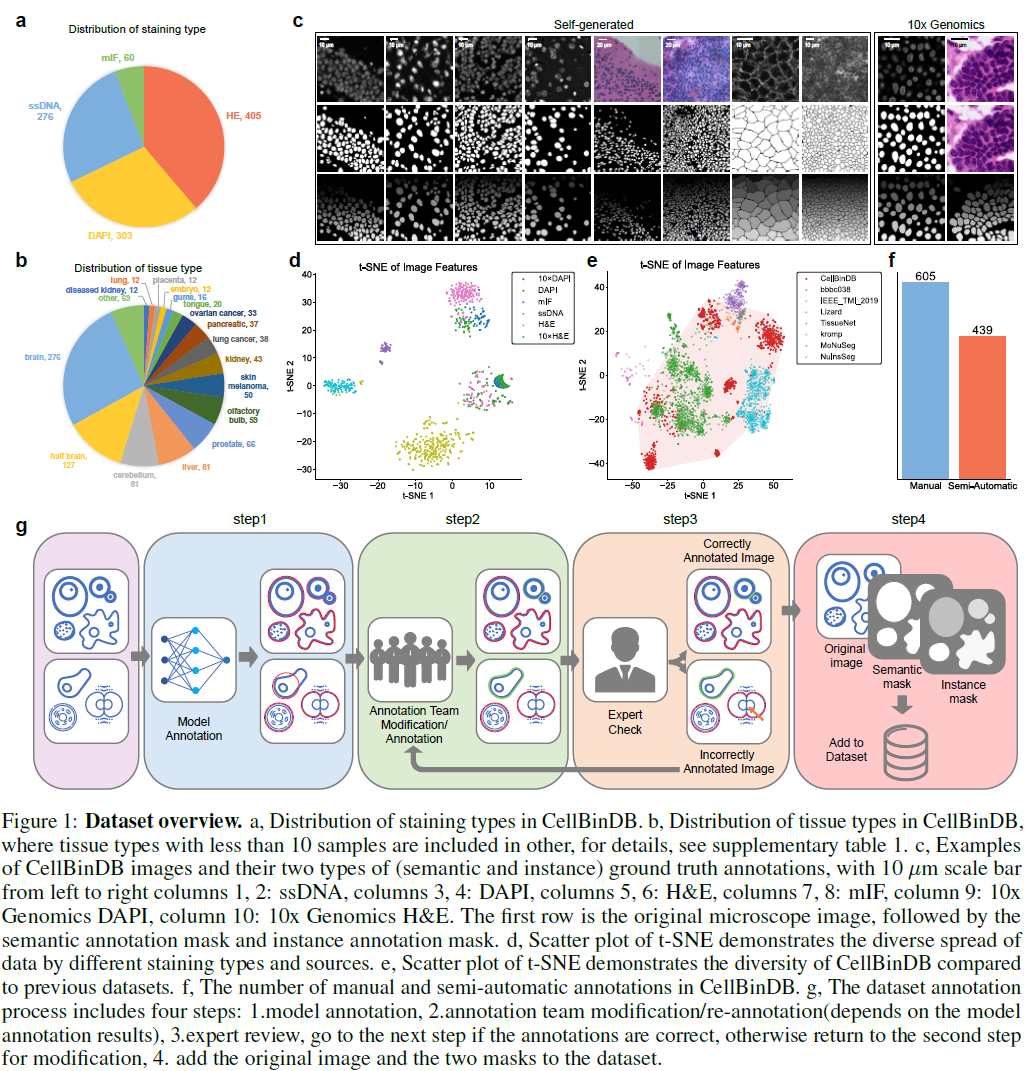
bioRxiv Fig.1より -
ちなみに上記モデルの多くでは、HE画像と膜蛍光画像はgrayscleの明暗逆転画像の方が精度が上がるとのこと。(膜画像も反転したら細胞質が明るく、細胞膜が暗くなる。既存モデルは細胞質の広がりを学習しているモデルが多いので、細胞質が明るい方が精度が上がるっぽい。)
-
ダウンロード先: Zenodoからダウンロード可能。
https://zenodo.org/records/14312044
Zenodoのv2のリンクからはCellBinDB以外のデータセットも配布されている。 https://zenodo.org/records/15110639
↓↓ その他 ↓↓
【 2018 Data Science Bowl 】
- 培養細胞の蛍光顕微鏡画像や明視野の病理画像から細胞核をセグメンテーションするKaggleコンペ
- Kaggleコンペの結果をまとめた論文?
Nature Methods, 2019: https://www.nature.com/articles/s41592-019-0612-7
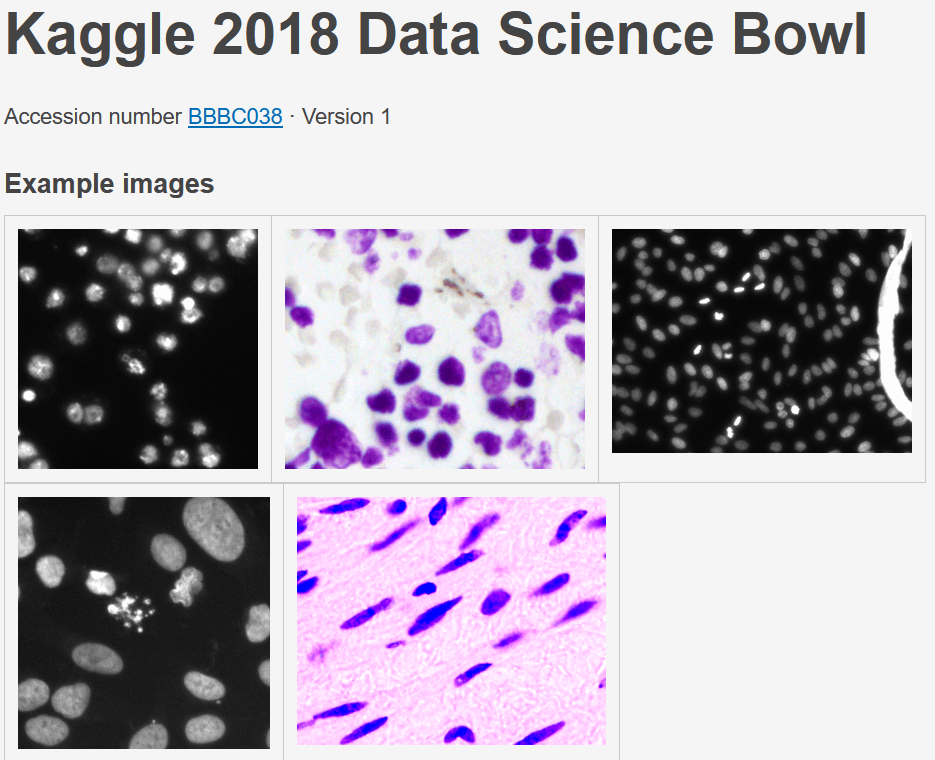
ダウンロード先リンクより
- 「stage1_train.zip」に画像と細胞核マスク画像がある。細胞核マスク画像はインスタンスごとに別の画像ファイルになっている。
以下は正解ラベルとしてはCell segmentationのラベルデータは無いが、訓練/検証に使用できるかもというdataset。
【 Kather100K dataset 】
- human colorectal cancer and healthy tissue
- 10万枚のHE画像
- 正解ラベル:
Adipose (ADI), background (BACK), debris (DEB), lymphocytes (LYM), mucus (MUC), smooth muscle (MUS), normal colon mucosa (NORM), cancer-associated stroma (STR), colorectal adenocarcinoma epithelium (TUM) - ダウンロード先: https://zenodo.org/records/1214456
【 PatchCamelyon (PCam) 】
- リンパ節のHE画像
- 96*96 pxのタイル
- 327680枚
- 正解ラベルは転移有り無しのバイナリラベル
- ダウンロード先: GitHubにGoogle driveリンクと展開コードの記載あり
https://github.com/basveeling/pcam/?tab=readme-ov-file#download
【 Breast Cancer Semantic Segmentation (BCSS) 】
- Grand Challengeのdataset。がん領域のSemantic segmentationタスク
- TCGAのHE画像
- ダウンロード先: https://github.com/PathologyDataScience/BCSS
GitHubページにGoogle driveのリンクがある。
【 BCCD 】
- 血液塗抹標本
- 白血球、赤血球、血小板の正解ラベル付きのバウンディングボックス
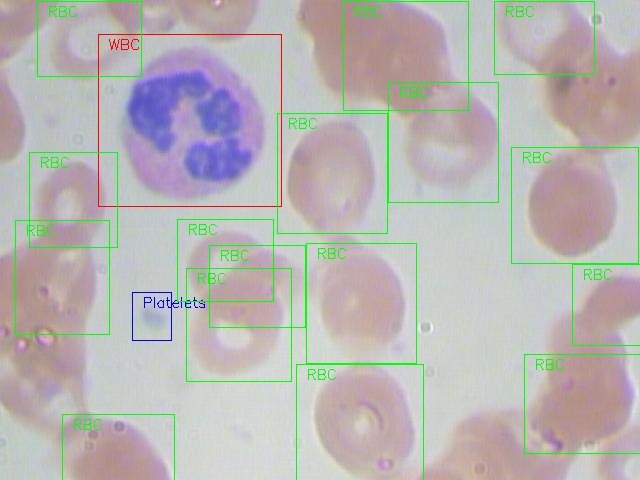
- GitHub: https://github.com/Shenggan/BCCD_Dataset
- ダウンロード先:
GitHubページ内のdownloadリンク。
【 Kaggle Datasets 】
Blood Cells Image Dataset
https://www.kaggle.com/datasets/unclesamulus/blood-cells-image-dataset
血液塗抹標本の細胞分類に使えそう。
Blood Cells Cancer (ALL) dataset
https://www.kaggle.com/datasets/mohammadamireshraghi/blood-cell-cancer-all-4class
血液塗抹標本から悪性リンパ腫細胞を見つける。
Blood Cell Segmentation Dataset
- Segmentation maskあり。
- 個々の細胞のマスクが分離しているのでインスタンスラベルへ簡単に変換可能
https://www.kaggle.com/datasets/jeetblahiri/bccd-dataset-with-mask

Discussion