【Plot】Pheatmapの基本の使い方
私が講習用に用意していた資料。pheatmapの日本語資料がそんなに多くないので公開しておく。
install.packages("pheatmap")
library(pheatmap)
【超基本の使い方】
数値だけのmatrixをpheatmap()機能に渡すだけ。文字の列が残っているとエラーが出る。
例えばirisデータでは5列目が文字なので、1から4列目を使う。
pheatmap(iris[,1:4])

matrixを指定するだけで、値の範囲 (最小値~最大値)に色が割り当てられ サンプルや列は階層的クラスタリングに基づいて並び替えられる。
【引数一覧】
引数名、デフォルト値、解説の順で説明する。量が多いので必要な箇所だけ辞書のように見て頂ければよい。
基本部分
-
mat:
plotに用いる数字型の行列 (matrix)。データフレームでも入力に使用できるが、数字以外の列があるとエラーがでる。なのでサンプル名などの情報は行列名に入れておく。
例えば行名に遺伝子、列名にサンプルなど。
-
color:
colorRampPalette(rev(brewer.pal(n = 7, name ="RdYlBu")))(100)
色情報のベクトル。デフォルトではRColorBrewerパッケージのRdYlBuパレットが100色分用意されている。
-
breaks: NA
色を割り当てる値のベクトル。color=引数はデフォルトで100色用意しているので、100個の値のベクトルを指定すれば、各値が各色に対応する。
何も指定しなければ、matrix中の最小値/最大値から色を割り当てる値が自動で決定される。
-
scale:
"none"
入力データを中心化 (平均を0)、スケーリング (標準偏差を1)にする。"row"で行ごと、"column"で列ごとに正規化が行われる。
クラスタリング
クラスタリング
-
cluster_rows/cluster_cols:
TRUE
階層的クラスタリングを行単位、列単位で行うかどうかTRUE/FALSEで指定。デフォルトでTrueなのでクラスタリングさせない場合はFALSEを明示。
※ データにNAがある場合はヒートマップ化はできてもクラスタリングはできないのでFALSEにする。データが1行or1列の場合もFALSEにする。
-
clustering_distance_rows/clustering_distance_cols:
"euclidean"
階層的クラスタリングに使用する距離行列の算出方法。データ間の類似度の指標。デフォルトではユークリッド距離。"correlation"を指定するとピアソン相関が使用される。
-
clustering_method:
"complete"
階層的クラスタリングで使用するアルゴリズム。内部でhclustパッケージを使用しているので、hclustで指定できるmethodが使用できる。
"complete": 最遠隣法。"single": 最近隣法。"average": 群平均法。"centroid": 重心法。"median": メディアン法。"ward.D"/"ward.D2": ウォード法 (ユークリッド距離をそのまま使用するならward.D, 平方ユークリッド距離を使うならward.D2)
-
clustering_callback:
identity2
使用することはまずない。使用例:https://stackoverflow.com/questions/54762405/pheatmap-re-order-leaves-in-dendogram
-
kmeans_k: NA
k-meansクラスタリングする際のクラスター数。非階層的クラスタリング手法。
-
cutree_rows/cutree_cols:
0
階層的クラスタリングをいくつのクラスターに分割して表示するか。hclustパッケージのcutree()機能を使っている。
plotのグラフィカルパラメーター
plotのグラフィカルパラメーター
-
show_rownames/show_colnames:
TRUE
ヒートマップに行名、列名を表示させるかどうか
-
main: NA
ヒートマップのタイトルに表示する文字
-
fontsize:
10:
全体的なフォントサイズ
-
fontsize_row/fontsize_col:
fontsize
行名、列名のフォントサイズ。指定が無ければfontsize=引数に従う。
-
angle_col:
270
列名の表示角度 (0, 45, 90, 270, 315)
-
border_color:
"grey60"
ヒートマップのセル境界の色。データが少ない時しか適応されない。
-
cellwidth/cellheight: NA
ヒートマップのセルの幅、高さを固定する場合に指定する。
-
treeheight_row/treeheight_col:
50
デンドログラム(樹形図)の表示サイズ
-
legend:
TRUE
凡例を表示するかどうか
-
legend_breaks: NA
凡例のbreakpointsを指定するベクトル
-
legend_labels: NA
凡例のbreakpointsに表示するラベルを指定するベクトル
-
gaps_row/gaps_col: NULL
ヒートマップを分割した時の分割幅。例えばデータが100個あるとき、c(5,20,40,80)のようにgapを入れたい箇所を指定する。
-
labels_row/labels_col: NA
入力matrixの行名列名を使用せずにカスタムでラベル付けする用のベクトル
-
filename: NA
ヒートマップを書き出す先のファイルパス。拡張子はpng,pdf,tiff,bmp,jpegに対応
-
width/height: NA
ヒートマップ全体の幅、高さ
-
silent:
FALSE
ヒートマップをRのViewerに表示をさせるかどうか。
-
na_col:
"#DDDDDD"
NAが入っている場合のNAセルの色
ヒートマップアノテーション
ヒートマップアノテーション
-
annotation_row/annotation_col: NA
ヒートマップにアノテーション付けする用のデータフレーム
-
annotation_colors: NA
ヒートマップのアノテーションの色を指定するためのリスト
-
annotation_legend:
TRUE
アノテーションの凡例を表示するかどうか
-
annotation_names_row/annotation_names_col:
TRUE
アノテーションの名前を表示するかどうか
-
drop_levels:
TRUE
ヒートマップに使用しない因子を凡例に残すかどうか
ヒートマップセルへの値表示
ヒートマップセルへの値表示
-
display_numbers:
FALSE
ヒートマップの各セルに値を表示させかどうかを指定。入力matrixと同じ行名列名のデータフレームを指定して、文字を表示することも可能。
-
number_format:
"%.2f"
セルに表示するラベルのフォーマット。表示小数点など。
-
number_color:
"grey30"
セルに表示するラベルの色
-
fontsize_number:
0.8
セルに表示するラベルのフォントサイズ
【Plotの色】
デフォルトのヒートマップの色は100色の色コードベクトルで、RColorBrewerパッケージのパレットから作られている。
library(RColorBrewer)
colorRampPalette(rev(brewer.pal(n = 7, name ="RdYlBu")))(100)

デフォルトの色ベクトル
colorRampPalette()機能は指定した色ベクトルからグラディエントな色ベクトルを作る機能を返す。得られた機能に欲しい色数を指定すると、その数だけの色コードベクトルが得られる。
例えば次のように青白赤の3色を100色に拡張できる。
pheatmap(iris[1:4],
show_rownames = F,
angle_col = 45,
color = colorRampPalette(c("blue","white","red"))(n=100)
)

与える色ベクトルを少なくすると、表現力が落ちてしまう。一方でこの方がビニングされてわかりやすく解釈できることもある。
pheatmap(iris[1:4],
show_rownames = F,
angle_col = 45,
color = colorRampPalette(c("blue","white","red"))(n=10)
)

10色だけ与えた場合
【データのスケーリング】
irisデータの場合、項目ごとに値の範囲が異なっており、同じスケールで表示させるとデータが見づらくなる。scale=オプションを使用すると、各列(もしくは各行)の平均を0, SDを1にスケーリングして表示してくれる。
pheatmap(iris[1:4],
show_rownames = F,
angle_col = 45,
scale = "column")

各列でデータのscale
項目(列)でscaleすると1つの項目内でのサンプル(行)の違いが見やすくなる。
pheatmap(iris[1:4],
show_rownames = F,
angle_col = 45,
scale = "column")

各行でデータのscale
1サンプル(行)の中でscaleすると、どの項目(列)が高いかという意義になる。
irisデータでは列によって値の範囲が大きく異なるので、その差が鮮明になるが、サンプル間の違いが潰れてしまう。
【ヒートマップアノテーション】
各サンプル、各項目に追加の情報があれば、それをヒートマップの左側や上部に表示することができる。これには数値だけでなく文字情報も使用できる。
アノテーション情報はdata.frameを別途用意する。サンプル(行)のアノテーション用には、行名にサンプル名、列にアノテーション項目を入れたdata.frameを用意する。
項目(列)のアノテーション用にも行名に項目名、列にアノテーション項目を入れたdata.frameを用意する。
※ 行の名前が無い(ただの数字の行番号)の場合はうまくいかないので、matrixの行名、列名を事前に定義しておく。
ここではmatrixの行名を次のように「ID〇〇」としておく。
demo <- iris[1:4]
rownames(demo) <- paste0("ID", rownames(demo))
そして同じ行名を持たせたアノテーション情報data.frameを用意する。ここではirisの5列目のSpecies列を使用する。
anno <- data.frame(
row.names = rownames(demo),
Species = iris$Species
)

行のアノテーション用data.frame
このdata.frameをannotation_row=引数に指定する。
pheatmap(demo,
show_rownames = F,
angle_col = 45,
scale = "column",
annotation_row = anno
)
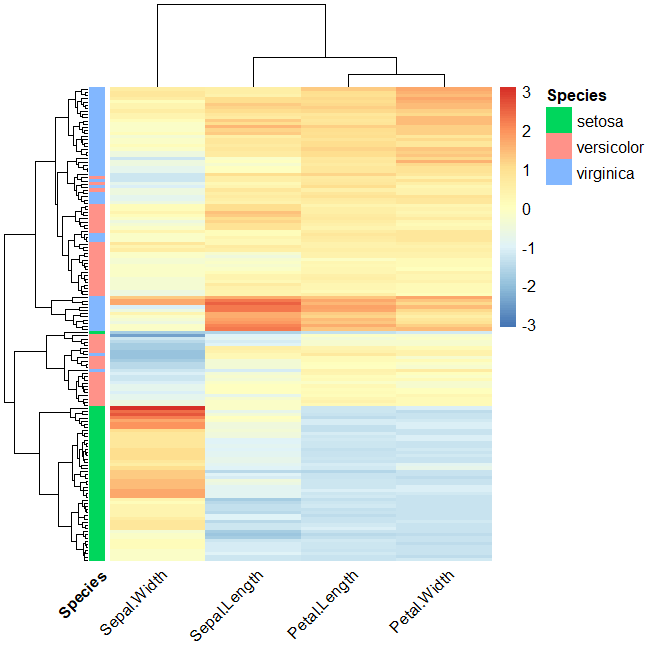
サンプルのSpecies情報をアノテーションとして表示
列にもアノテーション情報を追加してみる。そのために、列用に新たにdata.frameを用意する。
アノテーションdata.frame行名にはmatrixの列名を配置する。
anno_col <- data.frame(
row.names = colnames(demo),
var1=c("Sepal","Sepal","Petal","Petal"),
var2=c("Length","Width","Length","Width")
)

列のアノテーション用data.frame
これをannotation_col=引数に指定する。
pheatmap(demo,
show_rownames = F,
angle_col = 45,
scale = "column",
annotation_row = anno,
annotation_col = anno_col
)

アノテーションの色
アノテーションの各色を指定するには色情報をまとめたリストを作成する必要がある。
リストの名前属性にアノテーション名 (上の例だと、Species, var1, var2)、要素に名前付き色ベクトルを入れる。行用、列用のアノテーション情報をまとめて指定する。
文字/factorの場合
まず例として、Speciesの色を指定してみる。必要数だけの色のベクトルを作成し、その名前属性に対応する要素を入れる。要素が色情報、名前属性が水準となる。
# 色のベクトルを作成
species_color <- c("red","blue","green")
# 対応する水準をベクトルの名前属性に入れる。
names(species_color) <- c("setosa", "versicolor", "virginica")

アノテーション用の色ベクトル
そしてこれをリストにまとめる。リストの名前属性には対応するアノテーション名を入れる。
anno_colors <- list(Species = species_color)
annotation_colors=引数にアノテーション色ベクトルリストを渡す。
pheatmap(demo,
show_rownames = F,
angle_col = 45,
scale = "column",
annotation_row = anno,
annotation_col = anno_col,
annotation_colors = anno_colors
)

列のアノテーション色情報も同じリストに入れて指定する。
anno_colors$var1 <- c("Petal"="white", "Sepal"="black")
anno_colors$var2 <- c("Length"="gray", "Width"="lightgray")

pheatmap(demo,
show_rownames = F,
angle_col = 45,
scale = "column",
annotation_row = anno,
annotation_col = anno_col,
annotation_colors = anno_colors
)

数値の場合
数値の場合は、名前属性無しで色ベクトルを用意するだけで良い。
anno$group <- rep(1:5, each=30)
anno_colors$group <- c("white","lightgreen","green","darkgreen","black")
pheatmap(demo,
show_rownames = F,
angle_col = 45,
scale = "column",
annotation_row = anno,
annotation_colors = anno_colors
)

グラジエントな色ベクトルであればcolorRampPalette()機能をうまく使えば簡単に用意できる。
# デモ用にダミーの連続値を作成
anno$dummy <- sample(1:100, size=150, replace = TRUE)
anno_colors$dummy <- colorRampPalette(c("blue","white","red"))(100)
pheatmap(demo,
show_rownames = F,
angle_col = 45,
scale = "column",
annotation_row = anno,
annotation_colors = anno_colors
)

【階層的クラスタリング】
デフォルトで行と列それぞれで階層的クラスタリングが行われる。内部では、dist()機能でデータ間のユークリッド距離を基にした距離行列作成、hclust()による最遠隣法の階層的クラスタリングが使われている。データ量が非常に大きい場合は、階層的クラスタリングに時間がかかる。
クラスタリング手法
クラスタリング手法を変えるとクラスタリング結果が明らかに変わることがある。どの手法が正しいとかは無いので好きなものを使用すれば良い。
pheatmap(iris[1:4],
show_rownames = F,
angle_col = 45,
scale = "column",
clustering_method = "average")

average
pheatmap(iris[1:4],
show_rownames = F,
angle_col = 45,
scale = "column",
clustering_method = "ward.D")

ward.D
pheatmap(iris[1:4],
show_rownames = F,
angle_col = 45,
scale = "column",
clustering_method = "ward.D2")

ward.D2
階層的クラスタリングに基づいたplotの分割
階層的クラスタリングは各サンプル、各項目の類似度から樹形図を作成しており、各線を辿ると1つのデータに辿り着く。つまりクラスターはデータの数だけ作成できる。
これを指定したクラスター数で樹形図を切断することで、複数のデータを含むクラスターにまとめることができる。
pheatmap(iris[1:4],
show_rownames = F,
angle_col = 45,
scale = "column",
clustering_method = "ward.D2",
cutree_rows = 3
)

pheatmap(iris[1:4],
show_rownames = F,
angle_col = 45,
scale = "column",
clustering_method = "ward.D2",
cutree_rows = 5
)

サンプルだけでなく、項目も同様にクラスター分割できる。
pheatmap(iris[1:4],
show_rownames = F,
angle_col = 45,
scale = "column",
clustering_method = "ward.D2",
cutree_rows = 5,
cutree_cols = 2)

ヒートマップに表示されたサンプル順/項目順を取得
ヒートマップは大量の情報を扱うことが多いため、行名・列名を非表示にすることも多い。ただし、階層的クラスタリングに基づいて並び替えられた後のサンプル名・項目名を確認できないのは困る。
この情報を得るにはpheatmap()の出力を一旦オブジェクトに保存し、そのオブジェクト(リスト型)内の階層的クラスタリング情報にアクセスする。
# デモ用にわかりやすくするため行名をつけたものを使用。
demo <- iris[1:4]
rownames(demo) <- paste0("ID", rownames(iris))
# pheatmapの結果をオブジェクトpに保存
p <- pheatmap(demo,
show_rownames = F,
angle_col = 45,
scale = "column",
clustering_method = "ward.D2",
silent = T
)

pheatmapオブジェクト
この「tree_col」「tree_row」の箇所にそれぞれ列の階層的クラスタリング情報、行の階層的クラスタリング情報が保持されている。

pheatmapオブジェクト内の階層的クラスタリング情報
さらにtree_row$labelsにはmatrixの行名、tree_row$orderにはplotに表示された行順が保持されている。

行名とその表示順
つまり行名のindexにorder情報を入れれば、ヒートマップの並び順で行名が得られる。
p$tree_row$labels[p$tree_row$order]

行名をヒートマップの表示順に並び替え
各データが属するクラスター番号を取得
クラスター分割した時に、どのサンプルがどのクラスターなのかを確認する方法を紹介する。
pheatmapの出力を一旦オブジェクトに保存し、そのオブジェクト(リスト型)内の階層的クラスタリング情報を取得し、cutree()機能でクラスター分割を行う。
cutree(p$tree_row,k = 5)
各サンプルのクラスター番号がベクトルで得られる。名前属性にはmatrixの行名が入っている。
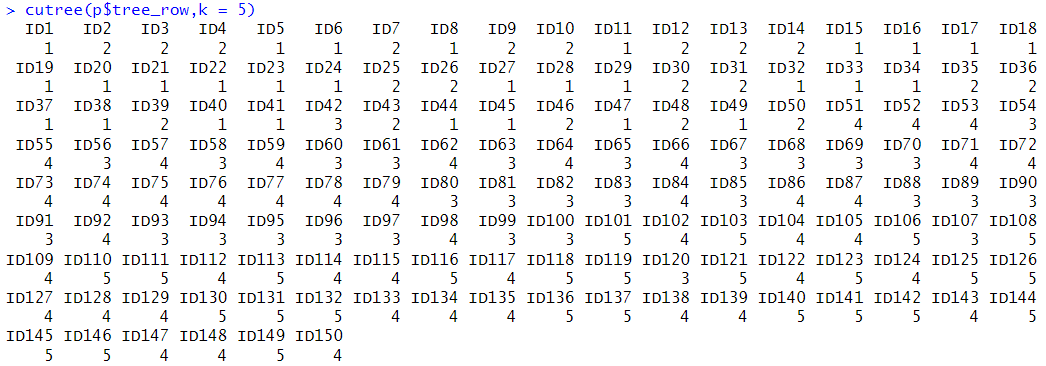
5つにクラスター分割した場合の、各サンプルが属するクラスター
pheatmapの表示順で並び替えてやれば同じクラスター番号でサンプルが並んでいることが確認できる。
cluster <- cutree(p$tree_row,k = 5)
cluster[p$tree_row$order]
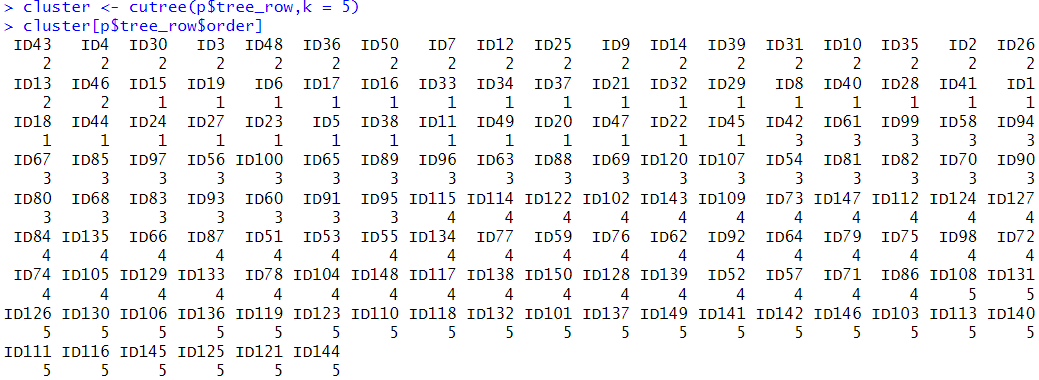
ヒートマップでの表示順とクラスター番号
※ pheatmapの表示順はクラスター番号の若い順という訳ではないことに注意しておく。
クラスター番号をアノテーションとしてヒートマップに表示
サンプルとクラスター番号の対応情報だけ得られても視認しづらいので、ヒートマップアノテーションとして表示させる例を紹介する。
demo <- iris[1:4]
rownames(demo) <- paste0("ID", rownames(iris))
p <- pheatmap(demo,
show_rownames = F,
angle_col = 45,
scale = "column",
clustering_method = "ward.D2",
silent = T
)
anno <- data.frame(
row.names = rownames(demo),
# クラスター番号は数字。連続値として扱われないように文字列に変換。
cluster = as.character(cutree(p$tree_row, k = 5)[rownames(demo)])
)
pheatmap(demo,
show_rownames = F,
angle_col = 45,
scale = "column",
clustering_method = "ward.D2",
cutree_rows = 5,
annotation_row = anno
)

【K-meansクラスタリング】
kmeans_k=引数にクラスター数を指定するだけでK-meansクラスタリングが実行できる。この方法は行にしか適応されないので、列に実行したければt()でmatrixを転置してから渡す。
※ K-meansはやる度に結果が変わる。
pheatmap(iris[1:4],
angle_col = 45,
kmeans_k = 5
)

K-meansの結果もpheatmap()の出力をオブジェクトに保存するとアクセスできる。
p2 <- pheatmap(iris[1:4],
angle_col = 45,
kmeans_k = 5,
)
p2$kmeans

【Tips】
行列を入れ替え
t()関数ででmatrixを転置する。その際に、引数のrowとcolも全て逆になるので注意。
pheatmap(t(demo),
show_colnames = F,
angle_col = 45,
scale = "row",
annotation_col = anno,
annotation_row = anno_col,
annotation_colors = anno_colors
)

1列or1行のデータを可視化するとき
デフォルトで階層的クラスタリングが実行されるが、1行or1列しか無ければ階層的クラスタリングができないためエラーが出る。

hclust(d, method = method) でエラー:
クラスタリングすべき 2 以上のオブジェクトが必要です
階層的クラスタリングが行われないように、cluster_row=/cluster_cols=をFALSEにしてやると可視化可能。
pheatmap(iris[,1,drop=FALSE],
cluster_cols = FALSE,
show_rownames = F
)

データにNAが含まれるとき
一部のNAはエラー無く進む。NAの箇所は灰色で表示される。
demo2 <- demo
demo2[c(1,5,10,25,30,50,100),c(2,4)] <- NA
pheatmap(demo2)
しかし、あるサンプルで全項目がNAの場合はエラーが出る。
demo3 <- demo
demo3[c(1,10,100),] <- NA
pheatmap(demo3)
Error in hclust(d, method = method): 外部関数の呼び出し (引数 10) 中に NA/NaN/Inf があります
これも階層的クラスタリングが行えないことに対するエラーなので階層的クラスタリングをFALSEにすると可視化可能。
pheatmap(demo3, cluster_rows = F)

外れ値に色を引っ張られないようにする
ヒートマップにマークを表示
Legendなどplotの編集
ComplexHeatmapパッケージを使ってpheatmap()機能を呼ぶことができる。このやり方ではComplexHeatmapのplot編集コマンドが使えるようになる。
Discussion