RDKit: 空白のmolオブジェクトを返す
はじめに
RDKitで、分子をグリッドに配置して構造描画する際、見た目を整えるために、空白を入れたくなりました。空白のmolオブジェクトを作ろうとしたのですが、どうもRDKitのバージョンによって、与えるSMILESの内容が違うようなので、まとめました。
概要
SMILESとして ' ' または '' を与え、Chem.MolFromSMILES()でmolオブジェクトにすると空白のmolオブジェクトが得られたが、RDKitのバージョンにより挙動が異なる。可能な範囲での検証結果を表1にまとめた。
SMILESからmolオブジェクトへ変換する代わりに、SMILESが空白の場合にNoneを与える構文の方が良いと思うが、意図通り動作する場合と、Kernel Restartingとなってプログラムが停止する場合があり、動作に不安があったため、種々試行をする過程で、表1の違いに気が付いた。
内容
1. 状況説明
Jupyter notebook上で、RDKitを用い、原料と生成物を並べて描画したいと思いました。
反応の事例として、
酢酸 + エタノール -> 酢酸エチル (eq1)
4-ヒドロキシブタン酸 -> γ-ブチロラクトン (eq2)
を選びました。副生する水は省略します。
eq1では、二分子が分子間で反応して生成物を与えていますが、eq2は一分子の分子内反応です。
従って、以下のスクリプトでは、二つの反応の生成物の場所(列に相当)がずれてしまいます(図1:図中青字は加筆)。
from rdkit import Chem
from rdkit.Chem import Draw
r11 = Chem.MolFromSmiles('CC(=O)O')
r12 = Chem.MolFromSmiles('CCO')p1 = Chem.MolFromSmiles('CC(=O)OCC')r21 = Chem.MolFromSmiles('OCCCC(=O)O')
p2 = Chem.MolFromSmiles('C1(=O)CCCO1')
eq1a = [r11, r12, p1]
eq2a = [r21, p2]
Draw.MolsToGridImage(eq1a,molsPerRow=3, subImgSize=(300,300))
Draw.MolsToGridImage(eq2a,molsPerRow=3, subImgSize=(300,300))
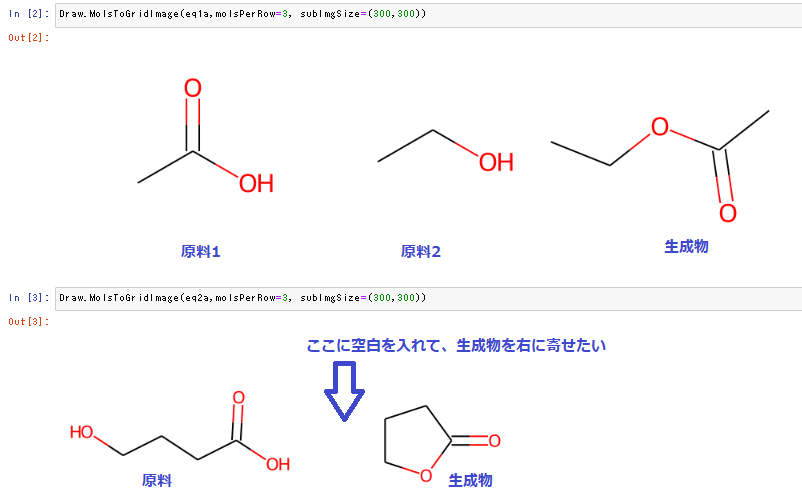
図1. 生成物が位置ずれした図。図中青字は加筆。
2. ダミーのSMILESを作って検証
チュートリアル等に情報が見当たらなかったため、eq2にもダミーのSMILESを入れ、空のmolオブジェクトを返してやればいいのではないか、と考えました。
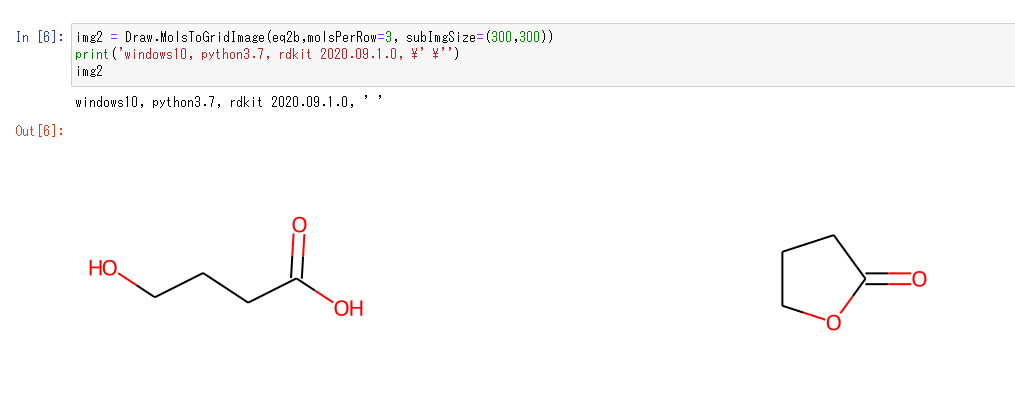
「空の文字列をSMILESとして与えればいいだろう(直観)」、ということで、' '(eq2b: シングルクオーテーションの間にスペースを入れる)と、''(eq3b: シングルクオーテーションの間に何も入れない)のどちらかだろう、と思って試したところ、最初に試した環境では、' '(シングルクオーテーションの間にスペースを入れる)で意図通りの描画ができました(図2)。 一方、 ''(シングルクオーテーションの間に何も入れない)では、他の原料、生成物含め、何も描画されませんでした。
検証環境: windows10, python3.7, rdkit 2020.09.1.0
r21 = Chem.MolFromSmiles('OCCCC(=O)O')
r22 = Chem.MolFromSmiles(' ')
p2 = Chem.MolFromSmiles('C1(=O)CCCO1')
r31 = Chem.MolFromSmiles('OCCCC(=O)O')
r32 = Chem.MolFromSmiles('')
p3 = Chem.MolFromSmiles('C1(=O)CCCO1')
eq2b = [r21, r22, p2]
eq3b = [r31, r32, p3]
img2 = Draw.MolsToGridImage(eq2b,molsPerRow=3, subImgSize=(300,300))
print('windows10, python3.7, rdkit 2020.09.1.0, \' \'')
img2
img3 = Draw.MolsToGridImage(eq3bmolsPerRow=3, subImgSize=(300,300))
print('windows10, python3.7, rdkit 2020.09.1.0, \'\'')
img3
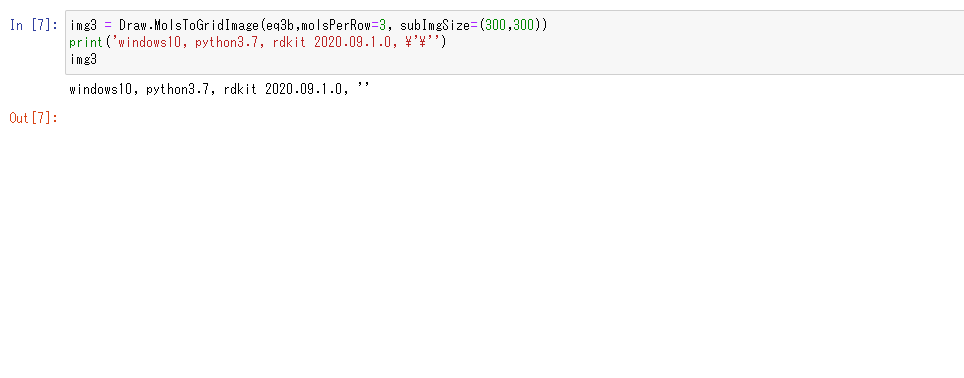
図2. 最初の検証環境。RDkit 2020.09.1.0を使用
3. 二つめの環境での検証
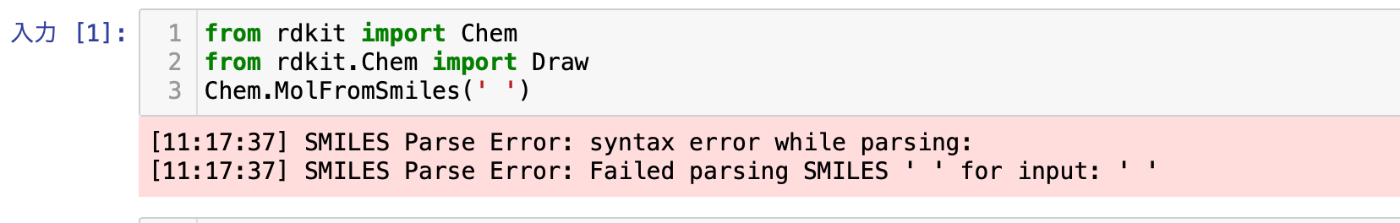
ところが、このnotebookを、別のPCで使ったところ、どうも挙動が異なりました。例えば、SMILESを' '(シングルクオーテーションの間にスペースを入れる)で与えると下記のような、error messageが出ます。
検証環境: macOS11.6, python3.8, rdkit 2022.03.02
Chem.MolFromSmiles(' ')
一方で、SMILESを''(シングルクオーテーションの間に何も入れない)で与えても、
Chem.MolFromSmiles('')
error messageは出ませんでした。
項目2.と同じスクリプトを走らせたところ、eq2bのみ、やはりerror messageが出るものの、どちらでも意図した描画はできました(図3)。
r21 = Chem.MolFromSmiles('OCCCC(=O)O')
r22 = Chem.MolFromSmiles(' ')
p2 = Chem.MolFromSmiles('C1(=O)CCCO1')
eq2b = [r21, r22, p2]
img2 = Draw.MolsToGridImage(eq2b,molsPerRow=3, subImgSize=(300,300))
print('macOS11, python3.8, rdkit 2022.03.2, \' \'')
img2
の場合、error messageは出るが、
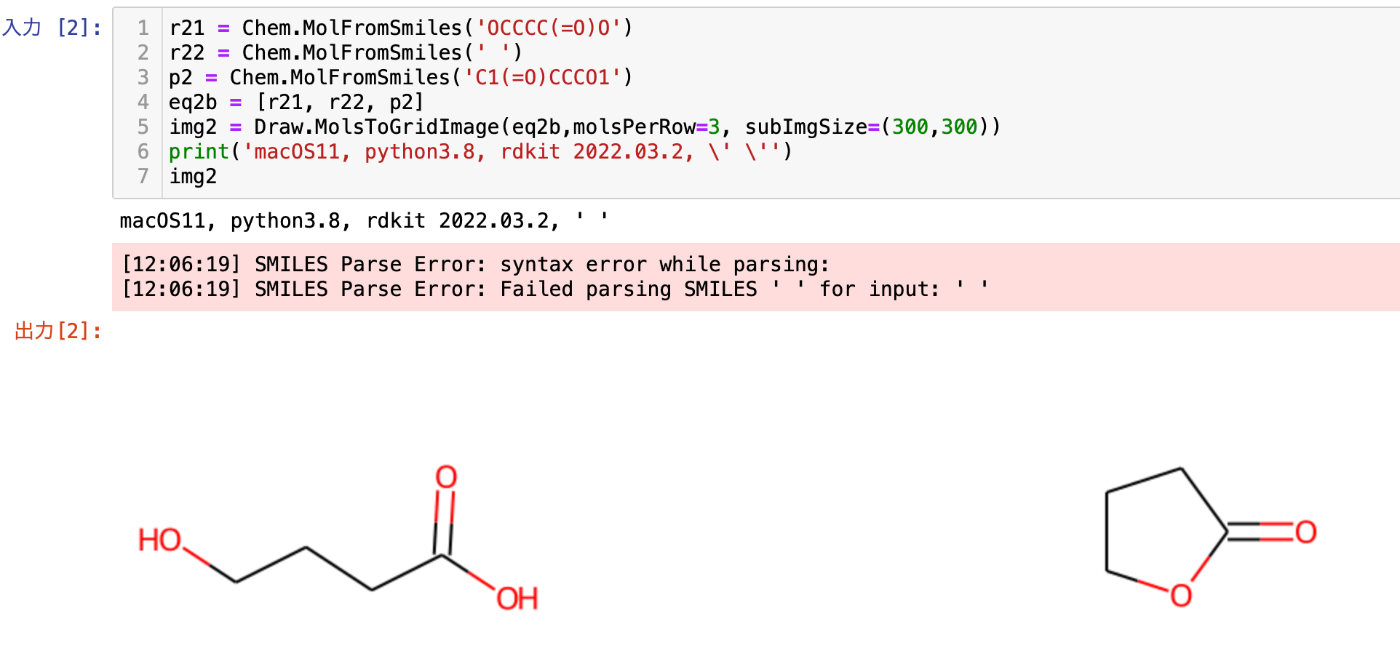
r31 = Chem.MolFromSmiles('OCCCC(=O)O')
r32 = Chem.MolFromSmiles('')
p3 = Chem.MolFromSmiles('C1(=O)CCCO1')
eq3b = [r31, r32, p3]
img3 = Draw.MolsToGridImage(eq3b,molsPerRow=3, subImgSize=(300,300))
print('macOS11, python3.8, rdkit 2022.03.2, \' \'')
img3
の場合
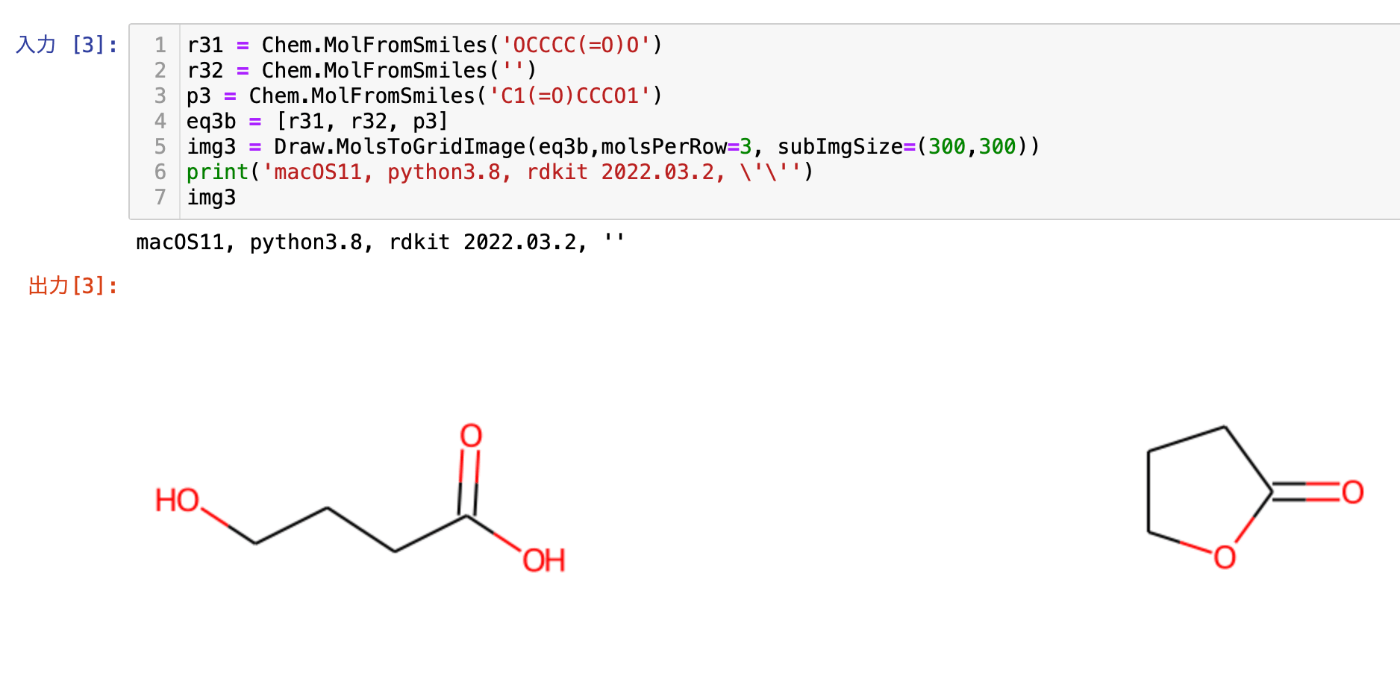
図3. 二番目の検証環境での出力
つまり、項目3の環境では、どちらの書き方でも望む描画はできるものの、error messageのことを考えると、項目2での環境とは反対に、空のmolオブジェクトを与えるSMILESの与え方として、 ' ' ではなく、 '' を使うべきのようです。
4. 種々の環境での検証
多分RDKitのバージョンの違いが原因ではないかとあたりをつけ、準備できる環境で検証しました。結果を表1にまとめています。entry 1と2が項目2に、entry 3と4が項目3の結果に相当します。OSやpythonのバージョンがばらばらで、統一された条件ではありませんが、何となく傾向がつかめました。
どうも、RDKitの2020.09のバージョンは、他のバージョンと挙動が異なるようです。また、このバージョンで意図通りの描画ができる与え方(' 'でスペースを入れる)と、windowsではerror messageが出ませんでしたが、ubuntsuではSMILES parse errorが表示されました。
表1. 空白のmolオブジェクトを返す時のSMILESの与え方
| entry | RDKit | python | OS | strings | 結果 |
|---|---|---|---|---|---|
| 1 | 2020.09.1.0 | 3.7 | windows10 | ' ' | 意図通りの描画 |
| 2 | 2020.09.1.0 | 3.7 | windows10 | '' | 空白(全く構造が表示されない) |
| 3 | 2022.03.2 | 3.8 | macOS11.6 | ' ' | error messageが出る。構造は意図したように描画される |
| 4 | 2022.03.2 | 3.8 | macOS11.6 | '' | 意図通りの描画 |
| 5 | 2022.03.2 | 3.10 | ubuntsu20.04LTS | ' ' | error messageが出る。構造は意図したように描画される |
| 6 | 2022.03.2 | 3.10 | ubuntsu20.04LTS | '' | 意図通りの描画 |
| 7 | 2020.09.3 | 3.9 | ubuntsu20.04LTS | ' ' | error messageが出る。構造は意図したように描画される |
| 8 | 2020.09.3 | 3.9 | ubuntsu20.04LTS | '' | 空白(全く構造が表示されない) |
| 9 | 2020.09.1 | 3.7 | ubuntsu20.04LTS | ' ' | error messageが出る。構造は意図したように描画される |
| 10 | 2020.09.1 | 3.7 | ubuntsu20.04LTS | '' | 空白(全く構造が表示されない) |
| 11 | 2019.09.3 | 3.8 | ubuntsu20.04LTS | ' ' | error messageが出る。構造は意図したように描画される |
| 12 | 2019.09.3 | 3.8 | ubuntsu20.04LTS | '' | 意図通りの描画 |
5. 別の解法
if文を使えば、SMILESが存在しない場合には、Noneを使ってnullオブジェクトを返すこともできます。実際、今回のケースでは、原料、生成物のSMILESをリストで与える、下記スクリプトで目的を達成できました。
ただ、同様の記述をした別のプログラムで、高い頻度でKernel Restartingとなってプログラムが停止する経験があり(何か別の要因があるのかもしれませんが)、動作に不安があるため、他のやり方を探していて、今回の違いに気づきました。
from rdkit import Chem
from rdkit.Chem import Draw
eq1 = ['CC(=O)O', 'CCO', 'CC(=O)OCC']
eq2 = ['OCCCC(=O)O', '', 'C1(=O)CCCO1']
def react(L):
R = []
for s in L:
if s != '':
m = Chem.MolFromSmiles(s)
else:
m = None
R.append(m)
return R
eq = react(eq1)
print('windows10, python3.7, rdkit 2020.09.1.0, \' \'')
Draw.MolsToGridImage(eq,molsPerRow=3, subImgSize=(300,300))
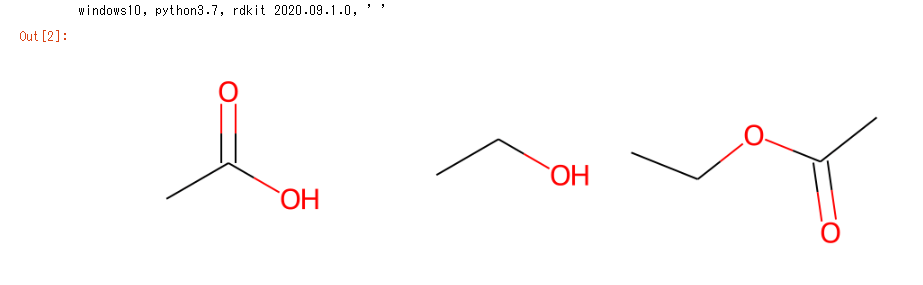
eq = react(eq2)
print('windows10, python3.7, rdkit 2020.09.1.0, \' \'')
Draw.MolsToGridImage(eq,molsPerRow=3, subImgSize=(300,300))

まとめ
RDKitのバージョンにより、微妙に動作が異なる場合もあるようで、プログラムを共有する場合には注意を要すると感じました。
Discussion