mlpack in R
mlpackってなに?
mlpackは、C++で実装された機械学習ライブラリです。header-onlyのライブラリで、Armadillo, ensmallen, cerealに依存しています(以前はBoostも使われていましたが、v4.0.0から依存が外れました)。ライセンスは3-clause BSD licenseです。
リポジトリのREADMEには次のように書かれています。
mlpack is an intuitive, fast, and flexible header-only C++ machine learning library with bindings to other languages. It is meant to be a machine learning analog to LAPACK, and aims to implement a wide array of machine learning methods and functions as a "swiss army knife" for machine learning researchers
いわゆる機械学習フレームワークみたいなものではなくて、いろいろな機械学習アルゴリズムのC++実装の詰め合わせみたいなライブラリです。
Python, Julia, GoとRのバインディングがあるほか、CLIも用意されています。この記事では、Rパッケージとしてのmlpackパッケージについて紹介します。
mlpack in Rを使ってみる
mlpackは2023年12月現在でCRANからインストールして利用できます。個人的にすごくオススメというよりは、なんかこういうのもあるよ、みたいな感じのパッケージです。
C++ライブラリとしては10年くらい前から開発されていますが、公式から提供されるRパッケージとして利用できるようになったのは比較的最近です。
Rcppの開発者であるDirk Eddelbuettelのブログ記事によると、以前はv1系のmlpackをラップしたRcppMLPACKというパッケージがあったようですが、2020年ごろには現在のmlpackパッケージの開発がはじまっており、前者のメンテナンスはすでに停まっています。
ランダムフォレスト
mlpack in R quickstart guideを参考に、とりあえずランダムフォレストを使ってみます。
Simple mlpack quickstart exampleの例そのままに、何らかのデータセットをdata.table::freadで読み込みます。このデータは10万行くらいあるのですが、とりえあず動かしたいだけにしては無駄に大きいので、ここではdplyr::slice_sampleで適当に行を減らしておきます。
require(dplyr)
df <- data.table::fread("https://www.mlpack.org/datasets/covertype-small.csv.gz")
df <- dplyr::slice_sample(df, prop = .1)
dplyr::glimpse(df)
#> Rows: 10,000
#> Columns: 55
#> $ elevation <int> 2873, 3146, 2801, 2977, 2867, 2954,…
#> $ aspect <int> 36, 326, 172, 313, 344, 76, 40, 90,…
#> $ slope <int> 19, 23, 14, 9, 7, 17, 21, 8, 13, 11…
#> $ horizontal_distance_to_hydrology <int> 216, 170, 182, 779, 124, 67, 67, 39…
#> $ vertical_distance_to_hydrology <int> 43, 37, 46, -57, 15, 16, -2, 66, 13…
#> $ horizontal_distance_to_roadways <int> 323, 3986, 1203, 1503, 3398, 1998, …
#> $ hillshade_9am <int> 215, 159, 229, 195, 206, 239, 216, …
#> $ hillshade_noon <int> 195, 208, 246, 233, 229, 206, 189, …
#> $ hillshade_3pm <int> 108, 186, 145, 178, 162, 92, 99, 12…
#> $ horizontal_distance_to_fire_points <int> 1550, 313, 1782, 2016, 3247, 2666, …
#> $ wilderness_area_1 <int> 1, 0, 0, 0, 1, 0, 0, 1, 1, 0, 1, 1,…
#> $ wilderness_area_2 <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,…
#> $ wilderness_area_3 <int> 0, 1, 1, 1, 0, 1, 1, 0, 0, 1, 0, 0,…
#> $ wilderness_area_4 <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,…
#> $ soil_type_1 <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,…
#> $ soil_type_2 <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,…
#> $ soil_type_3 <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,…
#> $ soil_type_4 <int> 0, 0, 1, 0, 0, 0, 0, 0, 0, 0, 0, 0,…
#> $ soil_type_5 <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,…
#> $ soil_type_6 <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,…
#> $ soil_type_7 <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,…
#> $ soil_type_8 <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,…
#> $ soil_type_9 <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,…
#> $ soil_type_10 <int> 0, 0, 0, 0, 0, 0, 1, 0, 0, 0, 0, 0,…
#> $ soil_type_11 <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,…
#> $ soil_type_12 <int> 0, 0, 0, 0, 1, 0, 0, 0, 1, 0, 0, 0,…
#> $ soil_type_13 <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,…
#> $ soil_type_14 <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,…
#> $ soil_type_15 <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,…
#> $ soil_type_16 <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,…
#> $ soil_type_17 <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,…
#> $ soil_type_18 <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,…
#> $ soil_type_19 <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,…
#> $ soil_type_20 <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,…
#> $ soil_type_21 <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,…
#> $ soil_type_22 <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,…
#> $ soil_type_23 <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 1, 0,…
#> $ soil_type_24 <int> 0, 1, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,…
#> $ soil_type_25 <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,…
#> $ soil_type_26 <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,…
#> $ soil_type_27 <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,…
#> $ soil_type_28 <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,…
#> $ soil_type_29 <int> 1, 0, 0, 0, 0, 0, 0, 1, 0, 0, 0, 0,…
#> $ soil_type_30 <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,…
#> $ soil_type_31 <int> 0, 0, 0, 1, 0, 0, 0, 0, 0, 0, 0, 0,…
#> $ soil_type_32 <int> 0, 0, 0, 0, 0, 1, 0, 0, 0, 1, 0, 0,…
#> $ soil_type_33 <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,…
#> $ soil_type_34 <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,…
#> $ soil_type_35 <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,…
#> $ soil_type_36 <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,…
#> $ soil_type_37 <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,…
#> $ soil_type_38 <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 1,…
#> $ soil_type_39 <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,…
#> $ soil_type_40 <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,…
#> $ label <int> 5, 1, 2, 2, 2, 2, 2, 1, 2, 2, 2, 1,…
Rで教師あり学習をやる場合、既存のRパッケージでは、訓練用データで学習したモデルを変数に格納してpredictという総称関数にnew_dataと一緒につっこむことで、その他のデータに対する予測をするという使い方をすることが多いと思います。一方で、mlpackの関数はそのような建付けにはなっていません。
たとえば、mlpack::random_forestで学習したモデルを使って予測するには、次のような雰囲気の使い方をします。
prepdata <-
mlpack::preprocess_split(
input = dplyr::select(df, !"label"),
input_labels = dplyr::select(df, "label"),
stratify_data = TRUE,
test_ratio = 0.3,
verbose = FALSE
)
str(prepdata)
#> List of 4
#> $ test : num [1:2997, 1:54] 2729 2912 2734 3165 3264 ...
#> $ test_labels : num [1:2997, 1] 2 2 2 2 1 2 1 1 2 2 ...
#> $ training : num [1:7003, 1:54] 2035 2754 2558 2207 2227 ...
#> $ training_labels: num [1:7003, 1] 3 3 3 4 3 3 3 3 3 3 ...
rf_model <- mlpack::random_forest(
training = prepdata$training,
labels = prepdata$training_labels,
minimum_leaf_size = 3,
num_trees = 300
)
rf_pred <- mlpack::random_forest(
input_model = rf_model$output_model,
test = prepdata$test,
test_labels = prepdata$test_labels
)
table(prepdata$test_labels, rf_pred$predictions)
#>
#> 1 2 3 5 6 7
#> 1 799 268 1 0 0 4
#> 2 226 1240 9 2 5 1
#> 3 0 16 176 0 1 0
#> 4 0 0 10 0 0 0
#> 5 0 47 1 1 0 0
#> 6 0 29 45 0 16 0
#> 7 56 0 0 0 0 44
なんか変な感じですね。慣れましょう。
LMNN
ドキュメントによると、mlpackには次のような関数があります。
mlpack 4.3.0 の関数
- classification:
- decision_tree()
- linear_svm()
- logistic_regression()
- nbc()
- perceptron()
- random_forest()
- softmax_regression()
- adaboost()
- regression:
- bayesian_linear_regression()
- lars()
- linear_regression()
- clustering:
- dbscan()
- gmm_train()
- gmm_generate()
- gmm_probability()
- hoeffding_tree()
- kmeans()
- mean_shift()
- geometry:
- approx_kfn()
- emst()
- fastmks()
- lsh()
- knn()
- kfn()
- krann()
- preprocessing:
- preprocess_split()
- preprocess_binarize()
- preprocess_describe()
- preprocess_scale()
- preprocess_one_hot_encoding()
- image_converter()
- misc. / other:
- cf()
- det()
- hmm_train()
- hmm_generate()
- hmm_loglik()
- hmm_viterbi()
- kde()
- nmf()
- transformations:
- kernel_pca()
- lmnn()
- local_coordinate_coding()
- nca()
- pca()
- radical()
- sparse_coding()
めずらしいところでは、Large Margin Nearest Neighbors (LMNN)というのがあります。deepではないmetric learning、距離学習というやつですね。
Pythonだとmetric-learnというライブラリがありますが、Rで距離学習ができるパッケージは他に思いつかないです。
modeldata::penguinsに対して使ってみます。まず、ふつうにPCAで2次元にして可視化してみます。
penguins <- tidyr::drop_na(modeldata::penguins)
require(ggplot2)
penguins |>
dplyr::select(!species) |>
mlpack::pca(new_dimensionality = 2, scale = TRUE) |>
purrr::pluck("output") |>
as.data.frame() |>
ggplot(aes(x = V1, y = V2)) +
geom_point(aes(col = penguins$species))
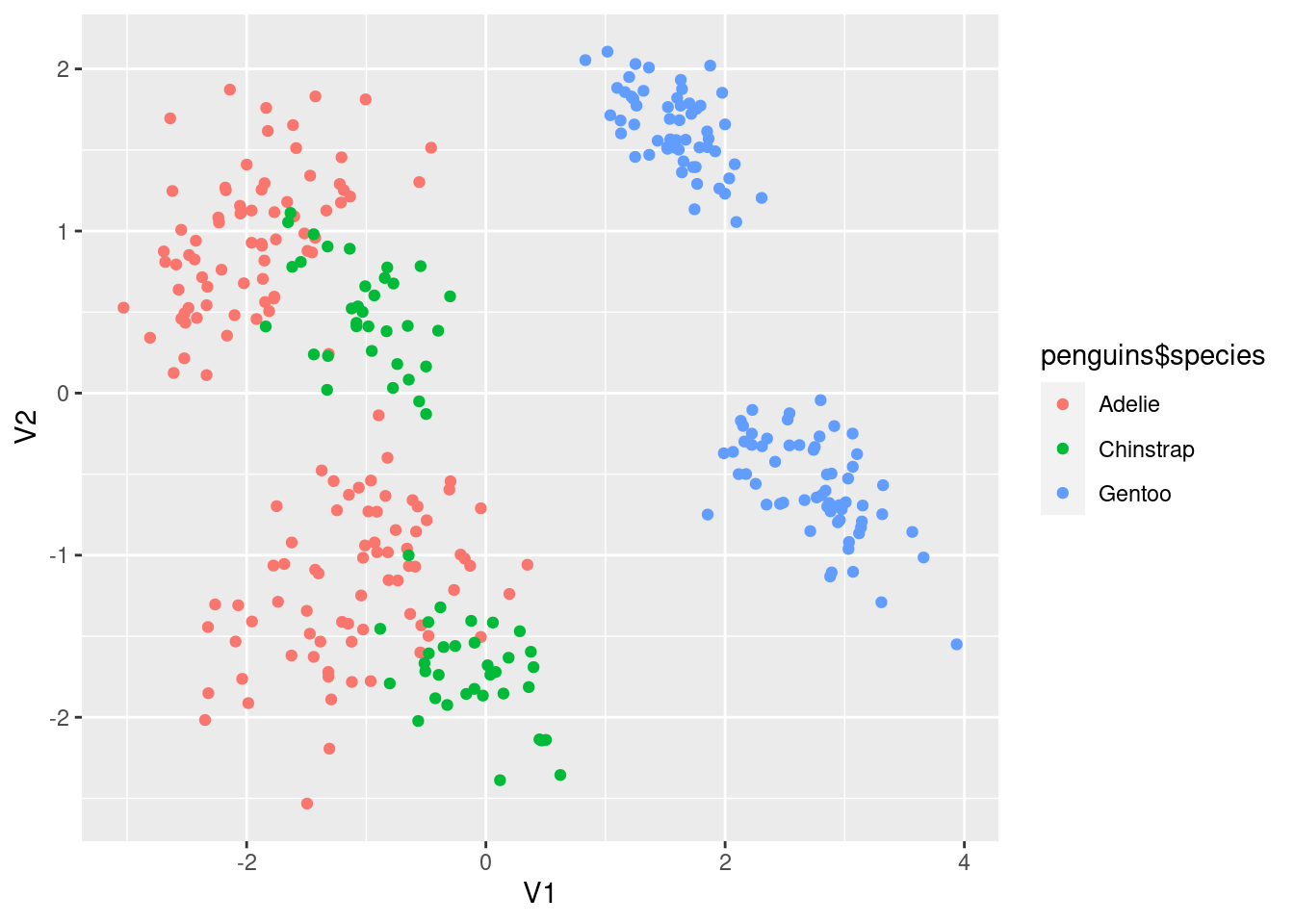
オス・メスで体格が違うので、いずれの種類でもまとまりが2つに分かれています。次にmlpack::lmnnを使って、変換されたデータについて同様に可視化してみます。
ret <- mlpack::lmnn(
input = dplyr::select(penguins, !species),
labels = as.matrix(as.integer(penguins$species)),
linear_scan = TRUE,
normalize = TRUE,
seed = 1218
)
ret$transformed_data |>
mlpack::pca(new_dimensionality = 2, scale = TRUE) |>
purrr::pluck("output") |>
as.data.frame() |>
ggplot(aes(x = V1, y = V2)) +
geom_point(aes(col = penguins$species))
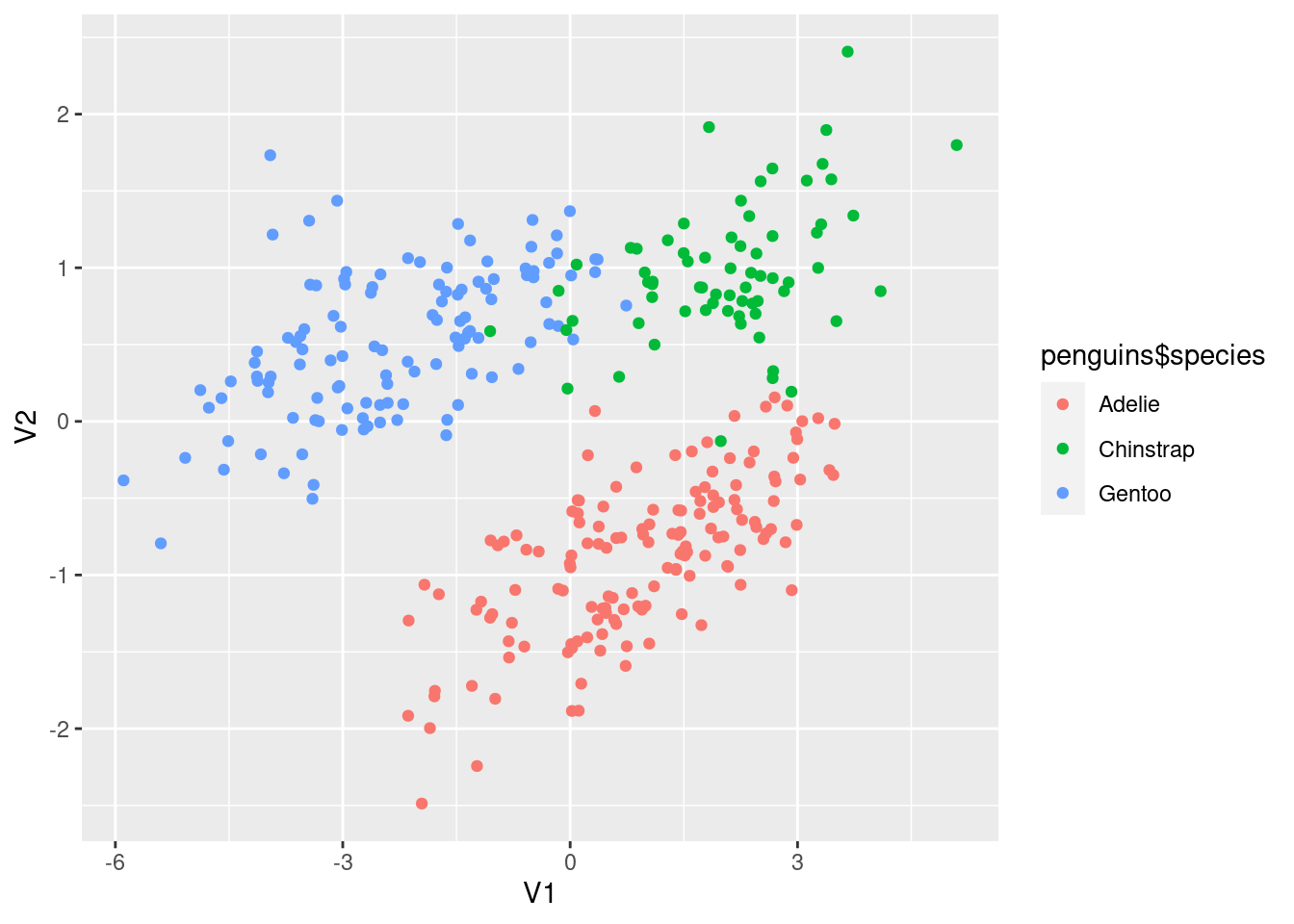
それぞれの種類がよりはっきりと一つのまとまりになっています。
ところで、LMNNよりむしろmlpack::pcaが扱いやすくてよいですね。他のPCAする関数とは違って、データにfactor型の列があってもそのままで大丈夫なようです。便利。
他のRパッケージとの比較
いくつかのmlpackの関数と他のRパッケージの関数を雑に比較してみます。
ランダムフォレスト
ランダムフォレストの場合、rangerのほうが速いようです。また、importanceを見たりはmlpackではできません。
microbenchmark::microbenchmark(
ranger = {
ranger::ranger(
x = as.data.frame(prepdata$training),
y = as.factor(prepdata$training_labels),
num.trees = 300,
mtry = floor(sqrt(ncol(prepdata$training))),
min.node.size = 3,
max.depth = 0,
num.threads = parallelly::availableCores()
)
},
mlpack = {
mlpack::random_forest(
training = prepdata$training,
labels = prepdata$training_labels,
num_trees = 300,
subspace_dim = floor(sqrt(ncol(prepdata$training))),
minimum_leaf_size = 3,
maximum_depth = 0
)
},
times = 5,
check = NULL
)
#> Unit: milliseconds
#> expr min lq mean median uq max neval
#> ranger 498.4793 503.0729 527.3027 505.2638 553.5399 576.1574 5
#> mlpack 1749.6105 1789.9572 1803.6196 1819.6237 1829.0773 1829.8295 5
k Nearest Neighbor
k-近傍法による分類ではなく、最近傍探索です。次の関数を試してみます。
mlpack::knndbscan::kNNFNN::get.knnx
# 日本にある鉄道駅の代表点の座標: https://paithiov909.r-universe.dev/jprailway/doc/manual.html#stations
x <- jprailway::stations |>
dplyr::select(lat, lng) |>
sf::st_drop_geometry()
dplyr::glimpse(x)
#> Rows: 11,427
#> Columns: 2
#> $ lat <dbl> 41.77371, 41.77264, 41.80356, 41.84646, 41.86464, 41.88697, 41.905…
#> $ lng <dbl> 140.7264, 140.7280, 140.7335, 140.7230, 140.7136, 140.6886, 140.64…
microbenchmark::microbenchmark(
mlpack = {
nn <- mlpack::knn(reference = x, k = 3, query = x[1:10, ], leaf_size = 20)
(nn$neighbors[, 2] + 1)
},
dbscan = {
nn <- dbscan::kNN(x, k = 3, query = x[1:10, ], bucketSize = 20)
(nn$id[, 2])
},
FNN = {
nn <- FNN::get.knnx(x, k = 3, query = x[1:10, ])
(nn$nn.index[, 2])
},
times = 5,
check = "equivalent"
)
#> Unit: milliseconds
#> expr min lq mean median uq max neval
#> mlpack 2.342788 2.483178 2.620054 2.600677 2.826408 2.847217 5
#> dbscan 1.318337 1.323457 1.638475 1.377101 1.793097 2.380383 5
#> FNN 3.429315 3.436054 3.610419 3.692489 3.737369 3.756870 5
とくに何も指定しない場合、いずれもkd木が使われます。与えるデータにもよるようですが、kd木ではdbscanが速そうです。
一方で、dbscanはkd木以外の木構造は選べません。このなかではmlpack::knnの実装が使える木構造の数がもっとも多く、ドキュメントによると、次の木構造が使えるようです。
kd-trees, vp trees, random projection trees, UB trees, R trees, R* trees, X trees, Hilbert R trees, R+ trees, R++ trees, spill trees, and octrees
不勉強なので、どういったデータに対してどんな木構造を使いつつ探索するとうれしいのかわからないですが、わかる人はいろいろできて便利なのかもしれません。
気になった点として、mlpack::knnで返される近傍点はなぜかゼロではじまるインデックスになっているようで、正しいインデックスを得るには1を足す必要があります。
非負値行列因子分解(NMF)
RでNMFできるパッケージとしては、NMFパッケージがあります。ここではCRANにあるバージョンではなく、GitHubにある開発版をインストールして使っています。
x <- modeldata::penguins |>
tidyr::drop_na() |>
dplyr::select(where(is.numeric))
microbenchmark::microbenchmark(
mlpack = {
nm <- mlpack::nmf(x, rank = 3)
(nm$w %*% nm$h)
},
NMF = {
nm <- NMF::nmf(x, rank = 3)
NMF::basis(nm) %*% NMF::coef(nm)
},
times = 20,
unit = "ms"
)
#> Unit: milliseconds
#> expr min lq mean median uq max
#> mlpack 0.501336 0.6238555 0.7711193 0.8153355 0.8874605 0.984438
#> NMF 237.599260 238.1018755 249.2619699 242.2818745 260.3845735 281.559038
#> neval
#> 20
#> 20
NMFは実装されているアルゴリズムが新しめなものばかりで、しかも多くのアルゴリズムが使えます。一方で、デフォルトで使うと妙に遅い気がします。このあたりはmlpackで代用してもよいのかなと思いました。
まとめ
mlpackは一つのパッケージ内でさまざまなアルゴリズムが実装されていて便利ですが、他の実装と比べて必ずしも速いわけではないです。また、Rパッケージとしてはまだちょっとこなれていないというか、扱いづらい部分もあります。
さまざまなアルゴリズムを利用するのにmlpackに一本化して乗り換えるというよりは、やりたいことによってはmlpackを選択肢の一つとして検討するくらいがよさそうです。

Discussion