Stanford Ribonanza RNA Foldingコンペ 上位解法まとめ
コンペURL
2020年にも同様のコンペがありましたが、今回はデータが100倍くらいになって来ています。
どんなコンペ?
RNA配列の各塩基の薬品との反応性を予測するコンペです。

RNAは上のように様々な形を取るのですが、他の塩基とくっついている部分は薬品と反応しづらく分解しづらいという特徴があります。反応しやすい文字の特徴が分かればmRNAワクチンの設計などにも役にたつと考えられます。
また、このコンペの問題を自然言語処理系の話にたとえていうと、各wordにスコアがついているので、単wordごとのスコアを予測する、という問題と解釈することもできます。
評価指標はMean Absolute Error(MAE)でした。
ベースライン解法
生のTransformerを使って各塩基ごとの回帰問題として解くという手法がベースライン解法として高評価を受けていました。
Pytorchだと以下のNotebookが高評価
tensorflowだと以下のNotebookが高評価でした。
上位Solutions
Leaderboard
1〜5位のソリューションを確認しています。
個人的には2ndのDiscussionがシンプルで一番理解しやすかった。
概要
-
基本的にはモデリングで工夫するコンペだった。上位の工夫は以下の2点。
- 各RNA配列のメタデータとして与えられるBPP Matrix(*後述)をAttentionに追加
- 学習用のデータと評価用のデータのRNA配列長が異なることから、Positional Encoding(位置エンコーディング)を絶対位置でないものに変更
-
データ数が増えていたため、前回のコンペで使えた手法(Pseudo labelingやデータの水増しなど)はあまりWorkしなかった
モデリング
1. ベースモデル
- Squeezeformer(2nd, 3rd)
... Transformerだと通常はマルチヘッドアテンションのあとに重ねられるフォワード層の前に、畳み込み層+フォワード層を入れたもの。
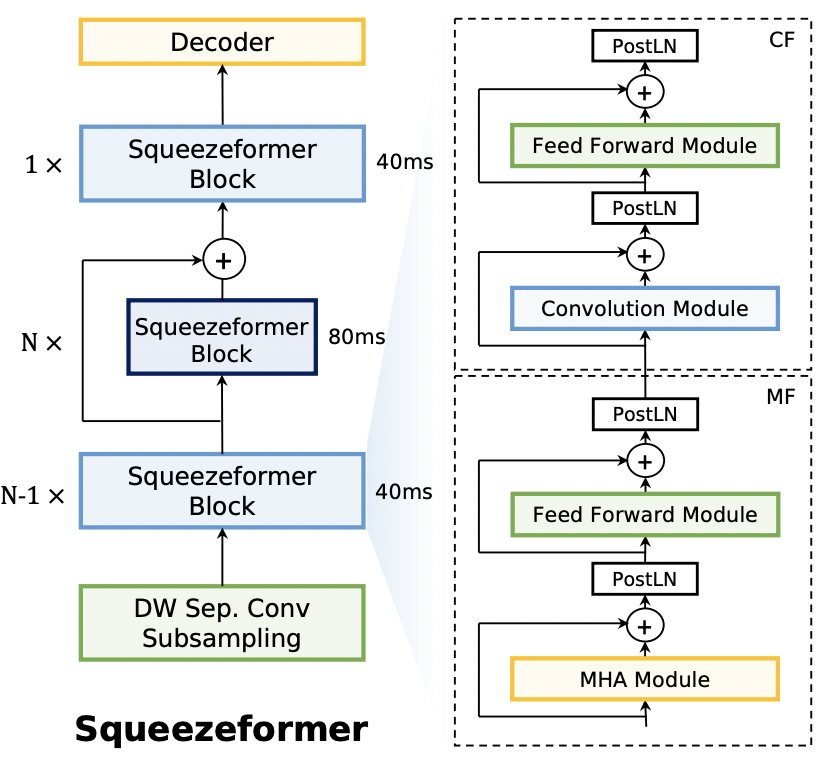
画像は公式レポジトリより
音声分野で使われているConformerをよりシンプルにしたもので、Googleの手話コンペでも使われており、シーケンス長が違うときに有効な手法らしい。
- RNAdegformer(4th)
前回のRNAコンペを通じて作成されたもので、以下に記載のBPP Matrixを位置エンコーディングのようにマルチヘッドアテンションに足し合わせる処理を行うモデル。
このモデルはめちゃくちゃ優秀で、これ使うだけで銀圏上位に入るみたいです。
2. 各塩基間の関連性行列(BPP matrix)を上手くあつかう
BPP matrixはホストから提供されたもので、各塩基間の関係性が示されたもの。ただしこの値もEternaFoldというソフトウェアによって予測されたもの。
この2次元行列の取り扱いはベースラインモデルでは使われていなかったものの、1st〜5thは以下のPositional Encodingと同様に畳み込み層でまとめ、マルチヘッドアテンションに足し合わせることで、BPPを使用していました。
1stの方の絵がわかりやすいです。

3. Positional Encoding(位置エンコーディング)
一般的なTransformersの処理において、単語の位置情報も情報として付与されています。一般的には三角関数を使った位置埋め込みがよく使われます。
しかし、学習用のデータと評価用のデータとで、文章の長さが異なることから、位置エンコーディングを工夫している人が多くいました。1〜5位まで見てみたところ、Dynamic position bias(動的位置バイアス)、Relative positional bias(相対位置バイアス)、AliBi positional encoding(AliBi 位置エンコーディング) が使われていました。
- Dynamic Positional Bias/Relative positional bias(1st, 5th)
Relative positional biasは絶対的な位置ではなく、相対的な位置を埋め込む仕組みであり、Dynamic Positional Biasはさらに相対位置埋め込みを学習可能にしたもの。
1stはAliBiも(学習を行わない)Relatve positional encodingも試したが、これが一番よいと結論づけたらしい。
- AliBi Positional encoding(2nd, 4th)
... Attention scoreの計算時において、KeyとQueryがどれだけ離れているかに応じてQueryがKeyに割り当てられるAttention scoreにペナルティを与えるもの。つまり、近い単語をより重要視するようになるということ。元論文図3がわかりやすい。
遠くにある塩基とも反応しそうだが、学習より推論の方がシーケンスが長い場合を想定されているため絶対的な位置のものよりはよかったということであろうか。
4. ヘッド
上位の多くがGRUをヘッドとして利用していました(2nd, 4th, 5th)。ちょっと精度が上がったらしい。
その他工夫
-
Pseudo labeling
-
上位勢は実験はしている。あまり精度は向上しなかったようだ。
-
Root Mean Square Layer Normalization
- https://arxiv.org/abs/1910.07467
- 4thや5thが使用していた。ちょっと早いLayer Normalizationらしい。
感想
-
Transformers系のモデルは前回のコンペは賞金圏内にはいなかった印象なのだが、今回(恐らくデータ量が増えたことにより)ゴリゴリ活躍していて熱い展開でしたね...!!
-
自分は3週間参加で25%くらいでした。。普通に過学習していた。。
Discussion