tidymodelsをもちいた生存時間モデル_R
概要
どうも始めまして製薬企業で疫学研究に関わっております新人エキガクニストです。
本記事ではモデリングの初めから終わりの一通りを解説することを目指しております。
- coxモデル、ランダムフォレスト、xgboostでのモデリング・評価
- パラメーターチューニング(1においても最も優れていたモデルでの)
- 特徴量評価(追記予定)
ご意見コメント大歓迎です!!
参考文献
- Rユーザーのためのtidymodels実践入門
https://amzn.asia/d/ayMq8bs - How long until building complaints are dispositioned? A survival
analysis case study
https://www.tidymodels.org/learn/statistics/survival-case-study/
内容
それでは実際のモデリングにGO!
パッケージ
library(pacman)
p_load("tidymodels")
p_load("censored") #tidymodelsの生存時間解析をサポート
p_load("glmnet") #cox比例ハザードモデル構築のため
p_load("aorsf") #ランダムフォレスト構築のため
p_load("mboost") #boostiモデルう構築のため
p_load("tidyverse")
p_load("survival")
使用するデータに関して
survivalパッケージのlungデータ(進行性肺がん患者のデータ)をもちいる。
今回は簡単にするため以下の列だけ
- time: 生存期間(日数)
- status: 打ち切り情報 1=打ち切り、2=死亡
- age: 年齢(年)
- sex: 性別 男性=1 女性=2
- ph.ecog: ECOGパフォーマンススコア 医師による評価。0=無症状、1=症状ありだが完全に活動可能、2=日中の50%未満をベッドで過ごす、3=日中の50%以上をベッドで過ごすが寝たきりではない、4=寝たきり
- wt.loss: 過去6ヶ月の体重減少(ポンド)
data(cancer,package="survival")
lung <- lung %>%
select(time,status,age,sex,ph.ecog,wt.loss) %>%
mutate(sex=as.factor(sex)) #カテゴリカルデータ化
glimpse(lung)

survオブジェクト列の作成
予測の目的変数としてはSurvオブジェクトが利用されるため、Survオブジェクトを作成する。
lung <- lung %>%
mutate(Outcome = Surv(time, status ==2),.keep="unused") # unused 新しいcolの計算にもちいられなかった列が残る
データ分割、データチェック
データを分割し、トレーニングデータをKaplan_Meier曲線をもちいて確認する
今回は簡単のため、test,train,validationの3分割とする。交差検証等をもちいたい場合は分割方法を変更する(要望をおおければ追記致します)
set.seed(123)
#データをテスト、トレイン、検証の3分割を一度に実施可能
lung_split <- initial_validation_split(lung)
lung_train <- training(lung_split)
lung_set <- validation_set(lung_split)
survfit(Outcome ~ 1 ,data= lung_train) %>% plot()

coxモデル、ランダムフォレスト、xgboostでのモデリング・評価
tidymodelsではモデルの条件と前処理の条件をWorkflowにセットしてモデル評価を実施する(以下の順序)。これにより流用しやすいかつ理解しやすいプログラムが作成できるらしい。。。
- 処理,モデル条件の設定
- workfowにセット
- 評価方法の指定(リンサンプル方法、評価指標)
モデル条件,前処理の設定
以下のコマンドで使用するモデルとデータに対して実施する前処理を定義します。
使用可能なモデルはcensoredのページを確認してみてください。
#モデルの前処理を規定、recipe内の引数として記載するformulaがモデルのformulaとしてもちいられる
rec <- recipe(Outcome ~ .,data = lung_train) %>%
step_dummy(sex) %>% #ダミーデーター化
step_naomit(all_predictors())
#モデルの条件(アルゴリズム、パラメーター等)
## cox比例ハザード
cox_spec <- proportional_hazards(penalty=0) %>%
set_engine("glmnet") %>% #裏で実際に動くパッケージ
set_mode("censored regression") #生存時間解析であることを記載
## ランダムフォレスト
rand_spec <- rand_forest() %>%
set_engine("aorsf") %>%
set_mode("censored regression")
## xgboost
boost_spec <- boost_tree() %>%
set_engine("mboost") %>%
set_mode("censored regression")
workflowにセット
models <- workflow_set(
preproc = list(rec),
models = list(cox = cox_spec,
rand = rand_spec,
boost = boost_spec),
cross = T
)
評価方法の指定
今回は評価方法としてtime-dependent AUCとC-index(Concordance_survival)をもちいる。
tme-dependent AUCは評価を実施する時点によって値が異なるため、時点も設定する必要がある
survival_metrics <- metric_set(roc_auc_survival, concordance_survival)
evaluation_time_points <- seq(0,800,50)
モデルの実装
モデルを実装と評価を行う。
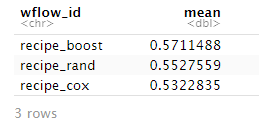
Cindexの評価においてはboost treeが最も精度が高いことがわかった。
models <- models %>% workflow_map(
fn = "fit_resamples",
resamples = lung_set,
metrics = survival_metrics,
eval_time = evaluation_time_points
)
models %>% rank_results(rank_metric = "concordance_survival",
select_best = T) %>%
filter(.metric=="concordance_survival") %>%
select(wflow_id,mean)
パラメーターチューニング、最終モデルの構築
上記からboost treeが最も精度が高い事がわかった。
そこでboost treeモデルのパラメーターチューンを行う
いくつかのパラメーターのチューニングが可能だか今回はtreesとtree_depthに対して実施する。
明示的にパラメーター
boost_spec_tune <- boost_tree(trees=tune(),tree_depth = tune()) %>% #チューニングしたいパラメーター=tune()とする
set_engine("mboost") %>%
set_mode("censored regression")
boost_wf <- workflow() %>% #workflowに設定
add_recipe(rec) %>%
add_model(boost_spec_tune)
model_tuned <- tune_grid( #パラメーターチューニングの実施
boost_wf,
resamples = lung_set,
grid = 10,
metrics = survival_metrics,
eval_time = evaluation_time_points,
)
最も精度のよいパラメーターを確認する。
show_best(model_tuned, metric = "concordance_survival", n = 5)
最終モデルの構築
パラメーターチューニングが完了したら、最もよいパラメーターをもちいてモデルの再学習をおこなう。
これまでtrainデータで学習し、validationデータで評価してきたが、最もよいパラメーターをもちいてtrainデータとvalidationデータを合わせたデータで学習をおこないtestデータで評価する。
param_best <- select_best(model_tuned, metric = "concordance_survival")
last_model_wflow <- finalize_workflow(boost_wf, param_best)
last_model_fit <- last_fit(
last_model_wflow,
split = lung_split,
metrics = survival_metrics,
eval_time = evaluation_time_points
)
精度指標の取り出し
collect_metrics(last_model_fit) %>%
filter(.metric=="concordance_survival")

学習が進んだらまた追記しますね~
Discussion