YAMLでデータ処理パイプラインを定義できる「DocETL」を試す
GitHubレポジトリ
ドキュメント
DocETL: 複雑な文書処理のためのシステム
referred from https://ucbepic.github.io/docetl/DocETLは、LLMを搭載したデータ処理パイプラインの作成と実行を行うためのツールである。複雑なデータに対する複雑なデータ操作を定義するための、ローコードで宣言型のYAMLインターフェースを提供する。
DocETLの使用タイミング
DocETLは、文書や構造化されていないデータセットの集合に対して複雑なタスクを実行し、正確性と出力品質を最大限に高めたい場合に最適な選択肢である。DocETLの使用を検討すべき状況は、以下の通りである。
- 複雑なタスクをMapReduceで表現したい場合(例えば、ドキュメントのマップを行い、マップコールの結果でグループ化およびリダクションを行う場合など)
- LLMの精度を最大限に高めるためのパイプラインや一連の操作の最適な記述方法が不明な場合
- LLMの推論を効果的に行うには長すぎる、または1つのプロンプトに収まらない長いドキュメントを処理する場合
- 検証基準があり、検証に失敗した際にタスクを自動的に再試行させたい場合
🚀 機能
- 豊富なオペレータスイート: エンティティの解決のための「resolve」や、ドキュメントを分割する際のコンテキスト維持のための「gather」といった特殊なオペレータを含む、複雑なデータ処理向けにカスタマイズされている。
- ローコードインターフェース: YAMLを使用して、パイプラインとプロンプトを簡単に定義できる。プロンプトは100%制御可能。
- 柔軟な処理: 法律、医療、社会科学などの分野におけるさまざまなドキュメントタイプと処理タスクに対応。
- 精度の最適化: 当社の最適化ツールは、LLMエージェントを活用して、お客様のパイプラインの論理的に等価なさまざまな書き換えを試行し、最も精度の高いバージョンを自動的に選択する。これには、精度が頭打ちになる前に、1回のリダクション操作で処理する文書の数の上限を見つけることも含まれる。
オープンソースで、ライセンスはMITライセンス
ドキュメントのGetting startedに従って進める。まずはColaboratoryで。
インストール
pipでパッケージインストールする方法とソースからインストールする方法が用意されているが、今回はパッケージで。extraにparsingを追加すると、Office文書(Word/Excel/PowerPoint)や音声なども扱えるようになるみたい。
!pip install docetl[parsing]
!pip freeze | grep docetl
docetl==0.1.5
これでdocetlのCLIが使えるようになる
!docetl version
DocETL version: 0.1.5
Tutorial
実際の文書処理をチュートリアルに沿って進めていく。
DocETLは内部でLiteLLMを使用しており、デフォルトではOpenAIを使用している様子。OpenAIのAPIキーをセットする。
import os
from google.colab import userdata
os.environ["OPENAI_API_KEY"] = userdata.get('OPENAI_API_KEY')
シンプルなJSONのデータを用意する。例では、医師と患者の会話のようなデータになっている。もともとは英語だったが日本語に翻訳した。これをmedical_transcripts.jsonというファイルで保存。余計なケツカンマが付与されていたが削除している。
%%writefile medical_transcripts.json
[
{
"src": "医師: こんにちは、ジョンソンさん。新しい薬、リシノプリルを飲み始めてから、体調はいかがですか?\n患者: ええ、先生。血圧は改善されたように感じますが、乾いた咳が続いています..."
},
{
"src": "医師: おはようございます、スミスさん。今日はメトフォルミン処方の経過観察ですね。\n患者: はい、先生。規則正しく服用していますが、いくつかの副作用が気になっています..."
}
]
そしてYAMLでパイプラインの定義を行う。ここでは、上記のデータから、使用されている一般的な薬名を抽出し、それぞれの用途と副作用をサマリとしてまとめる、というものを定義している。これをpipeline.yamlというファイルで保存する。
%%writefile pipeline.yaml
datasets:
transcripts:
path: medical_transcripts.json
type: file
default_model: gpt-4o-mini
operations:
- name: extract_medications
type: map
#sample: 1 #与えられたデータを減らしたい場合は以下のように指定すると、指定された数でランダムでサンプリングされる
output:
schema:
medication: list[str]
prompt: |
以下の医師と患者の会話の記録を分析してください:
{{ input.src }}
会話記録に記載されているすべての薬剤を抽出し、リスト化してください。
薬剤が記載されていない場合は、空のリストを返してください。
- name: unnest_medications
type: unnest
unnest_key: medication
- name: resolve_medications
type: resolve
blocking_keys:
- medication
blocking_threshold: 0.6162
comparison_prompt: |
以下の2つの薬剤のエントリーを比較してください:
エントリー1: {{ input1.medication }}
エントリー2: {{ input2.medication }}
これらの薬剤が同じまたは密接に関連している可能性があるかどうかを判断してください。
embedding_model: text-embedding-3-small
output:
schema:
medication: str
resolution_prompt: |
以下の一致した薬剤のエントリーに基づいて:
{% for entry in inputs %}
エントリー{{ loop.index }}: {{ entry.medication }}
{% endfor %}
このグループのエントリーを最もよく表す標準化された薬剤名を決定してください。
標準化された名前は、すべての一致したエントリーを最もよく表す、広く認識された薬剤名であるべきです。
- name: summarize_prescriptions
type: reduce
reduce_key:
- medication
output:
schema:
side_effects: str
uses: str
prompt: |
以下は、医師と患者の間の会話の記録です:
{% for input in inputs %}
会話記録 {{ loop.index }}:
{{ input.src }}
{% endfor %}
薬剤{{ reduce_key }}について、上記の会話に基づいて以下の情報を提供してください:
1. 副作用: {{ reduce_key }}に関して言及されたすべての副作用を要約してください。
2. 治療用途: {{ reduce_key }}が処方または推奨された医療条件や症状を説明してください。
要約する際には以下を守ってください:
- 提供された記録の情報にのみ基づく
- {{ reduce_key }}にのみ焦点を当て、他の薬剤には触れない
- すべての記録から関連する詳細を含む
- 明確で簡潔に書く
- 記録からの引用を含める
pipeline:
steps:
- name: medical_info_extraction
input: transcripts
operations:
- extract_medications
- unnest_medications
- resolve_medications
- summarize_prescriptions
output:
type: file
path: medication_summaries.json
intermediate_dir: intermediate_results
定義されている内容は以下となっているが、ここでは細かいところは触れない。
- 薬の抽出: 各会話ログを分析し、言及されているすべての薬を特定し、リスト化する。
- ネスティングの解除: 抽出された薬のリストを平坦化し、各薬(および関連データ)を個別の文書とする。この演算子は、pandasのexplode操作に類似している。
- 薬の標準化: 類似した薬の名称を統合し、エントリを標準化する。このステップは、同一の薬の異なるバリエーションやブランド名の統合に役立つ。例えば、ステップ1では「イブプロフェン」と「モートリン 800mg」が別々の薬として抽出され、ステップ3ではそれらが「イブプロフェン」という単一のエントリに統合される可能性がある。
- 要約の生成: 各固有の薬について、関連するすべてのトランスクリプトの情報に基づいて、副作用と治療用途の要約を生成する。
なお、余談だが2024/10/2時点のチュートリアルのYAML定義にはtypoと思われる箇所がある。上記のコードではそれを修正している。
(snip)
- name: summarize_prescriptions
(snip)
prompt: |
以下は、医師と患者の間の会話の記録です:
{% for value in values %} # ここ!
Transcript {{ loop.index }}:
{{ value.src }}
{% endfor %}
(snip)
でパイプラインを実行する。gpt-4o-miniで$0.1ぐらいかかるらしい。
!docetl run pipeline.yaml
パイプラインの実行が完了した。warningsがでているのはおそらく、OpenAIのPrompt Cachingによりusageのオブジェクトの内容が変わってしまったためだと思うので、気にしなくて良いと思う。
─────────────────────────────────────────── Syntax Check ───────────────────────────────────────────
Performing syntax check on all operations...
Syntax check passed for all operations.
──────────────────────────────────────── Pipeline Execution ────────────────────────────────────────
───────────────────────────────────────── Loading Datasets ─────────────────────────────────────────
Loaded dataset: transcripts
───────────────────────────── Executing Step: medical_info_extraction ──────────────────────────────
Running Operation:
Type: map
Name: extract_medications
/usr/local/lib/python3.10/dist-packages/pydantic/main.py:390: UserWarning: Pydantic serializer
warnings:
Expected `PromptTokensDetails` but got `dict` with value `{'audio_tokens': None, 'cached_tokens':
0}` - serialized value may not be as expected
return self.__pydantic_serializer__.to_python(
Processing map items: 100%|██████████| 2/2 [00:02<00:00, 1.27s/it]
[13:14:26] Operation extract_medications completed. Cost: $0.00
YAML定義にあるように結果がmedication_summaries.jsonに出力される
import json
with open('medication_summaries.json', 'r', encoding='utf-8') as file:
print(json.dumps(json.load(file), indent=2, ensure_ascii=False))
[
{
"side_effects": "リシノプリルの副作用として、患者は「乾いた咳が続いています」と述べています。",
"uses": "リシノプリルは、患者が血圧の改善を感じていることから、高血圧の治療目的で処方されています。",
"medication": "リシノプリル"
},
{
"side_effects": "医師と患者の会話では、メトフォルミンに関して具体的な副作用が言及されていませんでしたが、患者は「いくつかの副作用が気になっています」と述べています。",
"uses": "メトフォルミンは、患者が規則正しく服用していることから、糖尿病の治療に用いられる薬剤であると考えられます。",
"medication": "メトフォルミン"
}
]
DocETLにおけるパイプラインの定義
DocETLのキモはパイプラインの定義になる。これについてはドキュメントでは「コアコンセプト」として説明されており、以下の内容に分かれている。
- オペレータと検証
- 出力スキーマ
- パイプライン
- 最適化
順序的には「オペレータと検証」からなのだけど、全体の定義からおさえたほうがわかりやすそうな気がしたので、まず「パイプライン」を見てみる。
パイプライン
パイプラインは、データ処理の流れを定義したもの。DocETLで定義するパイプラインは以下の4つで構成される
- デフォルトモデル: パイプラインで使用するデフォルトの言語モデル
- データセット: パイプラインで処理する入力データ
- オペレータ: データの変換等を定義した個々の処理ステップ
- パイプライン定義: 処理ステップの流れや出力に関する設定
pipeline.yamlでは上記をこんな感じで設定する。
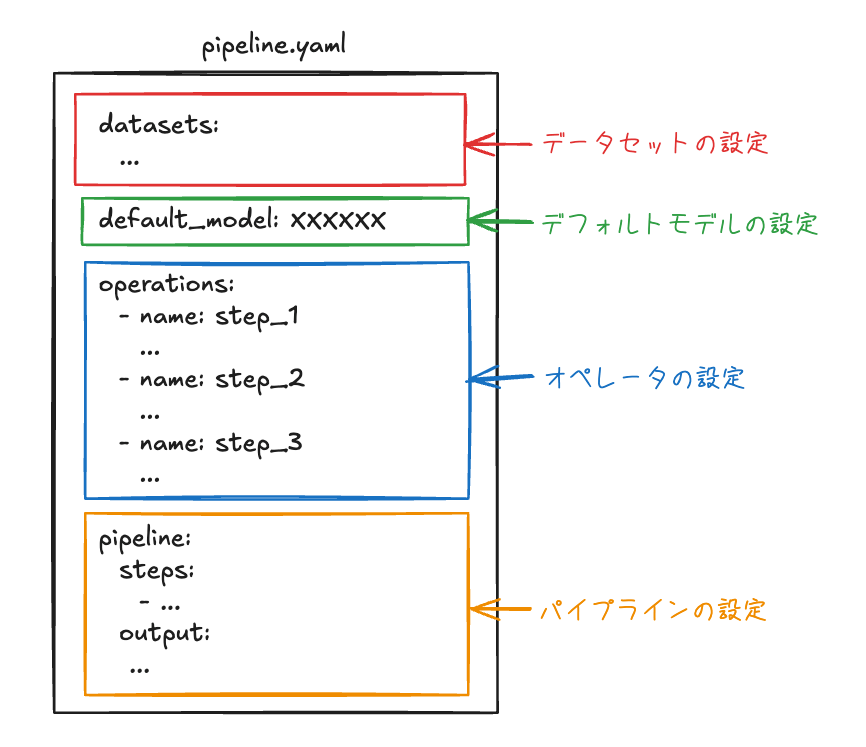
チュートリアルの例を参考に、それぞれを見ていく。
データセットの設定
データセットは、パイプラインで処理する入力データであり、個々のドキュメントを、JSONLのオブジェクト、もしくはCSVの行で記載する。
datasets:
transcripts:
path: medical_transcripts.json
type: file
上記の例では、medical_transcripts.jsonという「ファイル」をtranscriptsという名称で定義している。
上記以外にも、データを動的に(前)処理しながら入力データとして取り込む「Dynamic Data Loding」という機能もある様子だが、ここでは触れない。
デフォルトモデルの設定
デフォルトモデルの設定では、パイプラインの処理に使用するデフォルトのLLMを設定する。「デフォルト」とあるのは、DocETLでは各処理ごとにモデルを定義できるが、その定義がない場合に使用されるという意味。
default_model: gpt-4o-mini
オペレータの設定
オペレータは、データに対して実行される変換処理や分析処理の個々の定義。ここは後で細かく見るので割愛。チュートリアルの例ではざっくりこんな感じで定義されている。
operations:
- name: extract_medications # 薬名を抽出する処理
type: map
(snip)
- name: unnest_medications # 薬名の配列を辞書に変換する
type: unnest
(snip)
- name: resolve_medications # 薬名を標準化する処理
type: resolve
(snip)
- name: summarize_prescriptions # 薬の用途と副作用の要約を生成する処理
type: reduce
(snip)
パイプラインの設定
パイプラインの設定では以下の2つの設定を行う。
-
steps- オペレータの設定で定義された各オペレータをどの順序でデータに適用するか?
-
output- 最終的な結果を何に出力するか?
今回の例だと、
pipeline:
steps:
- name: medical_info_extraction
input: transcripts
operations:
- extract_medications
- unnest_medications
- resolve_medications
- summarize_prescriptions
output:
type: file
path: medication_summaries.json
intermediate_dir: intermediate_results
-
transcriptsに定義された入力データを、extract_medications->unnest_medications->resolve_medications->summarize_prescriptionsの順に処理する - 結果は
medication_summaries.jsonという「ファイル」に出力し、処理途中の中間データはintermediate_resultsディレクトリに出力する
という設定になる
オペレータ
データに対して具体的な処理を行うオペレータの定義について。
オペレータの属性
チュートリアルのオペレータの一つを例に取る。
- name: extract_medications
type: map
output:
schema:
medication: list[str]
prompt: |
以下の医師と患者の会話の記録を分析してください:
{{ input.src }}
会話記録に記載されているすべての薬剤を抽出し、リスト化してください。
薬剤が記載されていない場合は、空のリストを返してください。
個々のオペレータに共通する属性は以下。
-
name- オペレータの名前。ユニークなものである必要がある。
-
type- オペレータの種類。処理する内容に合わせて、用意されたものから選択する。
- LLMを使うオペレータと、LLM扶養で使えるオペレータがある
- LLMを使うもの:
map/reduce/filterなど - LLMを使わないもの:
split/gather/unnestなど
- LLMを使うもの:
で、LLMを使うオペレータタイプを使用する場合は以下の属性
-
prompt- LLMに渡すプロンプト。Jinja2のテンプレート記法で記述する。
-
output- LLMの出力結果をスキーマで定義する
-
model- 使用する言語モデル
- デフォルトモデルで指定したものとは別のものを使用する場合に定義する
先ほどの例だと、
- オペレータの名前は
extract_medications - オペレータの種類は
map。mapは入力されたデータのそれぞれに対してLLMを使って何かしらの変換などの処理を行うオペレータ。 - 結果は、
medicationという名前で、文字列の配列として出力する -
promptに、LLMに行わせる処理をJinja2テンプレートで記載。ここで、入力データがテンプレート内に展開される
という感じになっている。
オペレータの入出力
オペレータに渡される入力は
- パイプラインへの入力データ
- 1つ前のステップのオペレータで実行された結果
になる。例えば、一つ上で取り上げたオペレータは、パイプライン定義では以下のように、パイプラインの最初のステップになっている。
datasets:
transcripts:
path: medical_transcripts.json
type: file
(snip)
pipeline:
steps:
- name: medical_info_extraction
input: transcripts
operations:
- extract_medications # これ
(snip)
パイプラインの最初のステップでは、パイプラインへの入力データであるデータセットがオペレータへの入力となる。
[
{
"src": "医師: こんにちは、ジョンソンさん。新しい薬、リシノプリルを飲み始めてから、体調はいかがですか?\n患者: ええ、先生。血圧は改善されたように感じますが、乾いた咳が続いています..."
},
{
"src": "医師: おはようございます、スミスさん。今日はメトフォルミン処方の経過観察ですね。\n患者: はい、先生。規則正しく服用していますが、いくつかの副作用が気になっています..."
}
]
この入力データは、"map"により各データごとにinputとして渡され個別に処理される。プロンプトのテンプレートはこう。
prompt: |
以下の医師と患者の会話の記録を分析してください:
{{ input.src }}
会話記録に記載されているすべての薬剤を抽出し、リスト化してください。
薬剤が記載されていない場合は、空のリストを返してください。
つまり、実際に実行されるプロンプト、そして結果は以下のような感じになる。
以下の医師と患者の会話の記録を分析してください:
医師: こんにちは、ジョンソンさん。新しい薬、リシノプリルを飲み始めてから、体調はいかがですか?
患者: ええ、先生。血圧は改善されたように感じますが、乾いた咳が続いています...
会話記録に記載されているすべての薬剤を抽出し、リスト化してください。
薬剤が記載されていない場合は、空のリストを返してください。
→ 結果: 「リシノプリル」
以下の医師と患者の会話の記録を分析してください:
医師: おはようございます、スミスさん。今日はメトフォルミン処方の経過観察ですね。
患者: はい、先生。規則正しく服用していますが、いくつかの副作用が気になっています...
会話記録に記載されているすべての薬剤を抽出し、リスト化してください。
薬剤が記載されていない場合は、空のリストを返してください。
→ 結果: 「メトフォルミン」
そして、これらの出力を行うのが出力スキーマになる。
output:
schema:
medication: list[str]
今回はmedicationという名前で文字列の配列として出力されるが、mapは個別に処理するオペレータなので、こうなる。
{
"medication": ["リシノプリル"]
}
{
"medication": ["メトフォルミン"]
}
で、最終的な結果はこんな感じで出力されている様子。
[
{
"medication": "リシノプリル",
"src": "医師: こんにちは、ジョンソンさん。新しい薬、リシノプリルを飲み始めてから、体調はいかがですか?\n患者: ええ、先生。血圧は改善されたように感じますが、乾いた咳が続いています..."
},
{
"medication": "メトフォルミン",
"src": "医師: おはようございます、スミスさん。今日はメトフォルミン処方の経過観察ですね。\n患者: はい、先生。規則正しく服用していますが、いくつかの副作用が気になっています..."
}
]
これが次のオペレータへの入力となる。
mapでは、入力データに対して個別に処理を行うので、入力データを参照する際はinputとなるが、reduceでは集約して処理を行うため、複数形の"s"がついてinputsとなる。この場合はリストがそのまま渡されることになるので、以下のように、自分でループさせてテンプレート展開するらしい。
- name: summarize_prescriptions
type: reduce
reduce_key:
- medication
output:
schema:
side_effects: str
uses: str
prompt: |
以下は、医師と患者の間の会話の記録です:
{% for input in inputs %}
会話記録 {{ loop.index }}:
{{ input.src }}
{% endfor %}
(snip)
mapとreduceの違いはこんな感じかな?
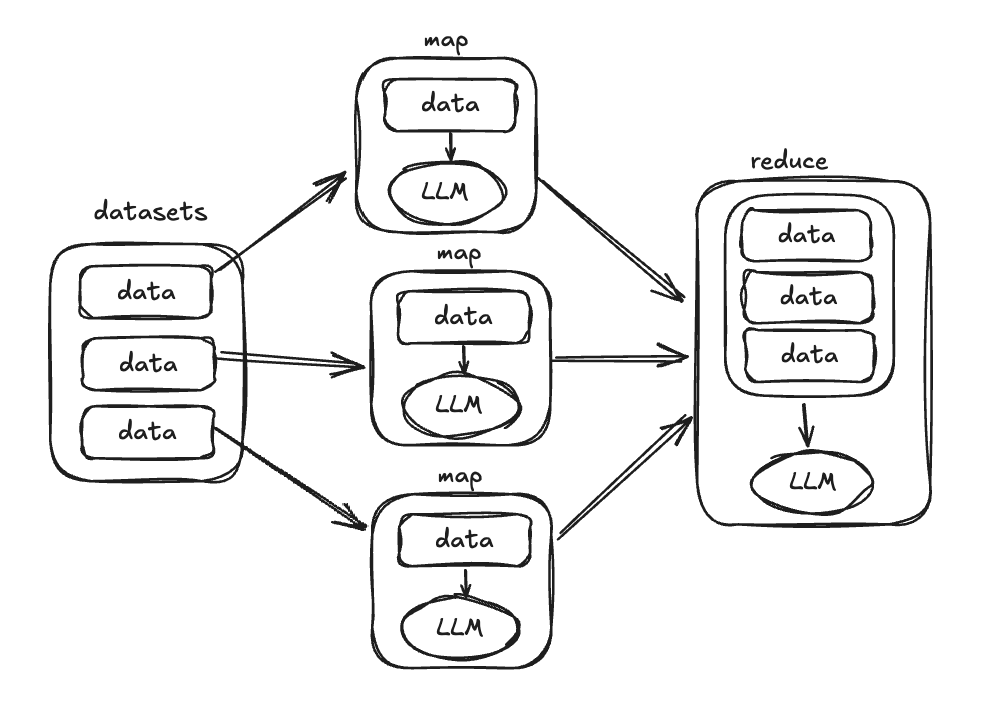
オペレータタイプは個別にリファレンスを見たほうが良さそう。
で、チュートリアルのパイプラインの例では使用されていないが、オペレータによって処理された結果を検証する仕組みがある。
例えばシンプルなものはvalidateで条件を指定する。
validate:
- len(output["insights"]) >= 2
- all(len(insight["supporting_actions"]) >= 1 for insight in output["insights"])
上記の例だと以下の条件を満たしているかをチェックする。
- 出力されたデータ
insightsの個数が2以上であること - 出力されたデータ
insightsに含まれるsupporting_actionsがすべて1以上であること
チェックをクリアできない場合、リトライさせることができる。
より高度なバリデーションとしてLLMを使うこともできる。
gleaning:
num_rounds: 1
validation_prompt: |
抽出されたデータについて、完全性と関連性を評価してください:
1. ログから得られたすべての主要なユーザー行動と問題点は、インサイトで取り上げられているか?
2. 補足的な行動は実際的であり、インサイトに関連しているか?
3. 重要な情報が抜け落ちていたり、関連性のない情報が含まれていたりしていないか?
このLLMを使ったバリデーションの処理は以下のように行われる
- 初期操作: LLMは元の操作プロンプトに基づいて初期出力を生成します。
- 検証: 検証プロンプトは、元の操作プロンプトと出力とともに、チャットスレッドに追加されます。 これはLLMに提出される。 検証プロンプトはチャットスレッドに追加されるため、変数を必要としないことに注意してください。
- 評価: LLMは検証プロンプトに従って出力の評価を応答します。
- 決定: システムは評価を解釈します:
- エラーや改善の余地がなければ、現在の出力が返されます。
- 改善が提案された場合、プロセスは続行される。
- 洗練化: 改善が必要な場合:
- 元の操作プロンプト、元の出力、およびバリデータのフィードバックを含む新しいプロンプトが作成される。
- これがLLMに提出され、改善された出力が生成される。
- 反復: ステップ2〜5を繰り返す:
- バリデータのフィードバックがなくなる (すなわち評価がパスする)、 あるいは
- 繰り返し回数が num_rounds を超える。
- 最終出力: 最後に洗練された出力が返される。
なるほど、検証するだけでなく、改善もできるのね。
出力スキーマ
上でも少し触れた出力スキーマだけども、出力スキーマはLLMを使ったオペレータにおいて出力フォーマットを定義するために使う。DocETLではStructured outputやTool useなどを使って、出力フォーマットを構造化している様子。
スキーマで定義できる型は以下。
-
string: 文字列 -
integer: 整数 -
number: 浮動小数 -
boolean: ブーリアン -
list: 配列。配列に入れるデータの型の指定も必要。 - オブジェクト:
{フィールド: 型}で指定。
など。例を見るほうがわかりやすいかも。
シンプルな例。
output:
schema:
summary: string
sentiment: string
include_item: boolean # `filter`オペレータで使える
複雑な例。オブジェクト型を含む場合はクォートする必要がある。
output:
schema:
insights: "list[{insight: string, confidence: number}]"
metadata: "{timestamp: string, source: string}"
ベストプラクティスにある通り、出力スキーマはシンプルにしたほうがいい。このあたりはStructured outputやTool useのプラクティスと同じ。
最適化
DocETLにはパイプラインの性能および精度を上げるために最適化の仕組みを備えている。例えばmapなんかがそれ。
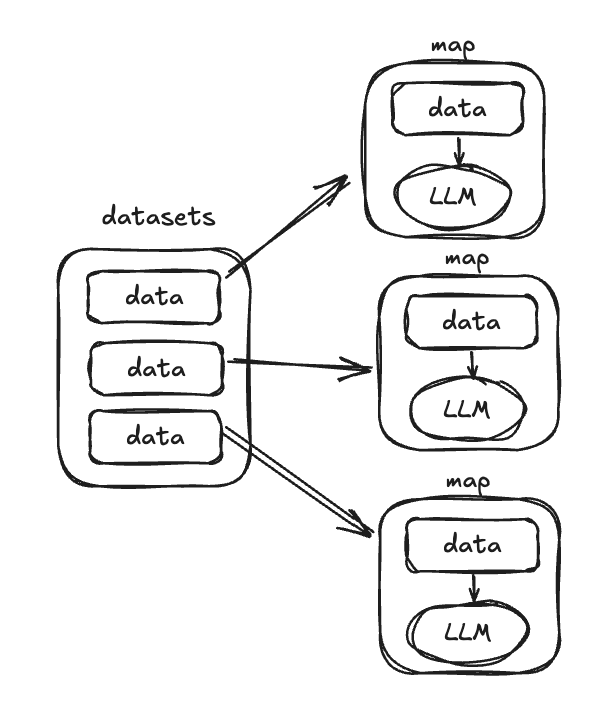
LLMを使う場合、処理の単位は小さくシンプルであるほうが、一般的には精度があがりやすく、並列で処理可能ならば並列でやるほうが処理時間も短くて済む。
これ以外にも内部的に性能を向上させるためのオプティマイザーがある。オプティマイザーは以下をやってくれる。
- 生成および評価エージェント: これらのエージェントが、定義済みの書き換えルールに従って、パイプラインのさまざまなプランを生成する。 評価エージェントはプランと出力を比較し、最適なアプローチを決定する。
-
オペレータの書き換え: オプティマイザーは、
optimize: trueを設定したパイプラインのオペレータを調べて、定義済みのルールを使用して書き換えを試みる。 - 出力: 最適化後、DocETL は最適化されたパイプラインを表す新しい YAML ファイルを出力する。
ということで元のpipeline.yamlのオペレータにoptimize: trueを追加してみる。
(snip)
operations:
- name: extract_medications
optimize: true
(snip)
- name: unnest_medications
optimize: true
(snip)
- name: resolve_medications
optimize: true
(snip)
- name: summarize_prescriptions
optimize: true
(snip)
(snip)
オプティマイザーを実行。
!docetl build pipeline.yaml
実行すると以下のような出力が表示される。おそらく最適化を行うためのサンプリング数なのだろうと思う。
───────────────────────────────────── Optimizer Configuration ──────────────────────────────────────
[05:23:47] Sample Size: {'reduce': 40, 'map': 5, 'resolve': 100, 'equijoin': 100,
そして、最適化後のYAMLファイルが、元のファイル名_opt.yamlとして生成される。
$ ls -lt pipeline*
-rw-r--r-- 1 root root 4505 Oct 3 06:44 pipeline_opt.yaml
-rw-r--r-- 1 root root 3252 Oct 3 06:44 pipeline.yaml
Unicodeエンコーディングされてるのでちょっとつらいのだけど。
import yaml
with open('pipeline_opt.yaml', encoding='utf-8')as file:
print(yaml.dump(yaml.safe_load(file), default_flow_style=False, allow_unicode=True))
datasets:
transcripts:
path: medical_transcripts.json
type: file
default_model: gpt-4o-mini
operations:
- name: extract_medications
optimize: true
output:
schema:
medication: list[str]
prompt: '以下の医師と患者の会話の記録を分析してください:
{{ input.src }}
会話記録に記載されているすべての薬剤を抽出し、リスト化してください。
薬剤が記載されていない場合は、空のリストを返してください。
'
type: map
- name: unnest_medications
optimize: true
type: unnest
unnest_key: medication
- blocking_keys:
- medication
blocking_threshold: 0.9899
comparison_prompt: '以下の2つの薬剤のエントリーを比較してください:
エントリー1: {{ input1.medication }}
エントリー2: {{ input2.medication }}
これらの薬剤が同じまたは密接に関連している可能性があるかどうかを判断してください。
'
embedding_model: text-embedding-3-small
name: resolve_medications
optimize: true
output:
schema:
medication: str
resolution_prompt: '以下の一致した薬剤のエントリーに基づいて:
{% for entry in inputs %}
エントリー{{ loop.index }}: {{ entry.medication }}
{% endfor %}
このグループのエントリーを最もよく表す標準化された薬剤名を決定してください。
標準化された名前は、すべての一致したエントリーを最もよく表す、広く認識された薬剤名であるべきです。
'
type: resolve
- name: summarize_prescriptions
optimize: true
output:
schema:
side_effects: str
uses: str
prompt: '以下は、医師と患者の間の会話の記録です:
{% for input in inputs %}
会話記録 {{ loop.index }}:
{{ input.src }}
{% endfor %}
薬剤{{ reduce_key }}について、上記の会話に基づいて以下の情報を提供してください:
1. 副作用: {{ reduce_key }}に関して言及されたすべての副作用を要約してください。
2. 治療用途: {{ reduce_key }}が処方または推奨された医療条件や症状を説明してください。
要約する際には以下を守ってください:
- 提供された記録の情報にのみ基づく
- {{ reduce_key }}にのみ焦点を当て、他の薬剤には触れない
- すべての記録から関連する詳細を含む
- 明確で簡潔に書く
- 記録からの引用を含める
'
reduce_key:
- medication
type: reduce
verbose: true
pipeline:
output:
intermediate_dir: intermediate_results
path: medication_summaries.json
type: file
steps:
- input: transcripts
name: medical_info_extraction
operations:
- extract_medications
- unnest_medications
- resolve_medications
- summarize_prescriptions
大きく書き換えられたところはないように思うのだけど、一箇所だけ。
blocking_threshold: 0.9899
ここは何かしら最適化の結果を踏まえて、パラメータが変更されたと思われる。
パイプライン作ってみたがイマイチ精度が良くない、とかの場合に試してみると良いのかもしれない。
以下にもドキュメントがある。
Python API
YAML、つまりローコードで書けるってのがDocETLのセールスポイントだと思うのだけど、Pythonでも書ける。
APIリファレンスはこちら
チュートリアルのYAMLをPythonに書き換えてみた。
from docetl.api import Pipeline, Dataset, MapOp, ReduceOp, UnnestOp, ResolveOp, PipelineStep, PipelineOutput
# Define datasets
datasets = {
"transcripts": Dataset(type="file", path="medical_transcripts.json"),
}
# Define operations
extract_medications_prompt = """\
以下の医師と患者の会話の記録を分析してください:
{{ input.src }}
会話記録に記載されているすべての薬剤を抽出し、リスト化してください。
薬剤が記載されていない場合は、空のリストを返してください。
"""
resolve_medications_comparison_prompt = """\
以下の2つの薬剤のエントリーを比較してください:
エントリー1: {{ input1.medication }}
エントリー2: {{ input2.medication }}
これらの薬剤が同じまたは密接に関連している可能性があるかどうかを判断してください。
"""
resolve_medications_resolution_prompt = """\
以下の一致した薬剤のエントリーに基づいて:
{% for entry in inputs %}
エントリー{{ loop.index }}: {{ entry.medication }}
{% endfor %}
このグループのエントリーを最もよく表す標準化された薬剤名を決定してください。
標準化された名前は、すべての一致したエントリーを最もよく表す、広く認識された薬剤名であるべきです。
"""
summarize_prescriptions_prompt = """\
以下は、医師と患者の間の会話の記録です:
{% for input in inputs %}
会話記録 {{ loop.index }}:
{{ input.src }}
{% endfor %}
薬剤{{ reduce_key }}について、上記の会話に基づいて以下の情報を提供してください:
1. 副作用: {{ reduce_key }}に関して言及されたすべての副作用を要約してください。
2. 治療用途: {{ reduce_key }}が処方または推奨された医療条件や症状を説明してください。
要約する際には以下を守ってください:
- 提供された記録の情報にのみ基づく
- {{ reduce_key }}にのみ焦点を当て、他の薬剤には触れない
- すべての記録から関連する詳細を含む
- 明確で簡潔に書く
- 記録からの引用を含める
"""
operations = [
MapOp(
name="extract_medications",
type="map",
prompt=extract_medications_prompt,
output={"schema": {"medication": "list[string]"}}
),
UnnestOp(
name="unnest_medications",
type="unnest",
unnest_key="medication",
),
ResolveOp(
name="resolve_medications",
type="resolve",
blocking_keys=["medication"],
blocking_threshold=0.6162,
comparison_prompt=resolve_medications_comparison_prompt,
embedding_model="text-embedding-3-small",
output={"schema": {"medication": "string"}},
resolution_prompt=resolve_medications_resolution_prompt,
),
ReduceOp(
name="summarize_prescriptions",
type="reduce",
reduce_key="medication",
prompt=summarize_prescriptions_prompt,
output={"schema": {"side_effects": "string", "side_effects": "string"}}
)
]
# Define pipeline steps
steps = [
PipelineStep(name="extract_step", input="transcripts", operations=["extract_medications"]),
PipelineStep(name="unnest_step", input="extract_step", operations=["unnest_medications"]),
PipelineStep(name="resolve_step", input="unnest_step", operations=["resolve_medications"]),
PipelineStep(name="summarize_step", input="resolve_step", operations=["summarize_prescriptions"]),
]
# Define pipeline output
output = PipelineOutput(type="file", path="medication_summaries.json", intermediate_dir="intermediate_results")
# Create the pipeline
pipeline = Pipeline(
name="medical_info_extraction",
datasets=datasets,
operations=operations,
steps=steps,
output=output,
default_model="gpt-4o-mini"
)
# Optimize the pipeline
optimized_pipeline = pipeline.optimize()
# Run the optimized pipeline
result = optimized_pipeline.run() # Saves the result to the output path
print(f"Pipeline execution completed. Total cost: ${result:.2f}")
実行すると、こんな感じで処理状況が出力される。
───────────────────────────────────────────── Optimizer Configuration ─────────────────────────────────────────────
[07:32:40] Sample Size: {'reduce': 40, 'map': 5, 'resolve': 100, 'equijoin': 100, 'filter': 5, builder.py:209
'split': 100, 'gather': 100}
Max Threads: 8 builder.py:210
Model: gpt-4o-2024-08-06 builder.py:211
Timeout: 60 seconds builder.py:212
───────────────────────────────────────── Beginning Pipeline Optimization ─────────────────────────────────────────
Syntax check passed for all operations. builder.py:194
Processing map items: 100%|██████████| 2/2 [00:00<00:00, 2.87it/s]
[07:32:41] Flushing cache to disk... utils.py:116
Cache flushed to disk. utils.py:118
Saved intermediate results to disk at builder.py:56
/root/.docetl/cache/medical_info_extraction/5ddc93c6b1435fcbf6d0b91bdc6bf1bd.json
Loading dataset from disk... 5ddc93c6b1435fcbf6d0b91bdc6bf1bd.json builder.py:50
Flushing cache to disk... utils.py:116
Cache flushed to disk. utils.py:118
Saved intermediate results to disk at builder.py:56
/root/.docetl/cache/medical_info_extraction/2b624ec44d6ac8d6578e4224bcebc630.json
Loading dataset from disk... 2b624ec44d6ac8d6578e4224bcebc630.json builder.py:50
[07:32:42] Comparisons saved by blocking: 0 (0.00%) resolve.py:343
Processing batches of 100 LLM comparisons: 100%|██████████| 1/1 [00:00<00:00, 1268.31it/s]
Number of keys before resolution: 2 resolve.py:440
Number of distinct keys after resolution: 1 resolve.py:441
Determining resolved key for each group of equivalent keys: 100%|██████████| 1/1 [00:00<00:00, 1.30it/s]
Self-join selectivity: 1.0000 resolve.py:468
Flushing cache to disk... utils.py:116
Cache flushed to disk. utils.py:118
Saved intermediate results to disk at builder.py:56
/root/.docetl/cache/medical_info_extraction/60f974a12282ee10a47d86b0f19d1abe.json
Loading dataset from disk... 60f974a12282ee10a47d86b0f19d1abe.json builder.py:50
Processing reduce items: 100%|██████████| 1/1 [00:00<00:00, 1.01it/s]
[07:32:43] Flushing cache to disk... utils.py:116
Cache flushed to disk. utils.py:118
Saved intermediate results to disk at builder.py:56
/root/.docetl/cache/medical_info_extraction/d407cd2b255730461564914952847093.json
Total agent cost: $0.00 builder.py:596
Total operations cost: $0.00 builder.py:599
Total cost: $0.00 builder.py:602
────────────────────────────────────────────────── Syntax Check ───────────────────────────────────────────────────
Performing syntax check on all operations...
Syntax check passed for all operations.
─────────────────────────────────────────────── Pipeline Execution ────────────────────────────────────────────────
──────────────────────────────────────────────── Loading Datasets ─────────────────────────────────────────────────
Loaded dataset: transcripts
────────────────────────────────────────── Executing Step: extract_step ───────────────────────────────────────────
Running Operation:
Type: map
Name: extract_medications
Processing map items: 100%|██████████| 2/2 [00:00<00:00, 347.17it/s]
[07:32:43] Operation extract_medications completed. Cost: $0.00 runner.py:245
✓ Intermediate saved for operation 'extract_medications' in step 'extract_step' at
intermediate_results/extract_step/extract_medications.json
Flushing cache to disk... utils.py:116
[07:32:44] Cache flushed to disk. utils.py:118
Step extract_step completed. Cost: $0.00 runner.py:137
─────────────────────────────────────────── Executing Step: unnest_step ───────────────────────────────────────────
Running Operation:
Type: unnest
Name: unnest_medications
Operation unnest_medications completed. Cost: $0.00 runner.py:245
✓ Intermediate saved for operation 'unnest_medications' in step 'unnest_step' at
intermediate_results/unnest_step/unnest_medications.json
Flushing cache to disk... utils.py:116
Cache flushed to disk. utils.py:118
Step unnest_step completed. Cost: $0.00 runner.py:137
────────────────────────────────────────── Executing Step: resolve_step ───────────────────────────────────────────
Running Operation:
Type: resolve
Name: resolve_medications
Comparisons saved by blocking: 1 (100.00%) resolve.py:343
Processing batches of 100 LLM comparisons: 0it [00:00, ?it/s]
Number of keys before resolution: 2 resolve.py:440
Number of distinct keys after resolution: 2 resolve.py:441
Determining resolved key for each group of equivalent keys: 100%|██████████| 2/2 [00:00<00:00, 3572.66it/s]
Self-join selectivity: 0.0000 resolve.py:468
Operation resolve_medications completed. Cost: $0.00 runner.py:245
✓ Intermediate saved for operation 'resolve_medications' in step 'resolve_step' at
intermediate_results/resolve_step/resolve_medications.json
Flushing cache to disk... utils.py:116
Cache flushed to disk. utils.py:118
Step resolve_step completed. Cost: $0.00 runner.py:137
───────────────────────────────────────── Executing Step: summarize_step ──────────────────────────────────────────
Running Operation:
Type: reduce
Name: summarize_prescriptions
Processing reduce items: 0%| | 0/2 [00:00<?, ?it/s]
Processing reduce items: 100%|██████████| 2/2 [00:02<00:00, 1.01s/it]
[07:32:46] Operation summarize_prescriptions completed. Cost: $0.00 runner.py:245
✓ Intermediate saved for operation 'summarize_prescriptions' in step 'summarize_step' at
intermediate_results/summarize_step/summarize_prescriptions.json
Flushing cache to disk... utils.py:116
Cache flushed to disk. utils.py:118
Step summarize_step completed. Cost: $0.00 runner.py:137
────────────────────────────────────────────────── Saving Output ──────────────────────────────────────────────────
💾 Output saved to medication_summaries.json
──────────────────────────────────────────────── Execution Summary ────────────────────────────────────────────────
Total cost: $0.00
Total time: 2.49 seconds
Pipeline execution completed. Total cost: $0.00
結果はYAMLでやったときと同じようにoutputで定義したファイルに出力される。
オペレータの定義の各ステップの結果はintermediate_dirで指定したディレクトリに出力される。
$ tree intermediate_results/
intermediate_results/
└── medical_info_extraction
├── extract_medications.json
├── resolve_medications.json
├── summarize_prescriptions.json
└── unnest_medications.json
1 directory, 4 files
こんな感じで順に内容が変わっていってるのがわかる。
[
{
"medication": [
"リシノプリル"
],
"src": "医師: こんにちは、ジョンソンさん。新しい薬、リシノプリルを飲み始めてから、体調はいかがですか?\n患者: ええ、先生。血圧は改善されたように感じますが、乾いた咳が続いています..."
},
{
"medication": [
"メトフォルミン"
],
"src": "医師: おはようございます、スミスさん。今日はメトフォルミン処方の経過観察ですね。\n患者: はい、先生。規則正しく服用していますが、いくつかの副作用が気になっています..."
}
]
[
{
"medication": "リシノプリル",
"src": "医師: こんにちは、ジョンソンさん。新しい薬、リシノプリルを飲み始めてから、体調はいかがですか?\n患者: ええ、先生。血圧は改善されたように感じますが、乾いた咳が続いています..."
},
{
"medication": "メトフォルミン",
"src": "医師: おはようございます、スミスさん。今日はメトフォルミン処方の経過観察ですね。\n患者: はい、先生。規則正しく服用していますが、いくつかの副作用が気になっています..."
}
]
[
{
"medication": "リシノプリル",
"src": "医師: こんにちは、ジョンソンさん。新しい薬、リシノプリルを飲み始めてから、体調はいかがですか?\n患者: ええ、先生。血圧は改善されたように感じますが、乾いた咳が続いています..."
},
{
"medication": "メトフォルミン",
"src": "医師: おはようございます、スミスさん。今日はメトフォルミン処方の経過観察ですね。\n患者: はい、先生。規則正しく服用していますが、いくつかの副作用が気になっています..."
}
]
[
{
"side_effects": "この会話において、リシノプリルの副作用として「乾いた咳」が言及されています。",
"uses": "リシノプリルは血圧を改善するために処方されており、患者は「血圧は改善されたように感じます」と述べています。",
"medication": "リシノプリル"
},
{
"side_effects": "メトフォルミンに関して、スミスさんは「いくつかの副作用が気になっています」と述べていますが、具体的な副作用の詳細は記録には明記されていません。",
"uses": "メトフォルミンはスミスさんに処方され、経過観察が行われています。これは主に糖尿病治療に用いられる薬剤です。",
"medication": "メトフォルミン"
}
]
最後のやつは最終結果と同じになる。
DocETLではデフォルトでキャッシュが有効になっているため、同じデータに対して同じ処理を実行したとした場合、キャッシュの結果が利用される。
キャッシュを削除するには以下を実行する。
!docetl clear
キャッシュはホームディレクトリ直下の.docetl/cacheと.docetl/llm_cacheに保存されている。
まとめ
最初見たときは、「文書処理」とあったので、PDFファイルあたりを処理するためのパイプラインが作れるのかなーと思って、過去に試したUnstractと同じようなものをイメージしてた。
ただ、Unstractは、明確に「ファイル」を入力データの対象としているように思えるのだけど、DocETLはもう少し大きな「データ」というイメージで、現時点では入力データはJSON/CSVとなっている。この点から、「文書」というよりは、もう少し広範な「データパイプライン」「ワークフローエンジン」のほうに近いのかなーと感じた。
ただ、何かしらの処理パイプラインを作れるツールは昔からいろいろあって、最近はこういうのもLLM対応しているものが増えていると思う。
以下で色々調べてみたけど、いくつかはLLM対応している様子
試せてないけどリアルタイム性がウリ
この手のツールは、
- 処理タスクを柔軟 or 簡単 に定義できる
- パイプラインの実行が効率的に行える
- 入出力のデータソースが豊富
みたいなところがウリだと思っていて、その点を踏まえると、現時点で入出力にJSON/CSVしか使えないDocETLはちょっと見劣りする気がするし、大量の「Unstructured」なデータ≒ファイルが多そう、と思うと、Unstractのほうがユースケースにピンポイントにマッチする場合も多そう。
あとローコードってのもウリなのかもしれなけど、YAMLで定義できるようなものは他にもあるのではないかと思うので、それほど大きな要素ではない気がする。
このあたりを踏まえて、DocETLのメリットってなんだろう?DocETLのデモを見てみる。
このパイプラインは、1960年までさかのぼる米国大統領討論会のテーマを分析し、テーマごとに民主党と共和党の見解の変遷を要約する。実行には0.29ドルかかった(最適化には0.86ドル)。
合計738,094語に及ぶ討論の記録を1回のプロンプトで分析するのは困難である。例えば、Gemini-1.5-Pro-002(2024年9月24日リリース)という1回のプロンプトで全データセットが与えられた場合、5つのテーマの進化(全ドキュメントを通じて)についてのみレポートされる。
DocETLは、民主党と共和党の見解の変遷を時系列で分析した152のテーマ別の包括的なレポートを作成する。ドロップダウンメニューからテーマを選択してレポートを閲覧できる。
なるほど、ロングコンテキストLLMに全部食わせるよりも、品質高い結果が得られると。
そして、開発者の方のポッドキャスト。
以下NotebookLMに読ませてみた。ポイントとなるところを太字にしている。
DocETLの強み
ソースに基づいてDocETLLの強みを説明します。
- DocETLは、LLMを活用したデータ処理パイプラインを構築および最適化するための宣言型フレームワークです。 つまり、ユーザーは、高レベルで操作のプロンプトを指定するだけで済みます。DocETLは、これらのプロンプトを書き換え、LLMが正確に実行できる小さな単位にデータをチャンクアップし、結果をつなぎ合わせます。
- DocETLは、インタラクティブに設計されています。 ユーザーは、最初の出力を確認してからプロンプトを調整できるため、最終的な出力に対する制御が強化されます。
- DocETLは、マップ、リデュースなど、一般的なデータ処理フレームワークと同様の操作タイプをサポートしています。 これらの操作はプロンプトによって強化されているため、ユーザーは自然言語でタスクを指定できます。
- DocETLは、タスク固有の検証プロンプトを合成することにより、パイプラインの自動評価をサポートしています。 これにより、LLMを使用して、精度、再現率、およびその他のタスク固有のメトリックに関してパイプライン出力を評価できます。
- DocETLは、フォールトトレランスのために複数のエージェントとメカニズムを使用する、エージェントベースのシステムです。 これにより、LLMの非決定的な性質にもかかわらず、DocETLが確実に動作することが保証されます。
- DocETLは、さまざまなドメインやタスクに柔軟に対応できます。 例えば、警察の不正行為の識別、製品レビューのマイニング、コースレビューの分析などに使用されています。
ソースではDocETLの具体的な強みは列挙されていませんが、上記の情報から、DocETLが複雑なデータ処理タスクを自動化するための強力で柔軟なフレームワークであることがわかります。
既存のパイプライン構築ツールと比較したDocETLの利点
ソースは、既存のパイプライン構築ツールと比較したDocETLの利点について明確に述べていません。しかし、ソースやこれまでの会話から、DocETLと従来のツールとの違いをいくつか推測することができます。
- LLMを活用した宣言型アプローチ: DocETLは、LLMを活用したデータ処理パイプラインを構築および最適化するための宣言型フレームワークであることが特徴です。これは、ユーザーが高レベルで操作のプロンプトを指定するだけで、DocETLが残りの処理を処理することを意味します。従来のツールは、より明示的なプログラミングを必要とする場合があり、特に複雑なパイプラインを扱う際には、より多くの専門知識と手作業が必要になります。
- インタラクティブ性と人間参加: DocETLは、人間参加とインタラクティブなパイプライン構築に重点を置いています。ユーザーは、初期出力を確認し、必要に応じてプロンプトを調整して、LLMの動作をガイドできます。これは、従来のツールでは、パイプラインの変更を実装するために、より複雑な再プログラミングが必要になる場合があるのとは対照的です。
- **自動評価と最適化: DocETLは、タスク固有の検証プロンプトを合成することにより、パイプラインの自動評価をサポートしています。これにより、ユーザーは、精度、再現率、その他のタスク固有のメトリックに関して、LLMを使用してパイプラインの出力を評価できます。さらに、DocETLは、さまざまなプランを生成および評価し、最適なパイプラインを自動的に見つける最適化機能を提供します。これらの機能は、従来のツールでは一般的ではない場合があり、手動による評価と最適化が必要になる場合があります。
- 柔軟性とドメイン適合性: ソースは、DocETLがさまざまなドメインやタスクにわたって効果的であることを示しています。警察の不正行為の識別、製品レビューのマイニング、コースレビューの分析など、幅広いユースケースに対応できます。この柔軟性は、LLMを使用する宣言型アプローチと、ユーザーが独自のタスク固有のプロンプトと検証基準を提供できる機能の組み合わせによるものです。
ただし、DocETLはまだ開発段階のプロトタイプであり、広く採用されている従来のツールと比較して、制限がある可能性があります。たとえば、ソースは、DocETLが主に非構造化データの処理に焦点を当てており、構造化データと非構造化データの両方を処理する必要があるユースケースには適していない可能性があることを示唆しています。さらに、DocETLのパフォーマンスとスケーラビリティは、大規模な実世界のデータセットでまだ徹底的に評価されていません。
要約すると、ソースはDocETLを、LLMを活用した宣言型アプローチ、インタラクティブなパイプライン構築、自動評価と最適化、幅広いドメインやタスクへの柔軟性など、いくつかの潜在的な利点を備えた有望なデータ処理パイプライン構築ツールとして提示しています。ただし、DocETLはまだ比較的新しいツールであるため、従来のツールと比較した相対的な成熟度とパフォーマンスを完全に評価するには、さらなる調査と開発が必要です。
なるほど、出力検証・評価やパイプライン最適化の仕組みがあるので、結果の精度や品質というところに強みがあるというのがウリになるのか。エージェントが採用されているというところもポイントかも。
実際のユースケースでの出力結果を評価しないと判断はできないけれども、なかなか良さそうである。既存のデータパイプライン・ワークフロー構築ツールでは後付でLLM対応されているケースも多いだろうけど、LLM前提で新しく作られたということもメリットなのかもしれない。
ちなみに一番最初のリリースから2週間しか経っていない上、以下のような入力データの対応も増えつつあるようなので、今後も期待できそう。
LLMへのアクセスはLiteLLMを使用しているようなので、Ollamaあたりも使える。以下にexampleがある。
exampleは他にも例があるので参考になりそう。
プロンプトジェネレータについての議論で、作者の方のコメント。DocETLについても触れられている。
論文はこちら
Claude-3.5-sonnetによる落合プロンプトの結果
DocETL: 複雑な文書処理のためのエージェント的クエリ書き換えと評価システム
どんなもの?
DocETLは、大規模言語モデル(LLM)を使用して複雑な文書を効率的に処理するための宣言的フレームワークです。従来のLLMベースの文書処理システムは、コストの削減に重点を置いていましたが、DocETLは精度の向上に焦点を当てています。このシステムの特徴は、文書の内容や処理タスクが複雑な場合でも、高い精度で情報を抽出・分析できることです。例えば、警察の記録から不正行為のパターンを特定したり、数百ページに及ぶ法的文書から特定の条項を識別したりすることができます。2024年10月時点で、GitHubで800以上のスターを獲得し、法律、医療、気候科学などの分野で活用されています。
先行研究と比べてどこがすごい?
従来のシステム(LOTUS、Palimpzest、Arynなど)は、ユーザーが定義した操作をそのまま実行し、コスト削減に重点を置いていました。これに対してDocETLの革新的な点は以下の3つです。第一に、複雑な操作を単純で正確な操作の連続に分解する新しい書き換えディレクティブを13種類導入しました。第二に、LLMエージェントを活用して、タスク固有の検証プロンプトを自動生成し、出力品質を評価します。第三に、機会主義的な最適化戦略を採用し、正確性が不十分な操作を特定して分解します。評価実験では、手作業で設計したベースラインと比較して、1.34から4.6倍高品質な出力を達成しました。
技術や手法の肝はどこ?
DocETLの核となる技術は3つあります。1つ目は、文書分割、射影合成、LLM中心の改善という3つのカテゴリーに分類される13種類の書き換えディレクティブです。これらは、長い文書を適切なサイズに分割したり、複雑な操作をより単純な操作に分解したりする際の指針となります。2つ目は、エージェントベースの評価システムで、LLMを使用してタスク固有の検証プロンプトを生成し、出力の品質を評価します。3つ目は、機会主義的な最適化アプローチで、操作が十分に正確でない場合に、その操作を再帰的に分解・最適化します。これらの技術により、複雑な文書処理タスクを高精度で実行することが可能になっています。
どうやって有効だと検証した?
DocETLの有効性は、3つの異なる非構造化文書分析タスクで検証されました。1つ目は警察の不正行為識別タスクで、227の文書を分析し、ベースラインと比較して1.34倍の精度向上を達成しました。2つ目はビデオゲームレビューの分極化特徴分析で、500のゲームの約40万件のレビューを分析し、引用と異なるゲームからの裏付けを含む報告を生成しました。3つ目は機密解除文書の分析で、733の記事を処理し、ベースラインと比較して1.82倍包括的な情報抽出を実現しました。さらに、先行研究のLOTUSとPalimpzestで提案されたタスクでも評価を行い、それぞれ16-21%と18%の性能向上を示しました。
議論はある?
DocETLの最適化プロセスには、いくつかの課題と限界があることが議論されています。第一に、AIエージェントに依存する最適化アプローチは、エージェントの能力に制限されるため、必ずしも最適な結果を得られない可能性があります。第二に、パイプラインの要件定義における不確実性の問題があり、ユーザーは初期出力を見てから要件が明確になることが多いとされています。これらの課題に対して、人間とシステムの協調的なアプローチや、ユーザーによる検証プロンプトの直接的な影響力の付与などの解決策が提案されています。また、システムの拡張性向上や、ローカルLLMとの互換性確保なども今後の課題として挙げられています。
次に読むべき論文は?
- "LOTUS: Enabling Semantic Queries with LLMs Over Tables of Unstructured and Structured Data" (Patel et al., 2024) - LLMを用いたデータ処理フレームワークの代表的な研究です。
- "A Declarative System for Optimizing AI Workloads" (Liu et al., 2024) - AIワークロードの最適化に関する宣言的システムについて説明しています。
- "Lost in the Middle: How Language Models Use Long Contexts" (Liu et al., 2024) - LLMの長文処理における課題について分析しています。
- "DSPy: Compiling Declarative Language Model Calls into State-of-the-Art Pipelines" (Khattab et al., 2024) - 宣言的なLLMコールの最適化に関する研究です。
- "Spade: Synthesizing assertions for large language model pipelines" (Shankar et al., 2024) - LLMパイプラインのための検証手法を提案しています。
13種類の書き換えディレクティブについて教えて
データ分解(Data Decomposition)カテゴリ
- 文書チャンキング(Document Chunking)
- 長い文書をLLMの処理可能なサイズに分割
- Split → Gather → Map → Reduce の形式で実行
- 例:文書を15,685トークンのチャンクに分割し、各チャンクに前後のコンテキストを付加して処理
- マルチレベル集約(Multi-Level Aggregation)
- データをより細かい粒度で段階的に集約
- Reduce𝐾,𝑥 ⇒ Reduce𝐾∪𝐾′,𝑦 → Reduce𝐾,𝑧 の形式
- 例:投票パターンを市町村レベルで集約した後、州レベルで集約
LLM中心の改善(LLM-Centric Improvements)カテゴリ
- グリーニング(Gleaning)
- LLMの出力を反復的に改善
- 検証エージェントがフィードバックを提供し、データ処理LLMが出力を改善
- 最大k回の反復を実行
- 重複キー解決(Duplicate Key Resolution)
- 意味的に同じだが表記が異なるキーを統一
- 例:「New York City」「NYC」「The Big Apple」を同一エンティティとして処理
射影合成(Projection Synthesis)カテゴリ
- チェイニング(Chaining)
- 複雑な操作を単純な操作の連鎖に分解
- Map𝑥 ⇒ Map𝑥1 → Map𝑥2 → ··· → Map𝑥𝑛 の形式
- 分離(Isolating)
- 独立した複数のサブタスクに分割して並列処理
- Map𝑦 ⇒ (Map𝑦1 ∥ Map𝑦2 ∥ ··· ∥ Map𝑦𝑚) → Reduce
- 事前集約(Pre-Aggregation)
- Reduce操作の前に関連データを抽出・準備
- Reduce𝐾,𝑥 ⇒ Map𝑦 → Reduce𝐾,𝑧
- 事前結合(Pre-Joining)
- 結合操作の前にデータを前処理
- Equijoin𝑥 ⇒ (Map𝑦,𝐿 ∥ Map𝑧,𝑅) → Equijoin𝑤
その他の補助的ディレクティブ
- メタデータ抽出(Metadata Extraction)
- 文書全体に関連するメタデータを事前に抽出
- ヘッダー系統の保持(Header Lineage)
- 文書の階層構造を維持するためのヘッダー情報の処理
- チャンクフィルタリング(Chunk Filtering)
- 関連性の低いチャンクを除外
- 入れ子結果のフラット化(Flattening Nested Results)
- 階層的な結果を単一のコレクションに変換
- バッチサイズ最適化(Batch Size Optimization)
- Reduce操作のバッチサイズを最適化
- 実験的に最適なバッチサイズを決定
これらのディレクティブは、単なるルールではなく、特定のタスクや文書の特性に応じてLLMエージェントが解釈・実装する抽象的なフレームワークとして機能します。各ディレクティブは無限の具体的な実装可能性を持ち、LLMエージェントがタスクに応じて最適な実装を選択します。
DocETLは文書処理パイプラインのように思えるのだが、なぜタイトルに「Query Rewriting」とあるのだろう?
「Query Rewriting」という用語がタイトルに含まれている理由について、以下のように分析できます:
1. データベース分野の伝統との関連
- DocETLは、データベース分野の伝統的なクエリ最適化の概念を拡張しています。
- 従来のデータベースでは、SQLクエリを書き換えて最適化を行いますが、DocETLは同様のアプローチを文書処理に適用しています。
2. 「クエリ」の解釈の拡張
- ここでの「クエリ」は、従来のSQL問い合わせだけでなく、文書に対する処理要求全般を指します。
- ユーザーが定義した処理操作(map、reduce、filter等)を「クエリ」として扱います。
3. 書き換えの本質
- DocETLの核となる機能は、複雑な処理要求(クエリ)を、より単純で効率的な操作の連鎖に「書き換える」ことです。
- 例:単一の大規模なmap操作を、split → gather → map → reduceという一連の操作に書き換えます。
4. 最適化の方法論
- 伝統的なクエリ最適化と同様に、入力された処理要求を別の等価な形式に書き換えることで最適化を行います。
- 13種類の書き換えディレクティブは、まさにこのクエリ書き換えのためのルールセットとして機能します。
5. システムの位置づけ
- DocETLは単なるパイプラインシステムではなく、処理要求を最適化するクエリオプティマイザとしての役割を持っています。
- 最適化の過程で、入力された処理要求を様々な形式に書き換えて、最も効果的な実行プランを見つけ出します。
つまり、「Query Rewriting」という用語は、このシステムの本質的な機能—処理要求の最適化された形式への変換—を強調するために使用されていると考えられます。これは、単にパイプラインを実行するだけでなく、その処理方法自体を最適化する高度な機能を持つシステムであることを示しています。
LLMにあたえるプロンプト≒クエリっていうわけではないってことかな?
その通りです。DocETLにおける「Query」は、個々のプロンプトのレベルではなく、より高次の処理要求を指しています。以下に詳しく説明します:
DocETLにおける「Query」の意味
- 個別のLLMプロンプト ≠ Query
- Query = ユーザーが定義した処理パイプライン全体
- map、reduce、filter等の操作の組み合わせ
- YAMLで記述された処理フロー全体
- 各操作に関連するスキーマや設定を含む
プロンプトとQueryの関係
- プロンプトは各操作の実装詳細の一部
- 一つのQueryの中に複数のプロンプトが含まれる
- プロンプトはQueryが書き換えられる過程で自動的に生成・修正される
具体例
- 元のQuery:「文書から警察の不正行為を抽出するmap操作」
- 書き換え後:
- 文書を分割するsplit操作
- コンテキストを付加するgather操作
- 各チャンクから情報を抽出するmap操作(新しいプロンプト)
- 結果を統合するreduce操作(別のプロンプト)
システムの役割
- 高レベルの処理要求(Query)を受け取る
- それを最適な実行可能形式に書き換える
- 書き換えの過程で必要なプロンプトを自動生成
- 各操作の効果を検証するためのプロンプトも自動生成
このように、DocETLでは「Query」は処理要求全体を指し、プロンプトはその実装の一部として扱われます。「Query Rewriting」は、処理要求全体の構造を最適化する過程を指しており、その過程でプロンプトの生成や修正も行われるという階層的な関係になっています。
