smina@macのインストールと使い方
OSSのdocking programといえばvinaですが、インストールやpdbqtファイル作成などの前処理が結構めんどくさいです。
sminaやgninaはvinaのfolkで、pdbやsdfファイルを使ってシンプルにdocking studyが出来るので、アカデミアや初学者の方にはオススメです。
ここではMacOSへのsminaのインストールと使い方をガイドします。windowsのことはわからないのでごめんなさい🙇
sminaのインストール
mac版のsminaはこちらからバイナリファイル(=CLIで実行できるアプリケーション)をダウンロードすることができます。多分クリックするとすぐにダウンロードが始まります。
適当な場所に移してパスを通しておくと、terminalからコマンドでdockingを実行できるので便利です。macやLinuxなどのUNIX系のOSの方は下記を参考にしてください。
mkdir ~/Documents/apps
mkdir ~/Documents/apps/smina
mv ~/Downloads/smina.osx ~/Documents/apps/smina
cd ~/Documents/apps/smina
mv smina.osx smina
ln -s ~/Documents/apps/smina /usr/local/bin/.
パスについてよくわからない方はこちらが参考になります。
下記のwhichコマンドで/usr/local/bin/sminaのように出力されれば、パスがちゃんと通っています。
which smina
# /usr/local/bin/smina
同じように、sminaという文字列のみterminalで実行して、sminaのヘルプが出力されればsminaコマンドが使えるようになっています。
sminaの基本使用
sminaはコマンドラインインターフェイス(CLI)で実行するアプリケーションです。
こちらのDocumentを参考に実行することができます。
サンプルコマンドはこんな感じです。
smina -r receptor.pdb -l ligand.pdb --autobox_ligand ligand.pdb -o docked.sdf --num_modes 1
続いて、既知のリガンド共結晶構造を使って上記のサンプルコードでdockingしてみます。
今回は6O0Pという共結晶構造についてテストしてみたいと思います。
標的とリガンドの背景については勉強しておきます。。とにかく大きいリガンドを試してみたかっただけです。。
下記のコマンドを実行してreceptorとligandのファイルをそれぞれ準備します。
mkdir ~/Documents/pdb/6o0p # お好きなdirectoryを作ってください。計算結果がここに出力されます。
cd ~/Documents/pdb/6o0p
wget https://files.rcsb.org/download/6O0P.pdb
grep ATOM 6O0P.pdb > rec.pdb
grep HETATM 6O0P.pdb > lig.pdb
ls # 6O0P.pdb lig.pdb rec.pdb
これで一応下準備は完了です。
lsでrec.pdbとlig.pdbが出来ていることを確認してください。
PyMOLを使ってどんなファイルが生成されているかも確認しておきましょう。
pymol lig.pdb rec.pdb
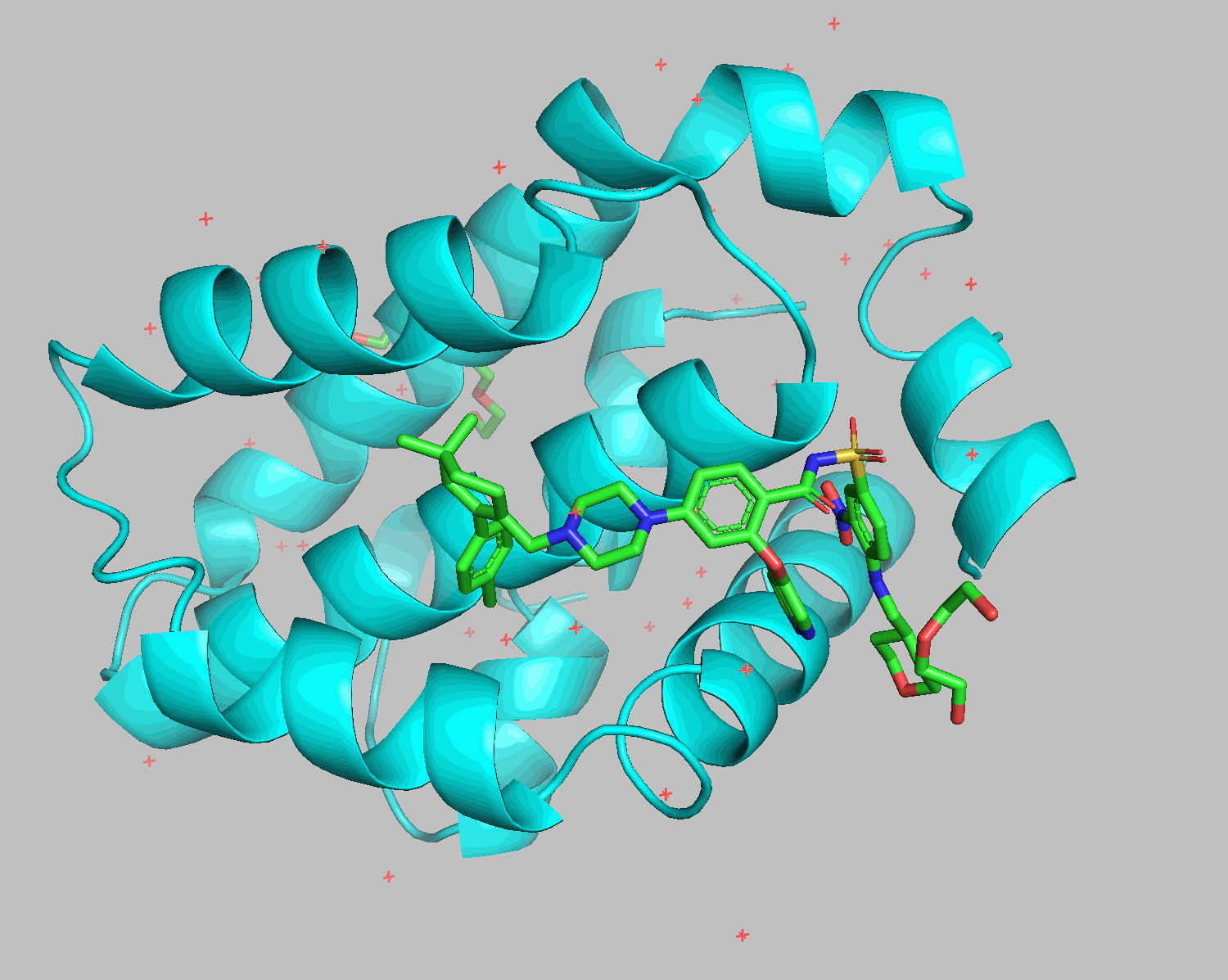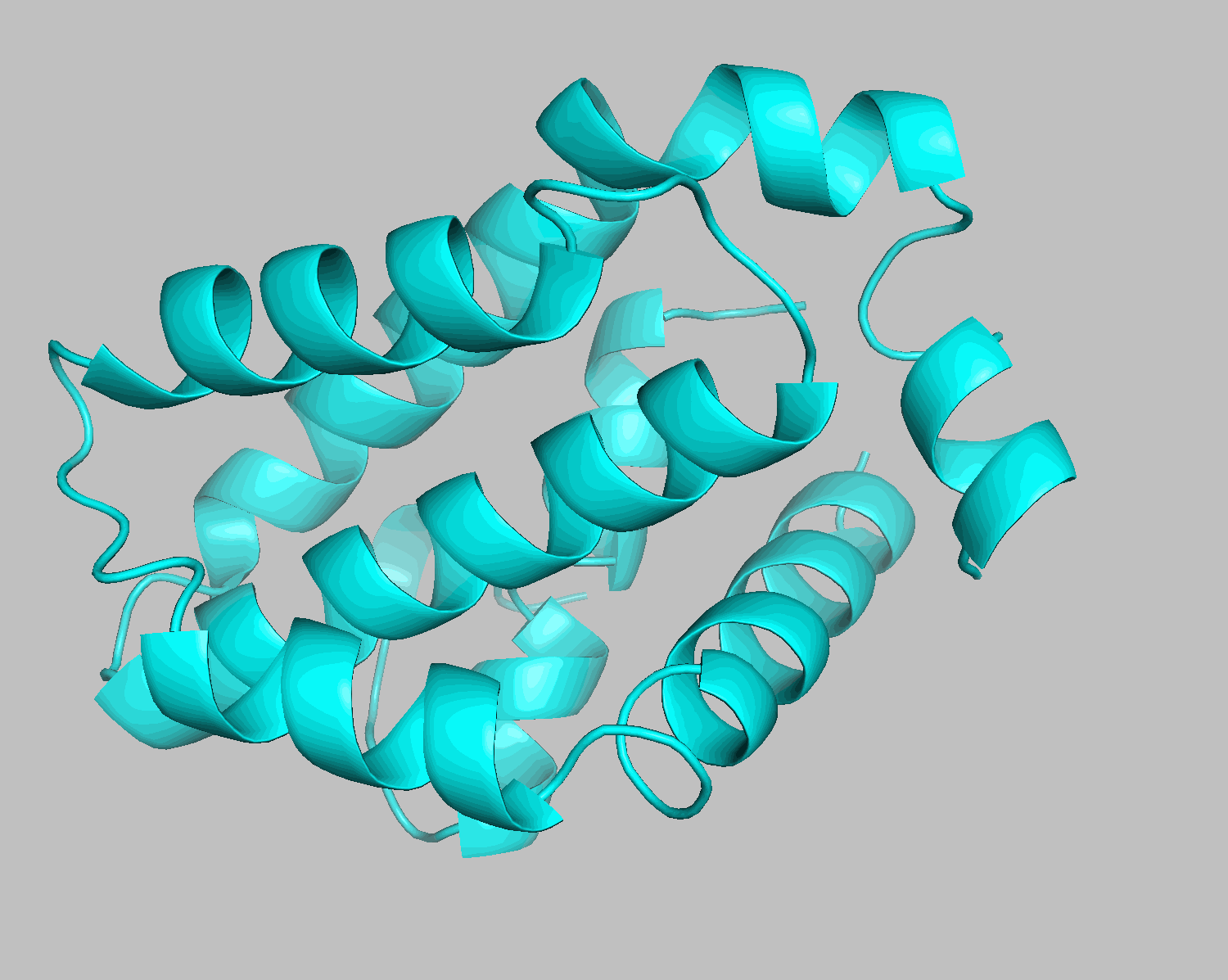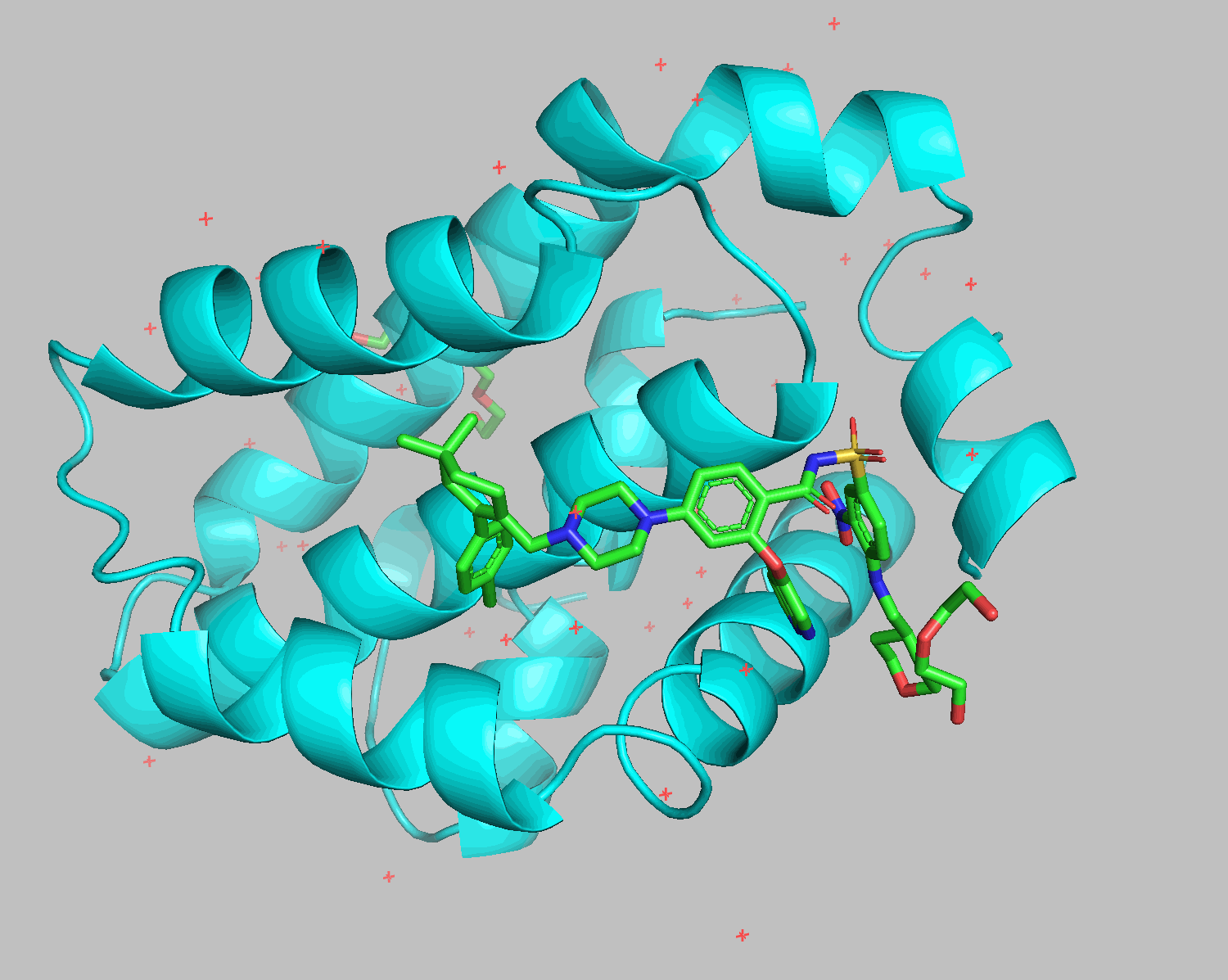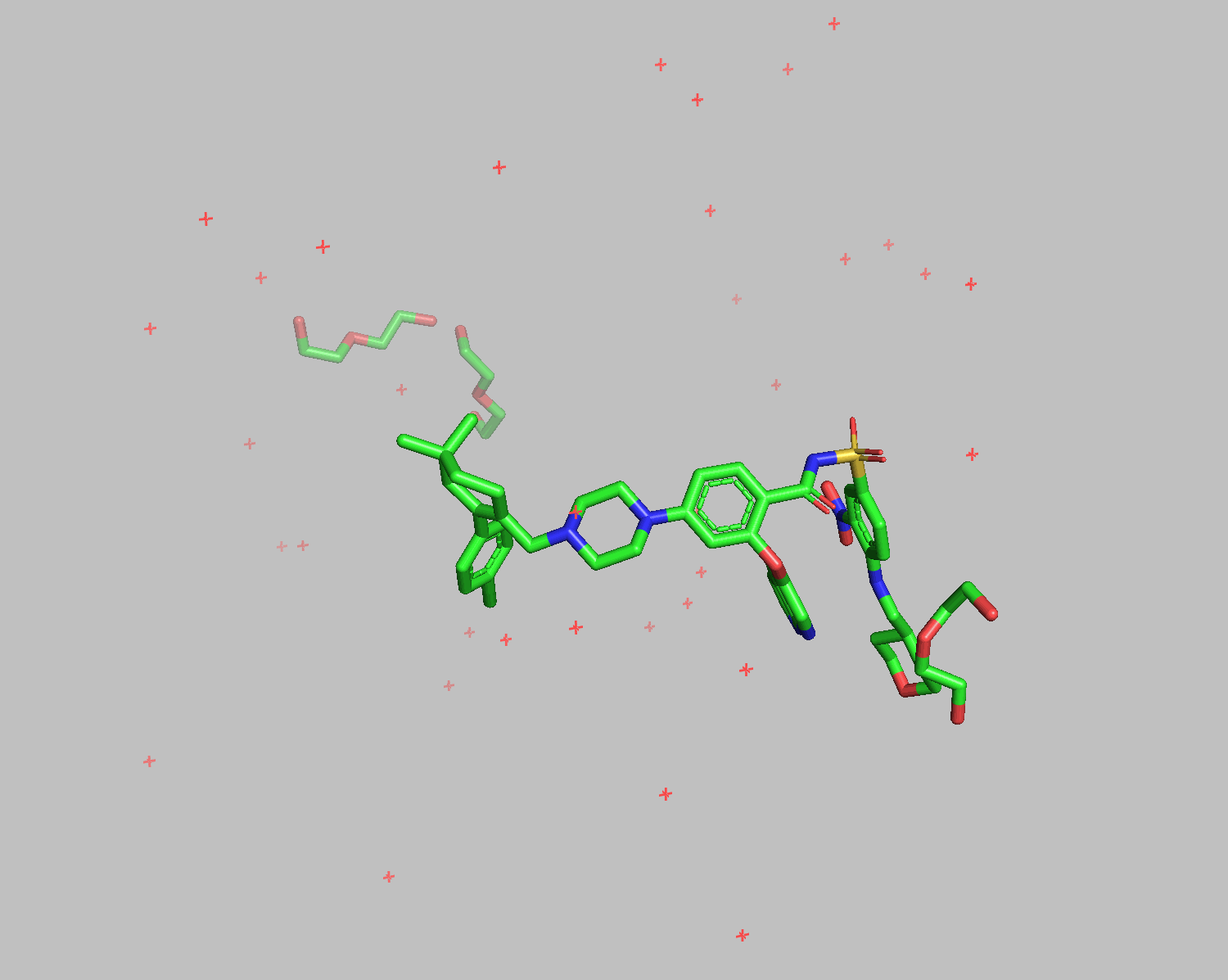
grep HETATM filenameはfilenameの中にあるHETATMを含む行を抽出するコマンドです。
6O0K.pdbの中に記載されているHETATMはリガンド以外に水や添加剤が含まれています。
sminaやgninaの便利なところの一つは、これらを排除してリガンド単体をdockingしてくれるところです。
sminaの実行
こちらのDocumentを参考にsminaを実行します。
smina -r rec.pdb -l lig.pdb --autobox_ligand lig.pdb -o docked.sdf --num_modes 1 --seed 0
実行結果をPyMOLで確認してください。PyMOLのコマンドラインから読み込めます。
load dock.sdf
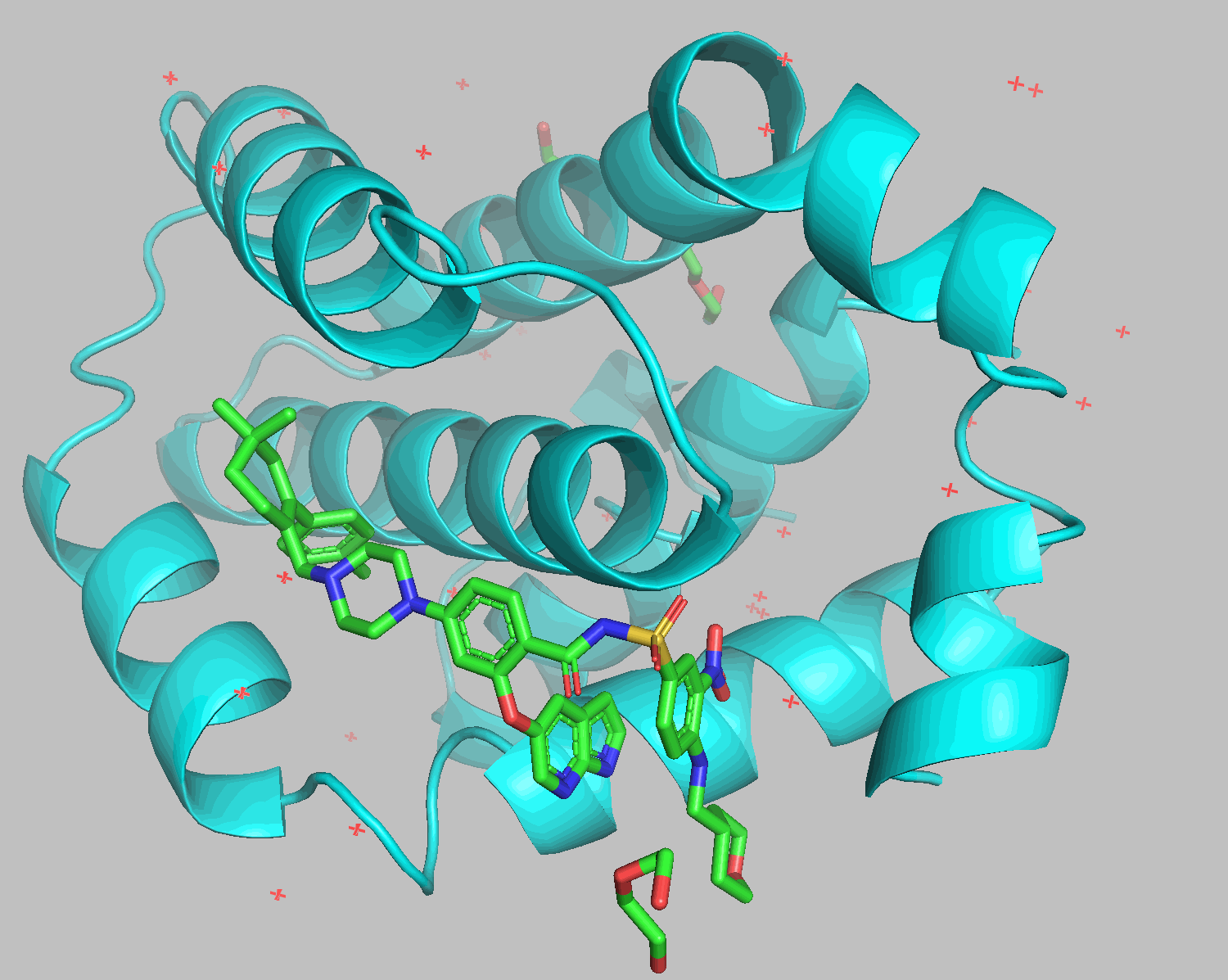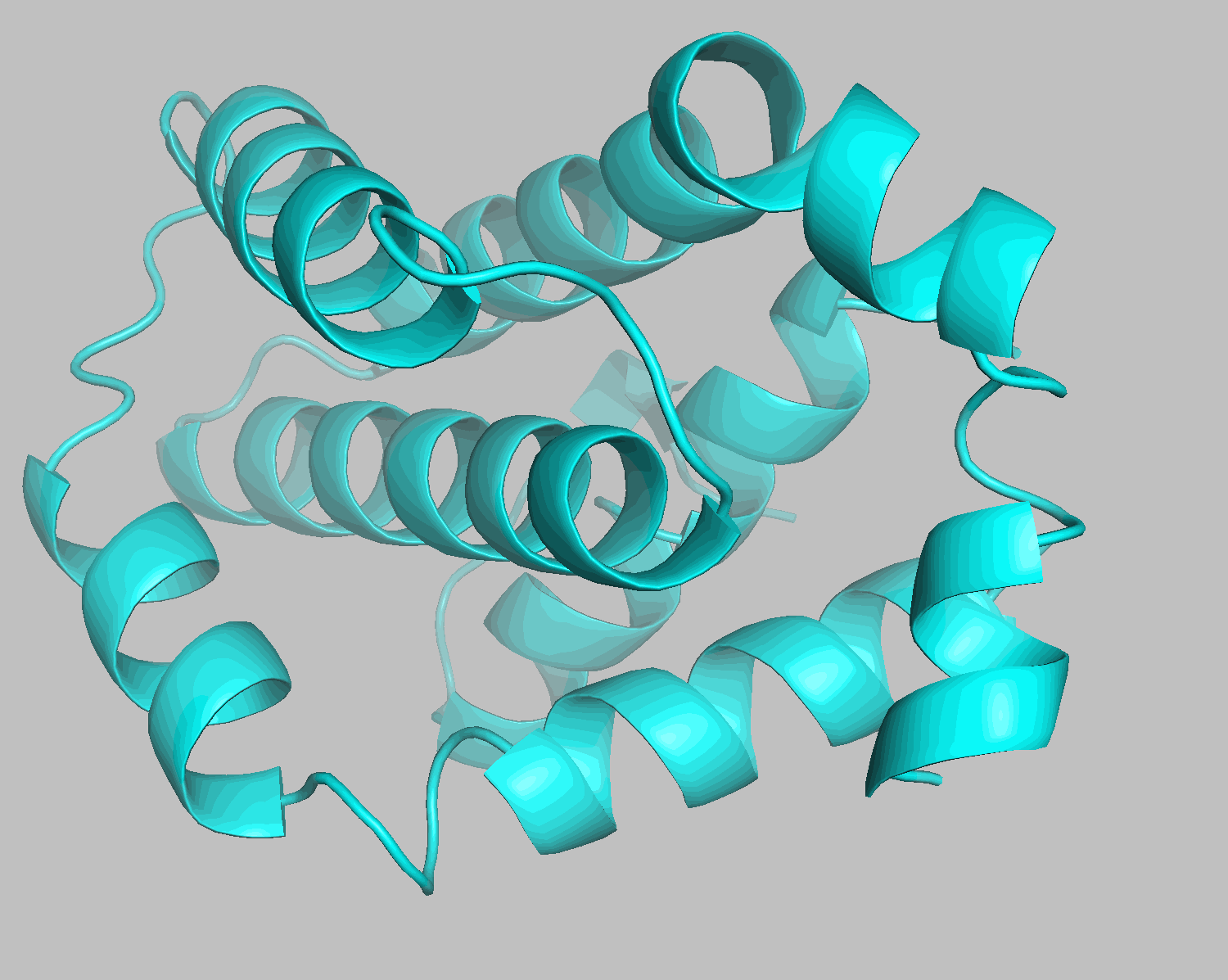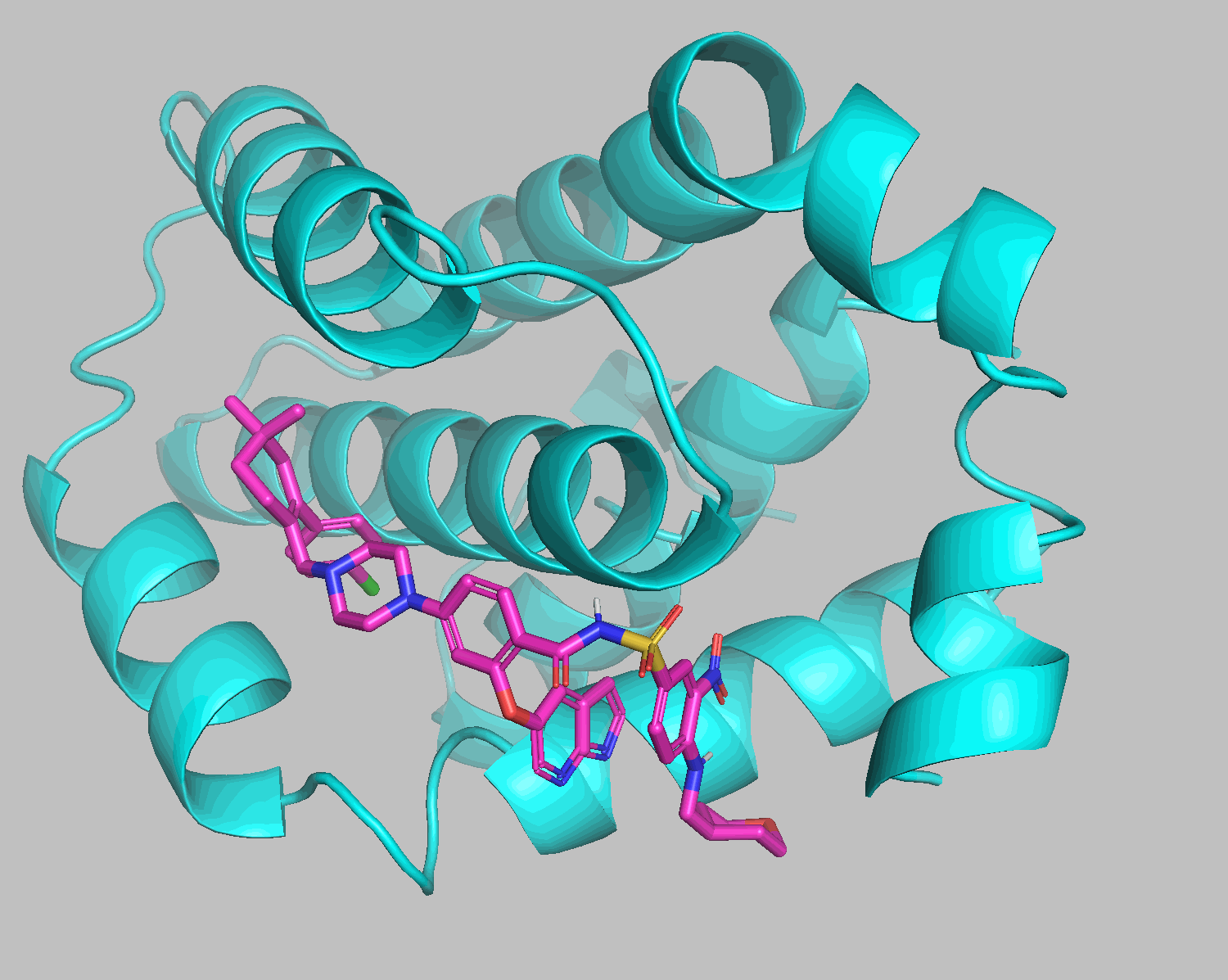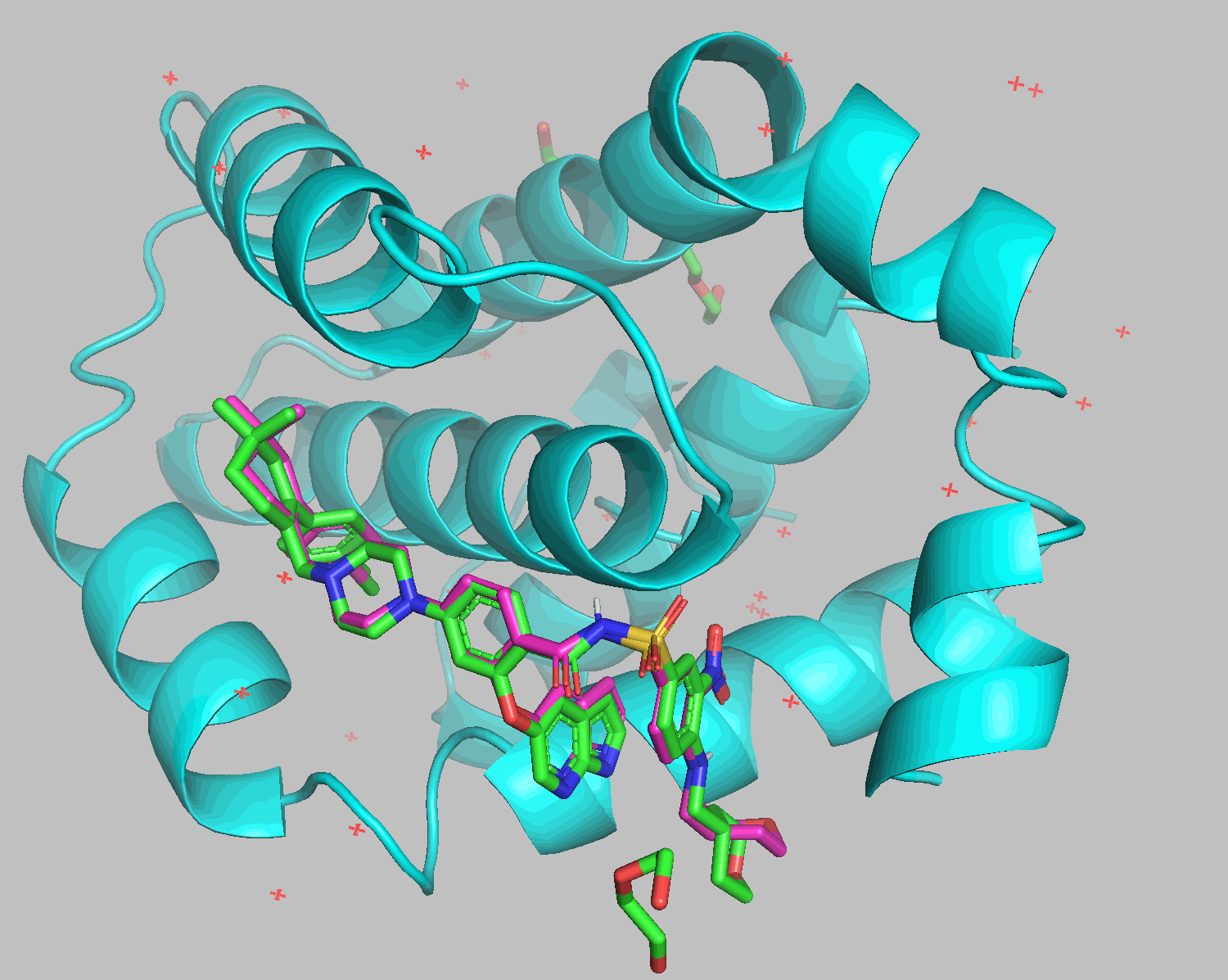
リガンドの手前にあったジエチレングリコールや水が排除された形でdockingされました。右下のTHPの部分以外はよく重なっています。
--autobox_ligandをrec.pdbでコマンドを実行すると、レセプター分子全体を探索させることができます。6O0Kではほぼ同じ結果が返ってきました。--seedオプションに同じ数値を用いることで同じ計算結果が得られるはずです。
vinaではpdbqtファイルの作成が必要ですが、sminaやgninaはpdbだけでdocking studyを実施できて非常に便利です。
Discussion