描画ソフトとしてのPyMOL
化学者にとって構造式は共通の言語と言えます。
変な話、英語が話せなくても構造式と矢印が書ければ多くを理解し合うことができます。それくらい重要なツールと言えると思います。
化学構造の定番といえばChemDrawやMarvin Sketchですが、これらは有償のツールです。大学であればフリーで使えるところも多いと思いますが(とは限らない?)、会社はもちろん、個人では気軽に使えるものではありません。
過去にはweb版とかもあったんですが、今は使えなくなったようです。
これら以外でも構造を描画したり、データとして保存してやり取りをするツールとしてOpen SourceのPyMOLを利用することも出来ます。
ここでは有名な薬剤と標的の複合体を題材に新しい阻害剤の描画を試みたいと思います。
下準備
まずはPyMOLを起動して、下記のPDBをfetchしてみましょう。
fetch 4i22
show lines, byres organic around 4
ぱっと見だけでわかる方はよく勉強されてる方だと思いますが、GefitinibとEGFR kinase domainの複合体です。
この手のSBDDの題材にはかなりよく使われる標的と薬剤です。
細かい操作は今回割愛しますが、gefitinibのキナゾリン骨格はEGFRのいわゆるヒンジ部分と相互作用しています。
窒素原子の一つがM793主鎖アミドのNHを捉えていますが、実は8位も3.2Åと相互作用できそうな距離にいますね(へぇ

「じゃあここも相互作用取りにいったらいいじゃん」と安直なmedicinal chemistなら考えてくれると思います(失礼
では早速そういった分子をデザインしてみましょう。
Builderの起動
External GUIの右側にあるBuilderをクリックすると、描画用のウィンドウが立ち上がります。使い勝手はSchrödingerのMaestroに近いです(そもそもPyMOLはSchrödinger社のソフトウェア)。

Builderの基本挙動と使い方
ChemDrawは炭素骨格を結合形成ベースで描画しながら、必要な原子や置換基で修飾していくイメージですが、PyMOLやMaestroのBuilderは原子を別の置換基や原子に置換していくイメージと言えばいいでしょうか。
前者は炭素骨格をベースに描画していくのに対して、後者は骨格伸長するために水素原子を炭素や官能基、環構造に置換します。
実際に動かしてみながらがわかりやすいと思います。
BuilderのCyclopentaneをクリックした後、右下のCreate As New Objectをクリックすると、新しいオブジェクトとしてCyclopentadieneが出力されます。

これをフランに変換したい場合は、右下のMouse Modeが3-Button Editingになっているのを確認してからメチレンをクリックし、BuilderのO(=酸素原子)をクリックします。これはChemDrawと同じ挙動だと思います。
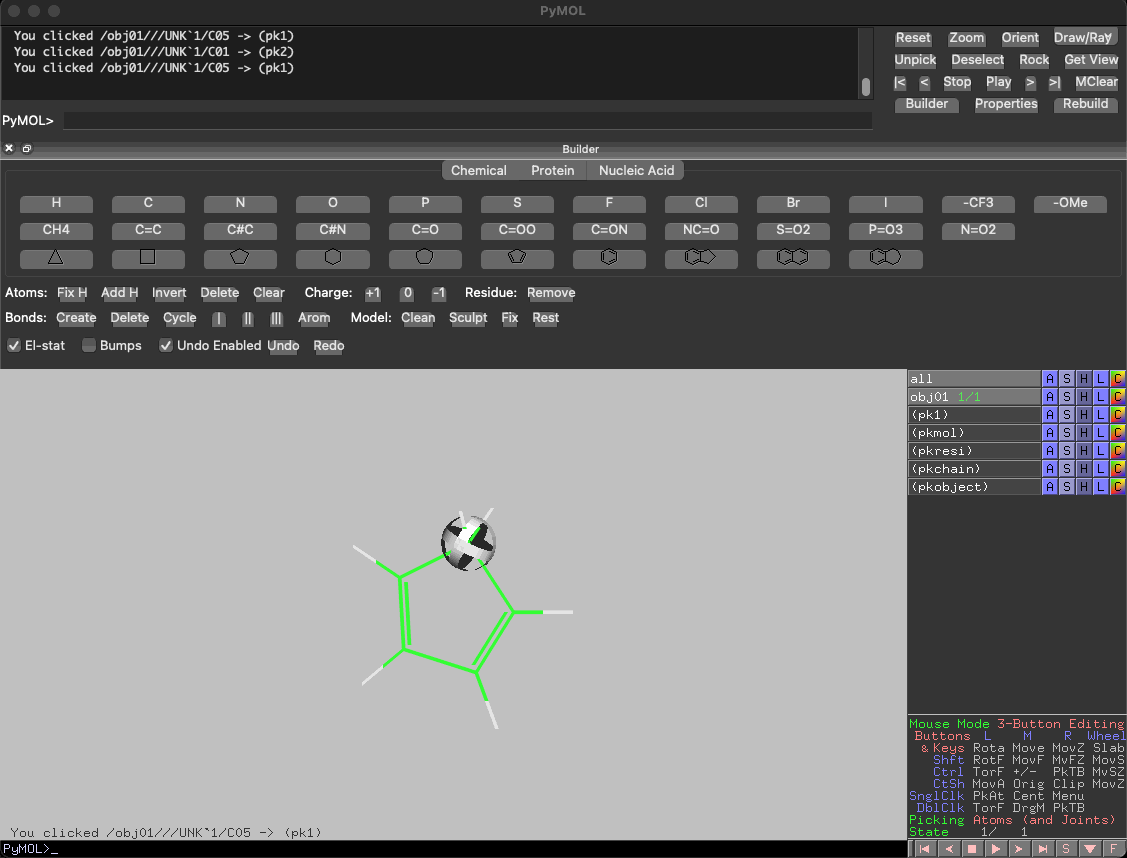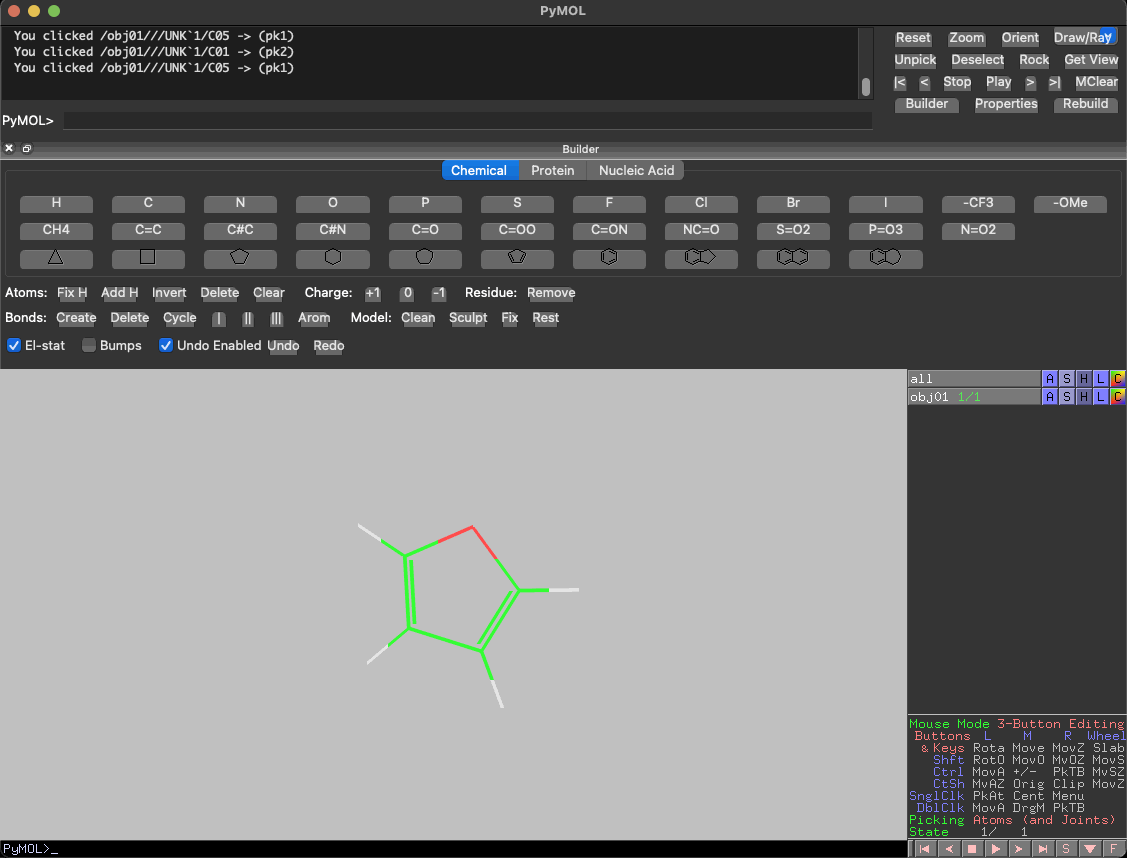
PyMOLで構造式を描いてみる
PyMOLとChemDrawの異なる点は炭素骨格の形成方法です。
ChemDrawはカーソルを結合にしてシクロペンタジエンのメチレンをクリックすると一炭素増炭されますが、PyMOLのBuilderは結合を伸ばしていくというより、ある原子を別の原子や置換基で置き換えます。
具体的には、シクロペンタジエンの増炭したい部分の水素を選択し、Builderで例えば炭素原子や他の置換基に置換します。
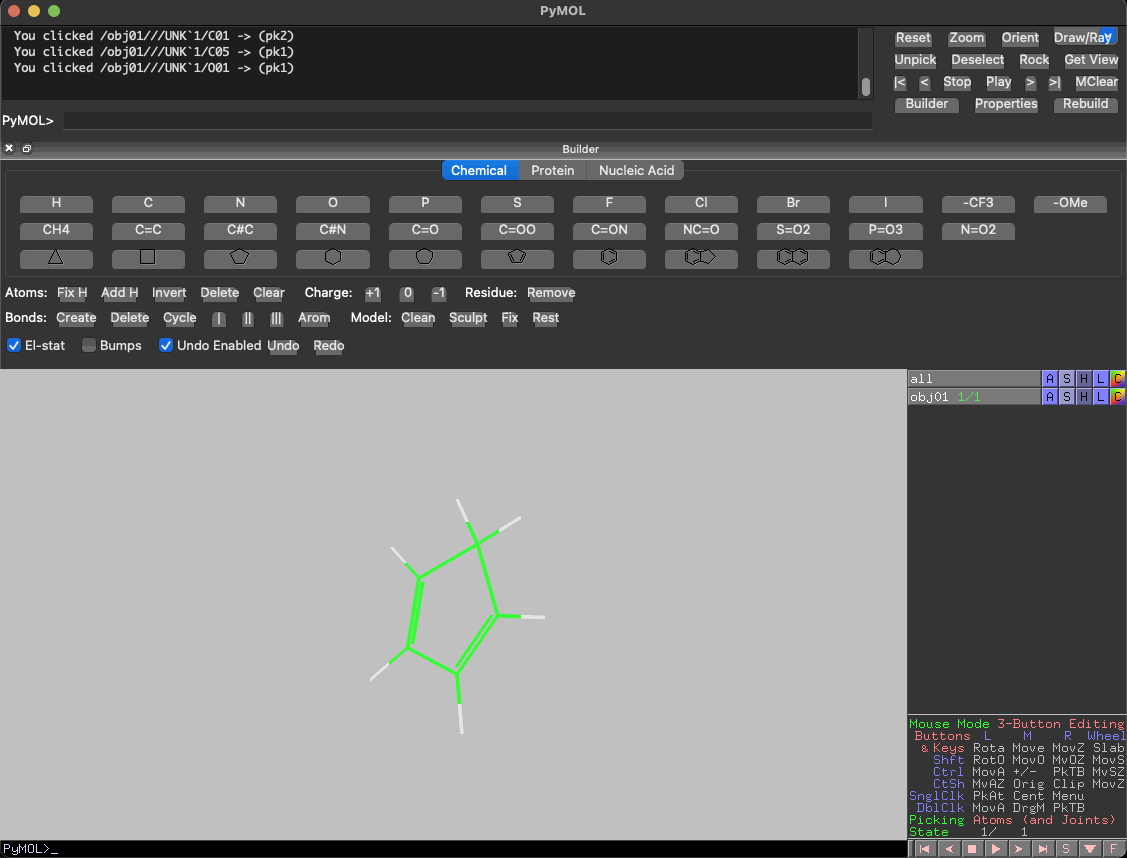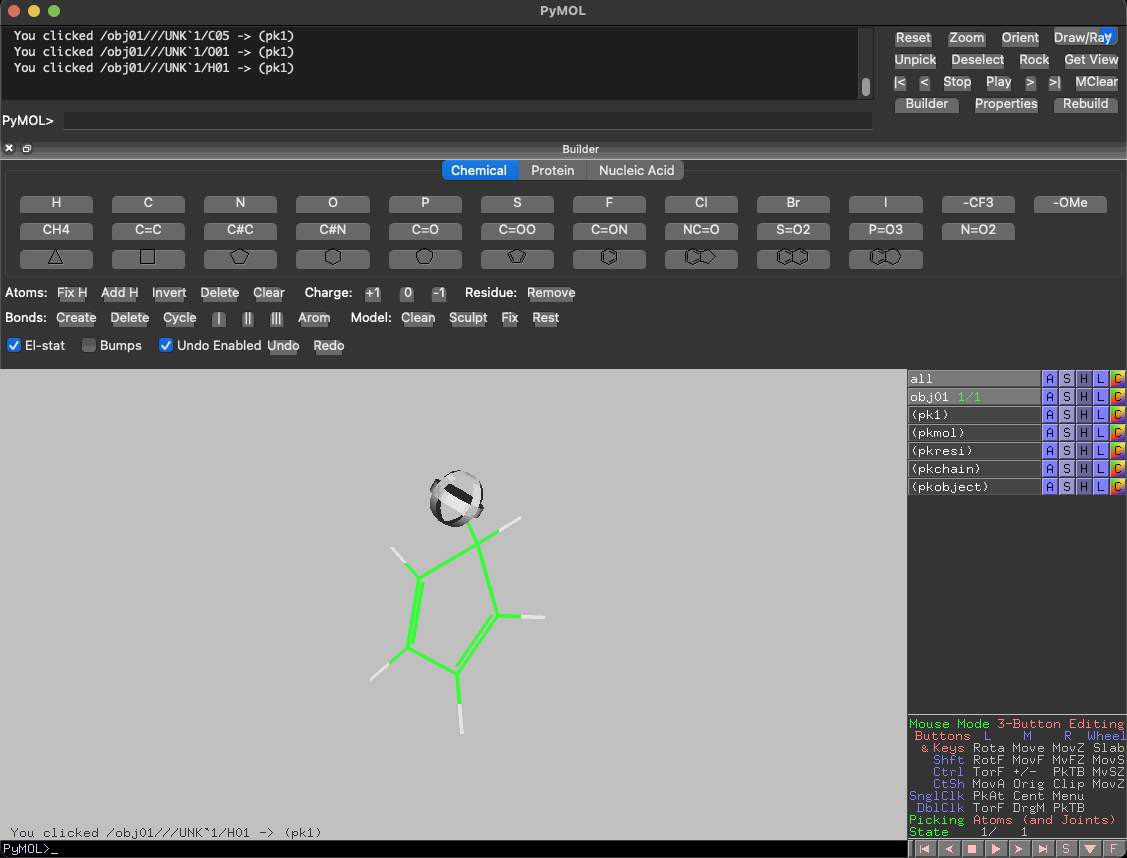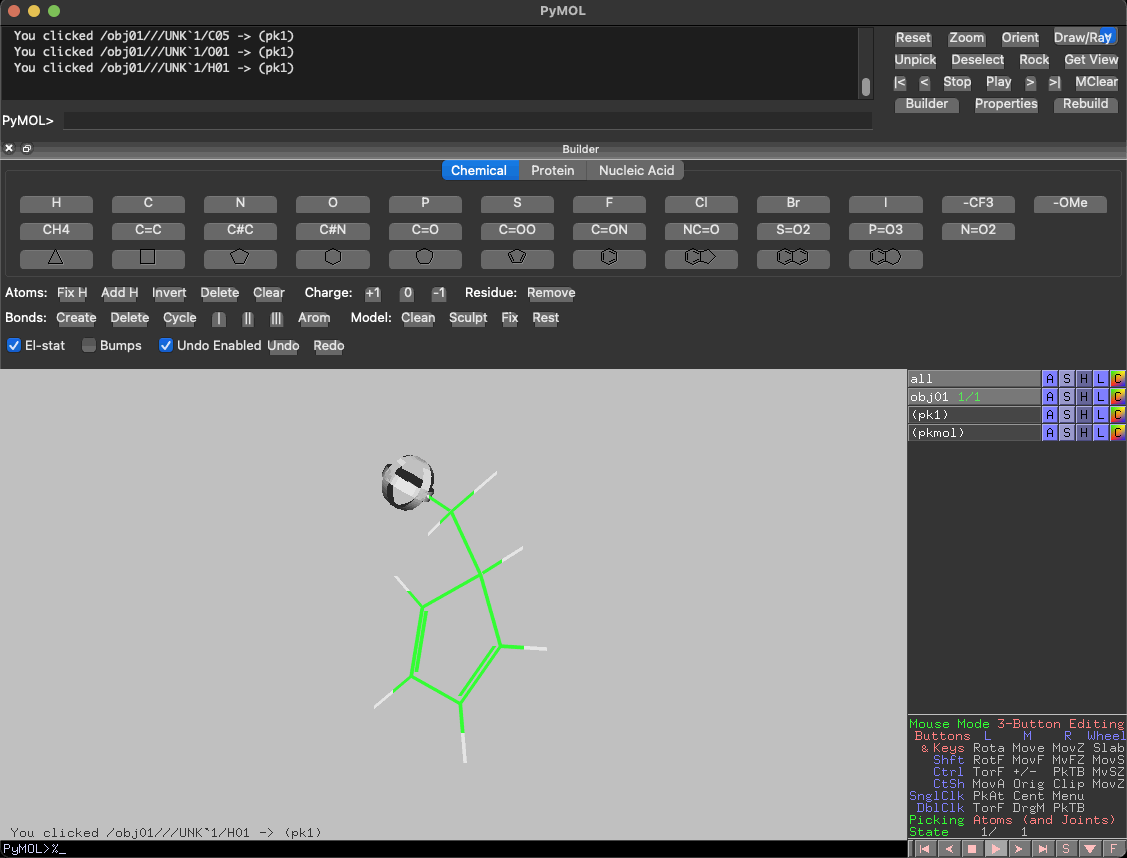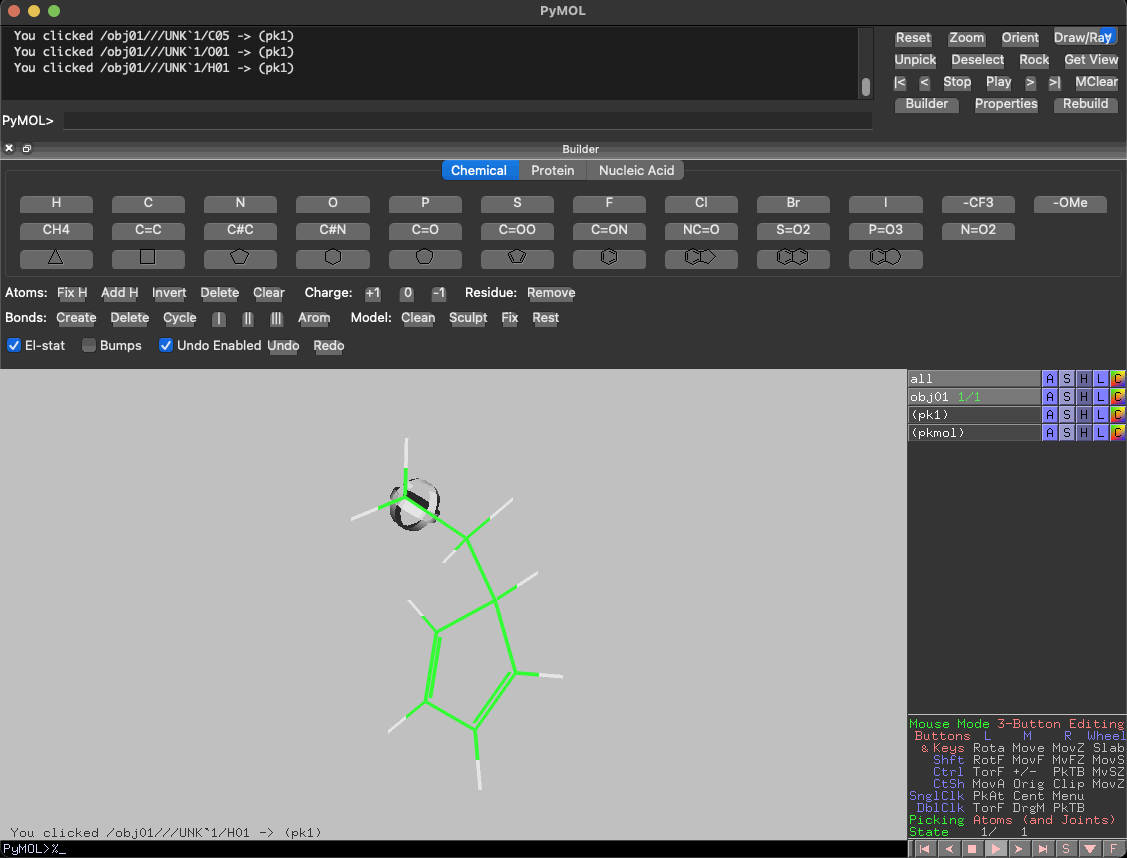
なんとなくわかってもらえたでしょうか。
原子だけでなく結合をベースに構造を描画できるのがChemDrawの特徴であるのに対し、PyMOLは原子を置換しながらでしか結合形成できません。
ChemDrawであれば結合を選択した後、環構造をツールバーから選べば簡単にインドールなどの縮環構造を描画できます。対してPyMOLでは基本的に原始しか選択できないので、水素原子を炭素原子に置換しながら最後に閉環する必要があります(多分、上手い書き方は私も知りたいです)。
先ほどのGefitinib-EGFRの構造を使ってジアザインドールの骨格に描き直してみましょう。
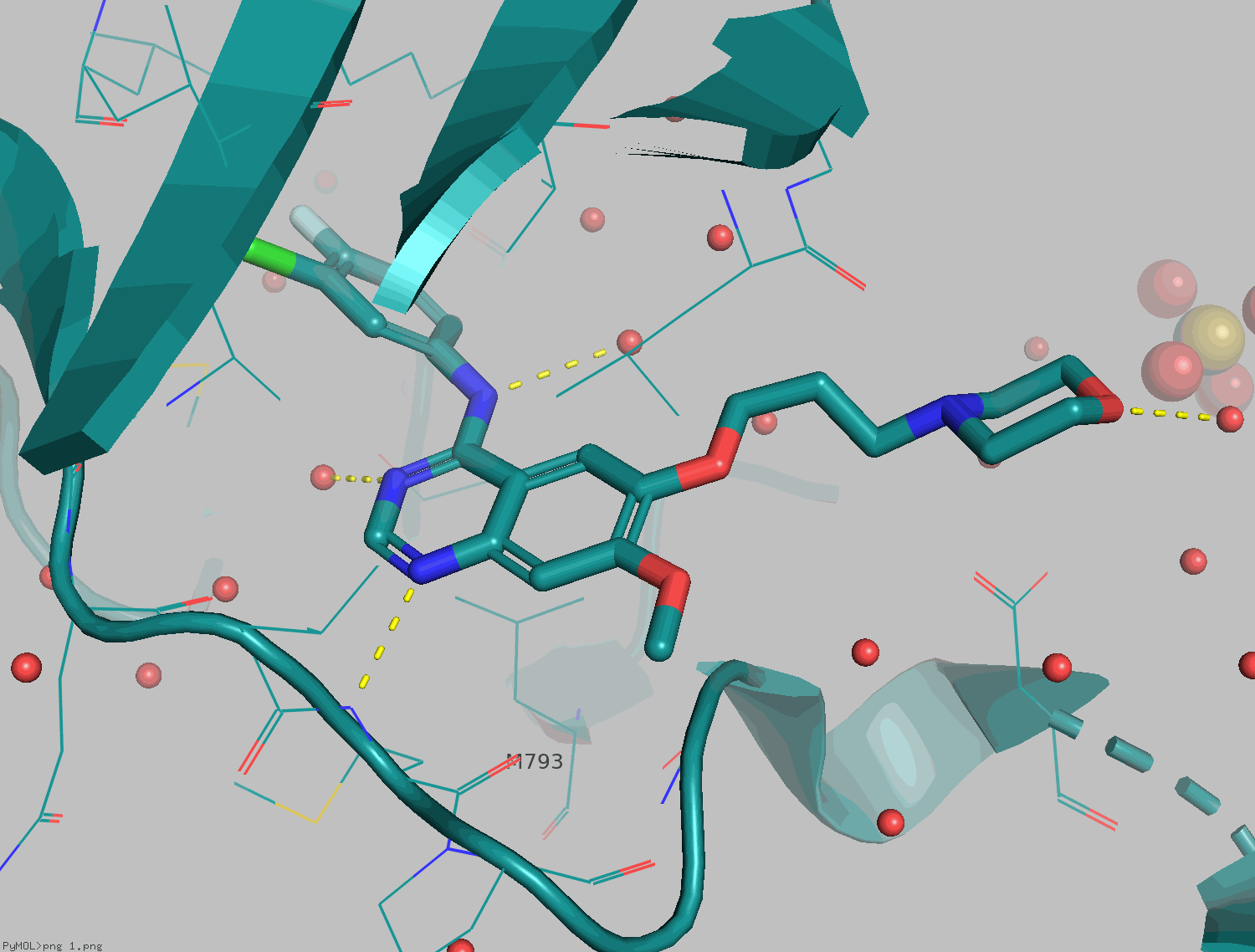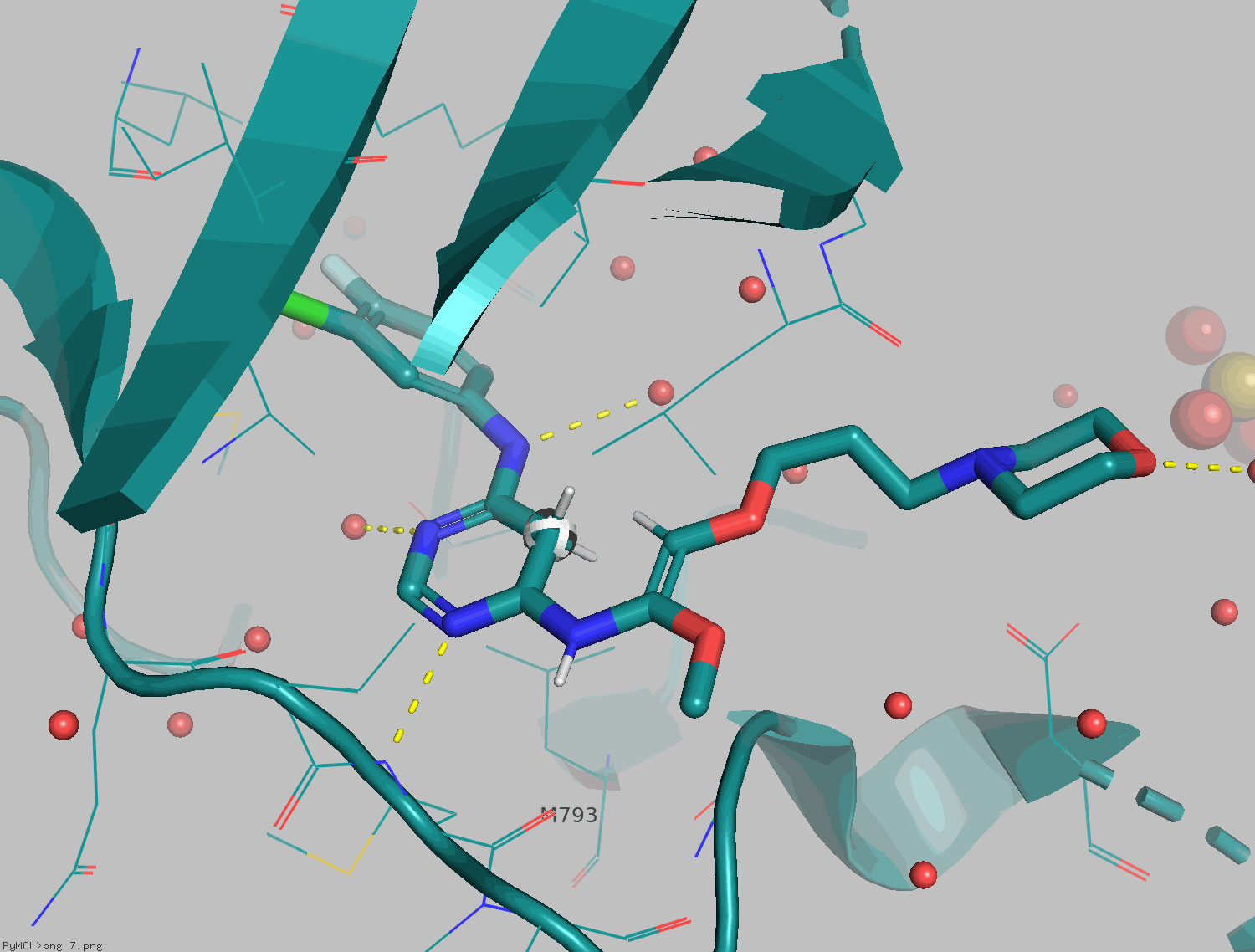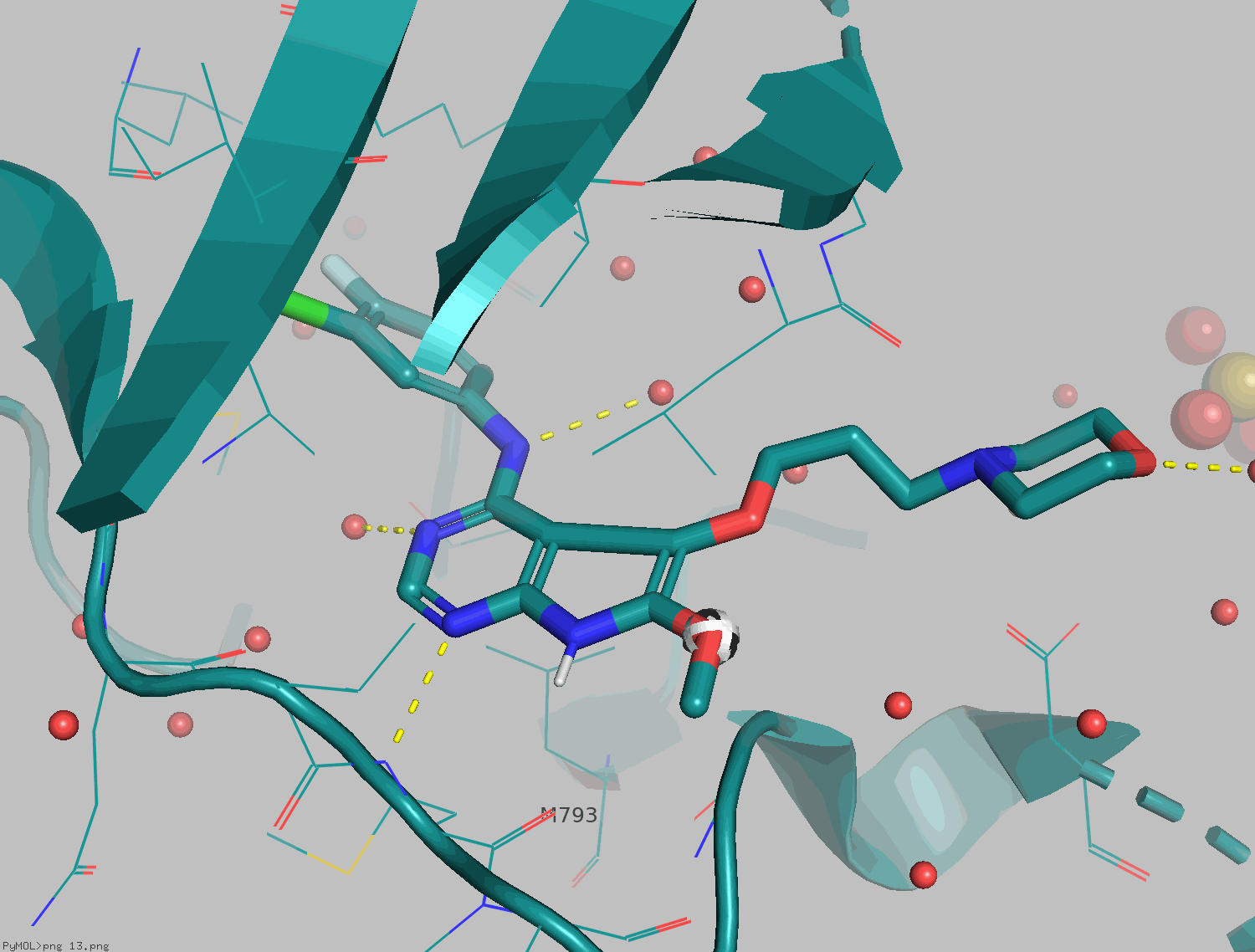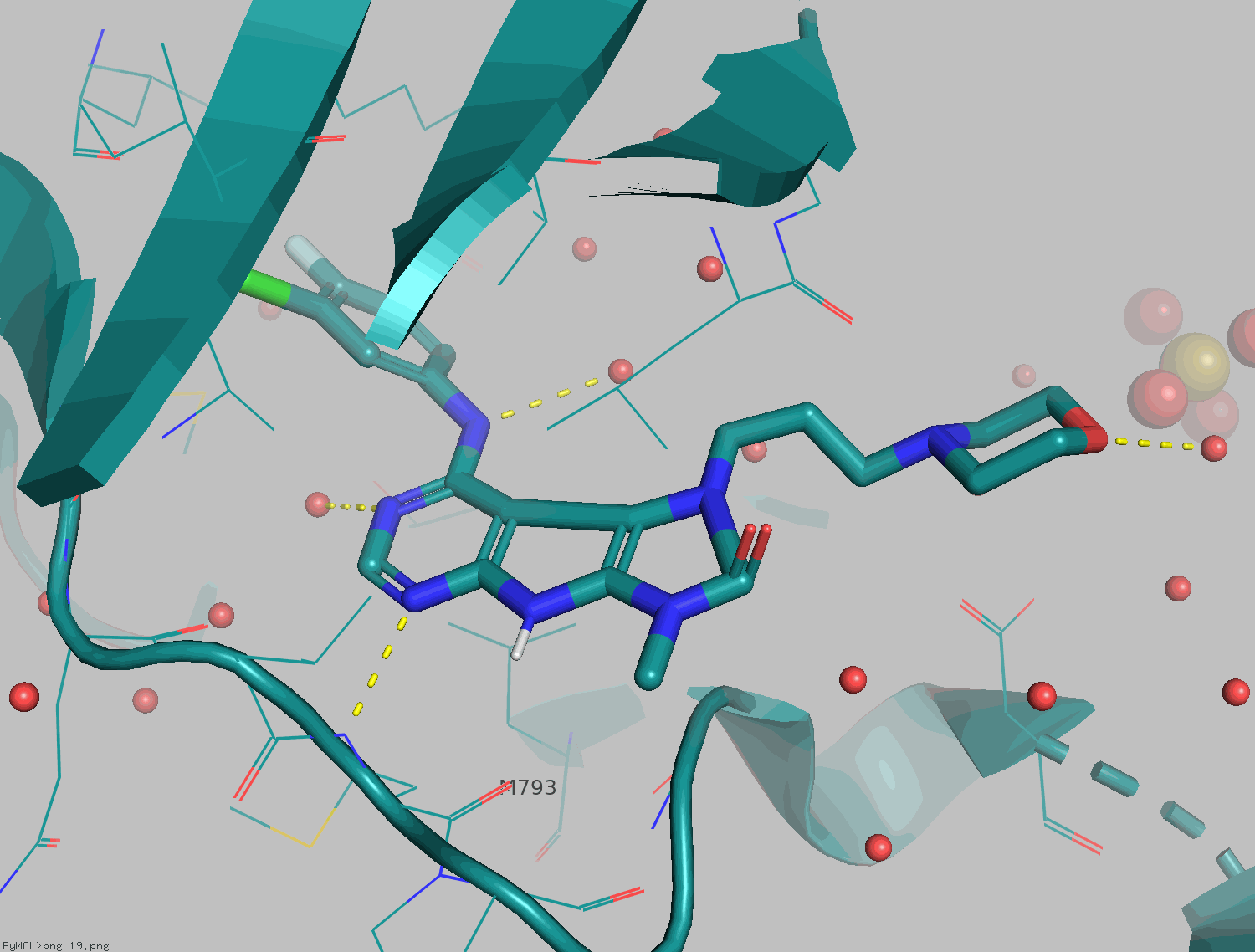
構造がめっちゃ汚いですね。ChemDrawであれば、分子を選択してctrl + shift+ Kで選択した構造を綺麗に描き直してくれる(=clean up)んですが、PyMOLやMaestroではminimizeにあたると思います。残念ながら私の環境ではminimizeがうまく動作してくれません...
こういった構造のclean upの方法の一つとして、一度SMILESに変換してから再度三次元構造を起こし直す方法があります。
というわけで、次回は描画した構造をSMILESの文字列に変換し、SDFに再変換することでPyMOL上で構造をキレイに書き直して(≒構造の正規)みたいと思います。
こちらのAg_smithさんの記事のPyMOL版です。(たまたま似たようなことをちょうどやってました)
続く。。。
Discussion