Endoscope Segmentation "Meta-Polyp" 実装と解説
Quoc-Huy Trinh
"Meta-Polyp: a baseline for efficient Polyp segmentation"
2023 IEEE 36th International Symposium on Computer-Based Medical Systems (CBMS) 742-747, IEEE Computer Society
DOI 10.1109/CBMS58004.2023.00312
https://doi.ieeecomputersociety.org/10.1109/CBMS58004.2023.00312
内視鏡画像におけるポリープ検出のモデルで、当時のSOTAモデルです。
題名通りベースラインとして使いたいのですが、TensorFlow実装だったのでPyTorchに直しがてらメモ代わりに記事にします。
画像は断りのない限り論文からの引用です。
概観
CAFormerをエンコーダとして、よくあるConv+UpSampleをデコーダにした、非常にオーソドックスなモデルで、データセット "Kvasir", "CVC-300", "CVC-ColonDB"でSOTAだった。
あまり新規性は無いが、同じデータセットを使うサーベイとして適量で読みやすかった。
この分野の実用的なサーベイはAwesome Video Polyp Segmentationがわかりやすい
モデル
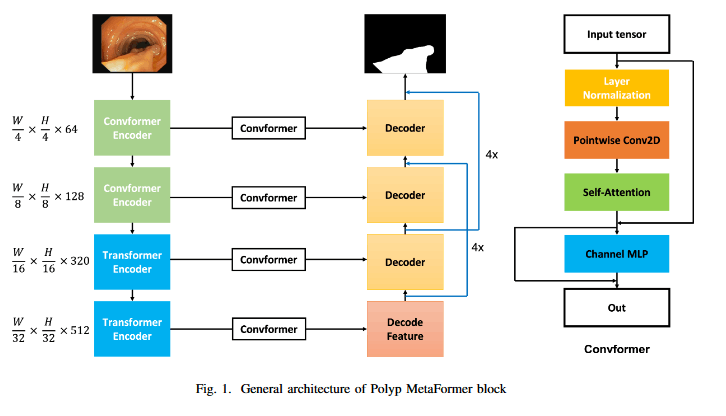
見ての通りで、CAFormer-S18をエンコーダとして、FPNをそれぞれの解像度でConvFormer(つまりMeta-FormerのTorken MixerをSepConvにしたもの)で変換し、解像度を上げながらConv1×1とConv3×3を順繰りに適用して元の解像度にする。
公式実装を見る
TensorFlow 2.11.0 で書かれているので、まずはこれを動かして計算グラフを読む。DockerでTF 2.11.0のイメージを組んで中で作業すると楽。
実際はlayerのファイルからデコーダを読めばいいだけなのだが、Kerasは書いたことも読んだことも無いので、ざっと眺めて断念した。
docker pull tensorflow/tensorflow:2.11.0
docker run --name meta_polyp -v /E/EXPERIMENT/meta_polyp:/workspace -i -t tensorflow/tensorflow:2.11.0
まずgitでリポジトリをコピーして、pythonでいろいろ使えるようにしておく。
cd /workspace
apt-get update
apt-get install -y git graphviz # graphvizは可視化でいる
git clone https://github.com/huyquoctrinh/MetaPolyp-CBMS2023.git
cd MetaPolyp-CBMS2023
pip install -r requirements.txt
pip install graphviz
これにより、実装のbuild_modelで得られるモデルの計算グラフをpngに書き出せばOK。
from tensorflow.keras.utils import plot_model
import model as m # 公式実装のファイル
model = m.build_model()
plot_model(model, show_shapes=True, expand_nested=True, to_file="model.png")
FLOPSとパラメータ数はprint(model.summary())でCLIに出力できる。以下はモデルMeta-Polypの計算グラフのうち、CAFormerからの出力部分、つまりデコーダをグラフにした画像である。
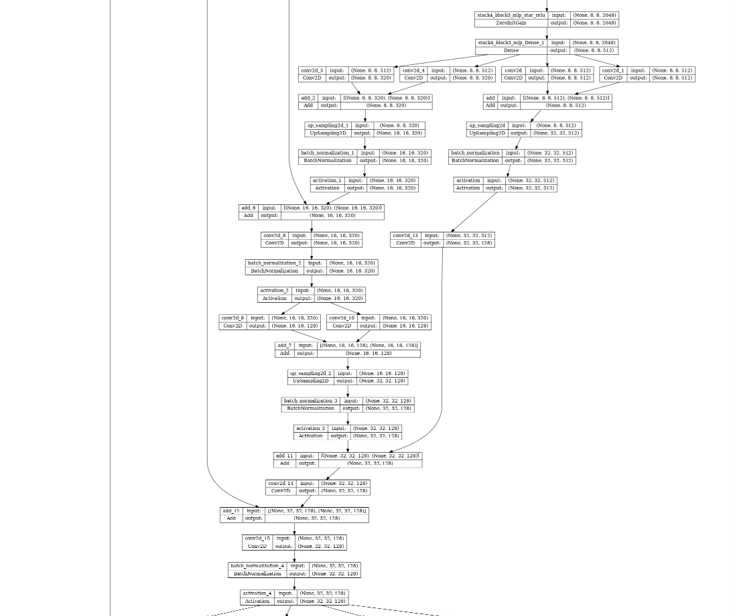

左の線はCAFormerからの中間出力で、これと逆方向への特徴量の結合、そして2つのショートカットコネクションで構成されていることがわかった。
再構築
PyTrorchではCAFormerはtimmに収録されているので、これを使う。
Yu, Weihao and Si, Chenyang and Zhou, Pan and Luo, Mi and Zhou, Yichen and Feng, Jiashi and Yan, Shuicheng and Wang, Xinchao
"MetaFormer Baselines for Vision"
2024, IEEE Transactions on Pattern Analysis and Machine Intelligence vol.46-2 896-912
DOI: 10.1109/TPAMI.2023.3329173
https://arxiv.org/abs/2210.13452
class MetapolypEncoder(nn.Module):
def __init__(self):
super().__init__()
model = timm.create_model(
"caformer_s18.sail_in22k_ft_in1k",
pretrained=True,
)
self.stem = model.stem
self.block0 = model.stages[0]
self.block1 = model.stages[1]
self.block2 = model.stages[2]
self.block3 = model.stages[3]
self.mlp0 = MetaFormerStage(64, 64, depth=1, mlp_act=nn.GELU, token_mixer=SepConv)
self.mlp1 = MetaFormerStage(128, 128, depth=1, mlp_act=nn.GELU, token_mixer=SepConv)
self.mlp2 = MetaFormerStage(320, 320, depth=1, mlp_act=nn.GELU, token_mixer=SepConv)
self.mlp3 = MetaFormerStage(512, 512, depth=1, mlp_act=nn.GELU, token_mixer=SepConv)
return None
def forward(self, x):
# x input [B,3,H,W]
x = self.stem(x)
x = self.block0(x) # output [B, 64, H/2, W/2]
x0 = self.mlp0(x)
x = self.block1(x) # output [B, 128, H/4, W/4]
x1 = self.mlp1(x)
x = self.block2(x) # output [B, 320, H/8, W/8]
x2 = self.mlp2(x)
x = self.block3(x) # output [B, 512,H/16,W/16]
x3 = self.mlp3(x)
return x0, x1, x2, x3
説明するほどでもないが、CAFormerの中間出力を引っ張ってきて、ConvFormerで処理している。出力される特徴マップはコメントの通り。
肝心のデコーダは以下のようになっている。実際はモジュールを分けて書くべき(公式はプログラム中央のfor内で組んでいる)だが、今回のモデルはスケーリングなども無いため直書きした。
class MetapolypDecoder(nn.Module):
def __init__(self, num_class):
super().__init__()
self.upscale2 = nn.Upsample(scale_factor=2, mode="bicubic")
self.upscale4 = nn.Upsample(scale_factor=4, mode="bicubic")
# 1/36スケール出力を受け取り、1/16と1/8(shortcut)スケールで返す
self.conv3_3 = nn.Sequential(
nn.Conv2d(512, 320, 3, padding=1, bias=False),
nn.GELU()
)
self.conv3_1 = nn.Sequential(
nn.Conv2d(512, 320, 1, bias=False),
nn.GELU()
)
self.ba3 = nn.Sequential(
nn.BatchNorm2d(320),
nn.ReLU(),
)
self.conv3_3s = nn.Sequential(
nn.Conv2d(512, 512, 3, padding=1, bias=False),
nn.GELU()
)
self.conv3_1s = nn.Sequential(
nn.Conv2d(512, 512, 1, bias=False),
nn.GELU()
)
self.ba3s = nn.Sequential(
nn.BatchNorm2d(512),
nn.ReLU(),
nn.Conv2d(512, 128, 3, padding=1, bias=False)
)
# 1/16スケール出力を受け取り、1/8スケールで返す
self.cba2 = nn.Sequential(
nn.Conv2d(320, 320, 3, padding=1, bias=False),
nn.BatchNorm2d(320),
nn.GELU()
)
self.conv2_3 = nn.Sequential(
nn.Conv2d(320, 128, 3, padding=1, bias=False),
nn.GELU()
)
self.conv2_1 = nn.Sequential(
nn.Conv2d(320, 128, 1, bias=False),
nn.GELU()
)
self.ba2 = nn.Sequential(
nn.BatchNorm2d(128),
nn.ReLU(),
)
# 1/8スケール出力と同じくshortcutを受け取り、1/4と1/2(shortcut)スケールで返す
self.c1 = nn.Conv2d(128, 128, 3, padding=1, bias=False)
self.cba1 = nn.Sequential(
nn.Conv2d(128, 128, 3, padding=1, bias=False),
nn.BatchNorm2d(128),
nn.GELU()
)
self.conv1_3 = nn.Sequential(
nn.Conv2d(128, 64, 3, padding=1, bias=False),
nn.GELU()
)
self.conv1_1 = nn.Sequential(
nn.Conv2d(128, 64, 1, bias=False),
nn.GELU()
)
self.ba1 = nn.Sequential(
nn.BatchNorm2d(64),
nn.ReLU(),
)
self.conv1_3s = nn.Sequential(
nn.Conv2d(128, 128, 3, padding=1, bias=False),
nn.GELU()
)
self.conv1_1s = nn.Sequential(
nn.Conv2d(128, 128, 1, bias=False),
nn.GELU()
)
self.ba1s = nn.Sequential(
nn.BatchNorm2d(128),
nn.ReLU(),
nn.Conv2d(128, 32, 3, padding=1, bias=False)
)
# 1/4スケール出力を受け取り、1/2スケールで返す
self.cba0 = nn.Sequential(
nn.Conv2d(64, 64, 3, padding=1, bias=False),
nn.BatchNorm2d(64),
nn.GELU()
)
self.conv0_3 = nn.Sequential(
nn.Conv2d(64, 32, 3, padding=1, bias=False),
nn.GELU()
)
self.conv0_1 = nn.Sequential(
nn.Conv2d(64, 32, 1, bias=False),
nn.GELU()
)
self.ba0 = nn.Sequential(
nn.BatchNorm2d(32),
nn.ReLU(),
)
# 1/2スケール出力と同じくshortcutを受け取り、1/2スケールで返す
# ここではsigmoidは通さない
self.head = nn.Sequential(
nn.Conv2d(32, 32, 3, padding=1, bias=False),
nn.BatchNorm2d(32),
nn.ReLU(),
nn.Conv2d(32, num_class, 1, bias=False),
)
return None
def forward(self, x0, x1, x2, x3):
o3s = self.ba3s(self.upscale4(self.conv3_1s(x3) + self.conv3_3s(x3)))
o3 = self.ba3(self.upscale2(self.conv3_3(x3) + self.conv3_1(x3)))
o2 = self.cba2(x2 + o3)
o2 = self.ba2(self.upscale2(self.conv2_3(o2) + self.conv2_1(o2)))
o1 = self.c1(o2 + o3s) + x1
o1s = self.ba1s(self.upscale4(self.conv1_1s(o1) + self.conv1_3s(o1)))
o1 = self.ba1(self.upscale2(self.conv1_3(o1) + self.conv1_1(o1)))
o0 = self.cba0(x0 + o1)
o0 = self.ba0(self.upscale2(self.conv0_3(o0) + self.conv0_1(o0)))
out = self.head(o0 + o1s)
return out
これを繋げれば完成。
class MetapolypModel(nn.Module):
def __init__(self, num_class):
super().__init__()
self.encoder = MetapolypEncoder()
self.decoder = MetapolypDecoder(num_class)
self.sigmoid = nn.Sigmoid()
self.upsample_x2 = nn.Upsample(scale_factor=2, mode='bicubic')
return None
def forward(self, x):
x0, x1, x2, x3 = self.encoder(x)
out = self.decoder(x0, x1, x2, x3)
out = self.sigmoid(out)
out = self.upsample_x2(out)
return out
このMetapolypModelを使うことでPyTrochでもモデルMeta-Polypを動かして検証できる。
論文中にはなかったが、Keras測定で公式実装のパラメータ数は28.7M、FLOPSは13.9G。
PyTorch実装ではパラメータ数34.8M、FLOPSは5.23G。入力も256×256で同じはずなのだが、何故か結果が全く違う。Kerasのカウント方法がわからないので真相は謎。ヘルプ求む。
学習
コメントアウトされた部分も含めて2パターンの戦略が書いてある。論文にはIoULoss(JaccardLoss)を使っていると書いてあったがなぜかDiceLossが使われていた。応用ML論文あるある。
- 画像 256×256
- バッチサイズ8
- 350 epoch
- DiceLoss
- 使われていない方(メモ?)
- AdamW(lr=1e-3, weight_decay=cosine_annealing_with_warmup ←?)
- SGD(lr=1e-4)
- 使われている方
- fn = PolynomialDecay(start_lr=1e-4, end_lr=1e-6, power=0.2, decay_steps=1000)
- AdamW(lr=1e-4, weight_decay=fn ←?)
WeghtDecayをスケジュールするのは珍しい... というか、KerasではWeightDecayにスケジューラを入れると学習率が変化するのかもしれない。それはそれで謎だが。
画像のオーグメンテーションは有名所を使っていると記述されているが、内視鏡セマンティックセグメンテーションでCutOutなどを使うのは悪手(マスクが変形されてテストデータの分布と差が大きくなる)だと考えられるが、大丈夫だったのだろうか。
Lightningによる実装
データセットを複数個纏めて整形したものを持っているので、今回はこれを10分割して性能評価してみる。計算リソースの関係で少し設定は異なる。Lightningを使った以下の実装を動かす。
import glob
import multiprocessing
from sklearn.model_selection import train_test_split
import torch
from torchvision.transforms import v2 as transforms
import lightning as pl
from model_metapolyp import MetapolypModel
from my_segutil import ImageLoader, MyResizeFlipCrop
from my_lossmetrics import IoUMetrics, DiceLoss
MAXEPOCH = 32
#---------------------------------------------------------------------------
# dataset
class ColonoscopeDataset(torch.utils.data.Dataset):
def __init__(self, images, masks):
self.images = images
self.masks = masks
self.loader = ImageLoader(images, masks)
self.transforms_share = MyResizeFlipCrop(size=224, clopscale=0.9, normrize_img=False)
self.transforms_image = transforms.Compose([
transforms.RandomGrayscale(p=0.1),
transforms.Normalize([0.485, 0.456, 0.406], [0.229, 0.224, 0.225]),
])
return None
def __len__(self):
return len(self.images)
def __getitem__(self, idx):
img, msk = self.loader(idx)
img, msk = self.transforms_share(img, msk)
img = self.transforms_image(img)
msk = transforms.functional.rgb_to_grayscale(msk)
msk = torch.where(msk > 0., 1., 0.)
return img.to(torch.float), msk.to(torch.float)
class ColonoscopeDataModule(pl.LightningDataModule):
def __init__(self, batch_size=32, num_workers=4):
super().__init__()
self.images = "dataset_segmentation/images/*.jpg"
self.masks = "dataset_segmentation/masks/*.png"
self.batch_size = batch_size
self.num_workers = num_workers
return None
def prepare_data(self):
return None
def setup(self, stage):
dataset_images_list = glob.glob(self.images)
dataset_images_list.sort()
dataset_masks_list = glob.glob(self.masks)
dataset_masks_list.sort()
x_train, x_test, y_train, y_test = train_test_split(
dataset_images_list,
dataset_masks_list,
shuffle=True,
random_state=1,
test_size=1/NUM_FOLD
)
self.train_dataset = ColonoscopeDataset(x_train, y_train)
self.test_dataset = ColonoscopeDataset(x_test, y_test)
return None
def train_dataloader(self):
train_dataloader = torch.utils.data.DataLoader(
self.train_dataset,
batch_size=self.batch_size,
shuffle=True,
num_workers=self.num_workers,
)
return train_dataloader
def val_dataloader(self):
test_dataloader = torch.utils.data.DataLoader(
self.test_dataset,
batch_size=self.batch_size,
shuffle=True,
num_workers=self.num_workers,
)
return test_dataloader
#---------------------------------------------------------------------------
# model
class MetapolypModelModule(pl.LightningModule):
def __init__(self):
super().__init__()
self.model = MetapolypModel(num_class=1)
self.loss = DiceLoss() # FocalIoULoss()
self.metrics = IoUMetrics()
return None
def forward(self, x):
y = self.model(x)
return y
def training_step(self, batch, batch_idx):
img, msk = batch
out = self.forward(img)
loss = self.loss(out, msk)
self.log("train_loss", loss, logger=True, prog_bar=True)
return {"loss": loss}
def validation_step(self, batch, batch_idx):
img, msk = batch
out = self.forward(img)
m = self.metrics(out, msk)
self.log("IoU", m, logger=True, on_epoch=True, prog_bar=True)
return {"metrics": m}
def configure_optimizers(self):
optimizer = torch.optim.AdamW(
self.parameters(),
lr=1e-4,
weight_decay=0.01)
# lr_scheduler = torch.optim.lr_scheduler.PolynomialLR(
# optimizer,
# total_iters=MAXEPOCH,
# power=0.2
# )
lr_scheduler = torch.optim.lr_scheduler.CosineAnnealingLR(
optimizer,
MAXEPOCH,
eta_min=0.00001)
return {"optimizer": optimizer, "lr_scheduler": lr_scheduler}
#---------------------------------------------------------------------------
# main
def main():
datamodule = ColonoscopeDataModule(batch_size=64)
model = MetapolypModelModule()
pllogger_csv = pl.pytorch.loggers.CSVLogger("./train_logs/", name="meta_polyp")
trainer = pl.Trainer(
logger=pllogger_csv
enable_checkpointing=True,
check_val_every_n_epoch=1,
accelerator="gpu",
devices=1,
max_epochs=MAXEPOCH,
)
trainer.fit(
model,
datamodule=datamodule
)
return None
if __name__ == "__main__":
multiprocessing.freeze_support()
main()
結果としてはIoU = 0.73ほどとなった。
参考として、U-Netと自作の軽量SegNeXtでも学習させたが、IoUはそれぞれ0.77と0.87で、CAFormerを使う今回の手法はパラメータ数6倍でTrain Timeも圧倒的に長い割にはちょっと微妙な結果になった。
FocalIoULossを使うと何故かCUDAが落ちてしまうので、論文実装通りDiceLossを使っているが、他2つはFocalIoULossを使っている。この結果をみると、FocalLossによる効果の方がモデル本体の性能より良く出ている可能性も考えられてしまう。CUDAのバグを改善できたらもう一度評価してみたい。他の原因としては、モデルの規模が大きい&Transformer系のAttntionを持っているために、データへの暴露回数(つまりepoch数)が足りないという原因も考えられる。Swinv2などを扱っていたときの経験からしても、Transfomer系のモデルはResNetなどのCNNと異なり、畳み込みという帰納バイアスがないためデータの学習が多く必要となる。公式実装では350 epochも回していたので、今回のデータセットの量がその3倍だと仮定すると、適切なepoch数は100 epochくらいなのかもしれない。その場合今回の実装で使った32 epochでは性能が出ないのも納得である。
とはいえ、Train Time, Inference Timeともに大きいので、実用上はどうかという問題は拭えない。CNNの帰納バイアスの強さが体に沁み渡る...
結果
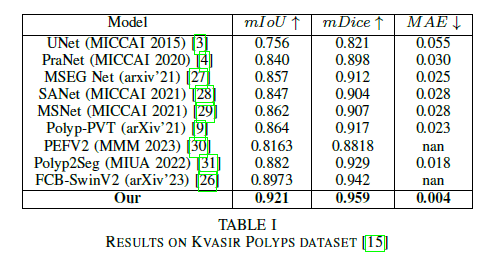
Kvasirの結果だけ見るが、ほかもだいたい同じような結果になっていた。この表の通り、Meta-Polypで使われたCAFormerのエンコーダデコーダモデルはセグメンテーションの精度が高く、構造もシンプル。2年前くらいにちょっと流行ったSwin-UNetもこのアーキテクチャのインスタンスだと考えることもできるかもしれない。
ただ、この論文ではパラメータ数やFLOPS、より現実的な推論時間などについて触れられていないので、少し惜しい。
ablation studyではU-Netとモジュールを差し替えた場合の精度について論じているが、そもそもU-Net自体古すぎて、論じられているグローバルな特徴に関してのCAFormerの性質かどうかは他のモダンなモデルにも当てはまる言い方であったり、惜しい所が散見される。CAFormerのブロック、特にMulti-Scale特徴量のConvFormerのToken-Mixer種別に関する差分を取って、学習後の出力特徴量のヒートマップを取るなどの実験(解析)をした方が内容も充実したと思う。
感想
2023までの内視鏡画像セグメンテーションの軽いサーベイとして読みやすく、モデルもシンプルに組めるベースラインとして良い手法だと感じた。
しかしパラメータ数などの比較がないなどの疑問が残る部分もあり、これに関しては自分で確認してみたが、やはりCAFormer-S18を丸々1つ使っているので非常に重い。自作モデルでかなり良い性能がでたので、もう少し詰めることができれば論文化できるかもしれない。と思いつつ、ただ性能の出るモデルを作るだけでなくドメイン知識を盛り込んだ新規性がほしいという考えもあり、このバランスは難しいところだと思う...
Discussion