faiss で部分集合を検索する [情報検索・検索技術 Advent Calendar 2023]
こちらは 情報検索・検索技術 Advent Calendar 2023 の 12/8 の記事です。
ベクトル検索をするときに部分集合の検索をしたい
昨今 RAG(Retrieval Augmented Generation) の影響でベクトル集合での検索がかなり流行っています。元々別に下火だったわけではないですが、注目度はかなり上がりました。
ほとんどの場合では faiss, 最近だと weaviate や chroma などの Vector DB を使うと思います。大体の場合でやりたいのは事前に登録したベクトル全てに対して検索をすることだと思いますが、時々部分集合に絞った検索をしたくなることがあるのではないでしょうか。
ここでいう部分集合に絞った検索は https://weaviate.io/developers/weaviate/concepts/prefiltering のような where 句で絞り込めるインタフェースとは違って、検索したい ID を事前に渡す方式をイメージしています。

ここで、subset はランダムな ID が渡されるものとします。
意外とこの部分集合のベクトルを検索する、というインタフェースを備えているライブラリはあまり多くない気がしています。ざっと調べた限りではメジャーな選択肢だと faiss しか見当たりませんでした。他にご存知のかたいましたらこっそり教えていただけると🙏
なぜ部分集合の検索をしたいのか?
ベクトル集合の検索をするとき、全集合を検索するほうが最適化されていて早いし、絞った集合を検索するなら最初からその絞った集合でインデックスを作る方が良いです。でも、時々検索したい集合がランダムに与えられる場合があるんじゃないでしょうか。
例えば
- Two-Stage レコメンドで RecVAE などでレコメンドできるアイテムを用意した後、2段目ではクエリとの類似度で並び替えて似ているアイテムを出したいとか
- 同様に何らかの手法でレコメンド候補を用意した後に、新着アイテムの中で近いアイテムに置き換えて出したいとか
いずれの方法も精度を上げたいというよりは、ビジネス要件に依るものがほとんどかなと思います。
faiss で部分集合の検索を行う
では早速本題に入っていきたいと思います。どうやって部分集合の検索を行うのかみていきましょう。
今回用いるのは meta が開発している OSS である faiss です。この手のライブラリの中ではそれなりに前からある、という感じです。発表されたのは 2017 年。
普通にベクトルの検索を行う場合は次のようなコードになります。
import numpy as np
import faiss
# データセットのパラメータを設定
d = 64 # ベクトルの次元
nb = 10000 # データベースに追加するベクトルの数
nq = 100 # クエリに使うベクトルの数
# データセットをランダムに生成(ここでは単に例として)
np.random.seed(1234) # 乱数シードを固定
db_vectors = np.random.random((nb, d)).astype('float32')
query_vectors = np.random.random((nq, d)).astype('float32')
# faiss インデックスの作成(ここでは、単純なフラット(L2距離)インデックスを使う)
index = faiss.IndexFlatL2(d) # L2距離でフラットインデックスを作成
# データベースのベクトルをインデックスに追加
index.add(db_vectors)
print('Number of vectors in the index:', index.ntotal)
# クエリを実行して最も近いベクトルを検索
k = 4 # 各クエリに対して上位k個の近傍を検索
D, I = index.search(query_vectors, k) # Dは距離のリスト、Iはインデックスのリスト
print('Distances:', D)
print('Indices:', I)
簡単ですね〜 インデックスの種類でどれを使うか、などこの先がかなり奥深いのですが、単に使うだけであればあまり考えることはありません。
でも今回行うのは部分集合での検索です。それを行うには少し工夫が必要になります。
IDSelectorBatch か IDSelectorBitmap か
faiss のドキュメントは GitHub Wiki にあります。ここを丹念に読んでいくと、次のようなページが見つかります。
It is possible to select a subset of vectors, based on their ids, to search from.
さて、先程も述べたように今回はランダムに ID が与えられるものとします。その時使える選択肢は Wiki によれば IDSelectorBatch か IDSelectorBitmap か、とのことです。
いずれの場合でもまず全集合でのスコアリングが行われた後、部分集合での候補がフィルタされています。
それぞれの説明を引用しつつ、中身を説明してみます。
IDSelectorBatch
IDselectorBatch combines a hashtable (unordered_set) and a Bloom filter. Thus the lookup is in constant time, but there is some memory overhead.
The hash function used for the bloom filter and GCC’s implementation of unordered_set are just the least significant bits of the id. This works fine for random ids or ids in sequences but will produce many hash collisions if lsb’s are always the same
かなり曖昧な説明になりますが、ここでいう Bloom Filter はビットを用いてある要素が集合に所属しているかを調べる手法です。そこで使われているハッシュ関数は least significant bits(最下位ビット) だよ、と書いてありますが、これはビットで表現されたときの一番右端の値のことを言います。ここを使ってフィルタリングをやっています。
IDSelectorBitmap
IDSelectorBitmap takes an array with 1 bit per vector id. It is particularly interesting when a significant subset of vectors needs to be handled and ids are sequential. Then it is more compact and faster than IDselectorBatch.
IDSelectorBitmap ではまず事前に全アイテムに対して、それが探索したい集合に所属するかどうかのフラグを立てておき、それを bitmap として持っておきます。これを使ったフィルタです。
計算量
| class | storage | lookup cost |
|---|---|---|
| IDSelectorBatch | O(k) | O(1) |
| IDSelectorBitmap | O(n) | O(1) |
storage は部分集合検索用のオブジェクトを用意するときにかかる計算量、lookup cost は検索をするときにかかる計算量です。ここで k は部分集合のサイズ、n は全集合のサイズを表しています。
部分集合が大きければ大きいほど IDSelectorBatch は時間がかかりますが、基本的に k < n なので、IDSelectorBitmap よりは IDSelectorBatch のほうが早そうですね。
IDSelectorBatch と IDSelectorBitmap の速度比較
大体そうなのか、ということはわかりました。せっかくなので実際の速度も調べましょう。
実際のノートブックはこちら。
まず、調べるのに必要な関数を用意していきます。
部分集合に対する検索を行うコードは以下を参照しつつ書きました。
import faiss
import numpy as np
import random
import pandas as pd
def init(total_numbers: int) -> tuple[faiss.IndexFlatL2, np.ndarray]:
# データセットのパラメータを設定
d = 64 # ベクトルの次元
nb = total_numbers # データベースに追加するベクトルの数
nq = 100 # クエリに使うベクトルの数
# データセットをランダムに生成
np.random.seed(1234)
db_vectors = np.random.random((nb, d)).astype('float32')
query_vectors = np.random.random((nq, d)).astype('float32')
index = faiss.IndexFlatL2(d)
index.add(db_vectors)
print('Number of vectors in the index:', index.ntotal)
return index, query_vectors
def subset_search_batch(total_numbers: int, index: faiss.IndexFlatL2, query_vectors: np.ndarray, search_num: int) -> None:
search_target_ids = random.sample(range(total_numbers), search_num)
sel = faiss.IDSelectorBatch(search_target_ids)
params = faiss.SearchParametersIVF(sel=sel)
similar_recipe_scores, similar_recipe_indices = index.search(
query_vectors, k=10, params=params
)
def subset_search_bitmap(total_numbers: int, index: faiss.IndexFlatL2, query_vectors: np.ndarray, bitmap: np.ndarray, search_num: int) -> None:
search_target_ids = np.array(random.sample(range(total_numbers), search_num))
bitmap[search_target_ids] = True
bitmap = np.packbits(bitmap, bitorder='little')
sel = faiss.IDSelectorBitmap(bitmap)
params = faiss.SearchParametersIVF(sel=sel)
similar_recipe_scores, similar_recipe_indices = index.search(
query_vectors, k=10, params=params
)
subset_search_batch・subset_search_bitmap で実際の検索処理を行います。
search_target_ids = np.array(random.sample(range(total_numbers), search_num))
とあるように、特にシードを固定せずに毎回ランダムな検索候補が与えられるものとします。また、index.search に渡す k は特に変化させず毎回 10 としました。
後はこれを使って、次のようなコードで速度を測ります。
total_numbers = 10000
index, query_vectors = init(total_numbers)
bitmap = np.zeros(total_numbers, dtype=bool)
# セル上で実行しているものとしてください
%%timeit -r 100
subset_search_batch(total_numbers, index, query_vectors, search_num=10)
%%timeit -r 100
subset_search_bitmap(total_numbers, index, query_vectors, search_num=10)
ipython, jupyter notebook で使える magic command: timeit を使っています。timeit はデフォルトだと 7回実行する設定ですが、今回は先述したようにランダムに検索候補を選ぶので結果がブレやすいことから実行回数を 100 と多めに設定しました。
全集合のサイズが 10,000 のとき
まずは小手調べで全部で 10,000 個のベクトルがある集合からいきます。ここから部分集合の数を 10→100→1000 と増やしていった場合の結果がこちらです。

予想通りで、IDSelectorBatch を使う方が良さそうですね。ただ、どちらも std がかなりでかいです。IDSelectorBatch については検索候補をランダムに生成している影響で、程よく ID をビットで表現したときに検索しやすい、しにくい構造になってしまっているんでしょうか。
IDSelectorBitmap ですが、lineprof で見てみるとこんな感じです。(なお、ランダムで生成した結果を検索対象とする都合で以下の結果では IDSelectorBatch と IDSelectorBitmap であまり差が出ない結果となっています)
Timer unit: 1e-09 s
Total time: 0.0301414 s
File: <ipython-input-6-6b84a203f4fc>
Function: subset_search_bitmap at line 9
Line # Hits Time Per Hit % Time Line Contents
==============================================================
9 def subset_search_bitmap(total_numbers, index, query_vectors, bitmap, search_num):
10 1 131080.0 131080.0 0.4 search_target_ids = np.array(random.sample(range(total_numbers), search_num))
11 1 10575.0 10575.0 0.0 bitmap[search_target_ids] = True
12 1 30179.0 30179.0 0.1 bitmap = np.packbits(bitmap, bitorder='little')
13 1 56108.0 56108.0 0.2 sel = faiss.IDSelectorBitmap(bitmap)
14 1 36807.0 36807.0 0.1 params = faiss.SearchParametersIVF(sel=sel)
15 2 29876130.0 1e+07 99.1 similar_recipe_scores, similar_recipe_indices = index.search(
16 1 483.0 483.0 0.0 query_vectors, k=10, params=params
17 )
対して IDSelectorBatch だとこんな感じでした。
Timer unit: 1e-09 s
Total time: 0.0361767 s
File: <ipython-input-5-6b84a203f4fc>
Function: subset_search_batch at line 1
Line # Hits Time Per Hit % Time Line Contents
==============================================================
1 def subset_search_batch(total_numbers, index, query_vectors, search_num):
2 1 87654.0 87654.0 0.2 search_target_ids = random.sample(range(total_numbers), search_num)
3 1 12163894.0 1e+07 33.6 sel = faiss.IDSelectorBatch(search_target_ids)
4 1 63514.0 63514.0 0.2 params = faiss.SearchParametersIVF(sel=sel)
5 2 23861126.0 1e+07 66.0 similar_recipe_scores, similar_recipe_indices = index.search(
6 1 545.0 545.0 0.0 query_vectors, k=10, params=params
7 )
IDSelectorBitmap は bitmap[search_target_ids] = True あたりが全集合のサイズが増えていくごとにどんどん伸びていきそうですね。
IDSelectorBatch は faiss.IDSelectorBatch(search_target_ids) でパラメータを作っているところでドカッと処理がかかっています。
なので、全集合のサイズが小さく、部分集合のサイズが大きい場合は IDSelectorBitmap のほうが安定する場面もありそうです。
全集合のサイズが 100,000 のとき
続いて、全集合のサイズを10倍にします。
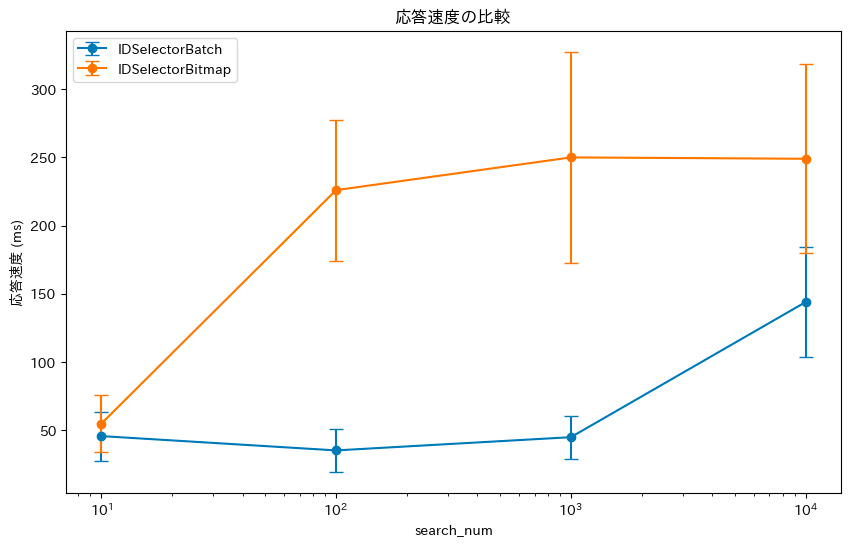
こうしてくるとどんどん差が開いていきますね。
全集合のサイズが 1,000,000 のとき
さらにもう10倍サイズを増やしてみました。

意外なことにこの場合では IDSelectorBitmap が拮抗するところがありました。k のサイズが小さい場合はほとんど互角で、ただ k が増えていくごとに差は開いていきました。
まとめ
ということで、faiss を使ってベクトルの部分集合を検索する方法について見てきました。
一部シチュエーションで IDSelectorBitmap が良い場合がありましたが、殆どの場合で IDSelectorBatch の方が優れていたことを考えると、大体どの場面でも IDSelectorBatch を利用するのが良さそうです。
ただ、注意点として探索したい部分集合のサイズ: k が増えるごとに相当遅くなっていく性質を持っています。また、探索したい部分集合によっては予想以上に長い時間がかかってしまうこともあり、その点も注意が必要です。
よいベクトル検索ライフを〜!
Discussion
部分空間という表現を部分集合に変更しました
context: https://x.com/fukkaa1225/status/1732910823398650207?s=20