scikit-learn の PCA で GPU を活用する
目的
Scikit-learnが実験的にGPU対応していたので調査してみた! という良記事があって、一部分 scikit-learn で GPU を活用できるようなので PCA (主成分分析) を試してみたい。
どのくらい対応している?
11.1. Array API support (experimental) を見ると LinearDiscriminantAnalysis 以外にも decomposition.PCA もいけそうなので、今回はこれを Google Colab 上の T4 で試す。
実装
まずは、従来通りに CPU で試して、次に PyTorch の Tensor を突っ込んでみるという形をとる。
以下で最新の scikit-learn と、GPU を活用する時に必要になる array-api-compat をインストールする。
%%bash
pip install -qU "scikit-learn>=1.4.0" array-api-compat
次に必要なモジュールを import する。
from __future__ import annotations
import os
import matplotlib.pyplot as plt
import numpy as np
from sklearn import config_context
from sklearn.decomposition import PCA
import torch
from torch.utils.data import Subset
import torchvision
import torchvision.transforms as transforms
データセットは何でも良いのだが、今回は QMNIST を使って数字の「0」と「1」のデータで PCA してみる。
root = os.path.join(os.getenv('HOME'), '.torch')
transform = transforms.Compose([
transforms.ToTensor(),
transforms.Normalize(0.5, 0.5)
])
dataset = torchvision.datasets.QMNIST(
root=root,
train=True,
download=True,
transform=transform
)
indices = []
for i, target in enumerate(dataset.targets):
target = int(target[0])
if target == 0 or target == 1:
indices.append(i)
zero_one_dataset = Subset(dataset, indices)
print(len(zero_one_dataset))
12665
お手頃なサイズ感である。
CPU 版
まずは CPU で試すのでデータの準備をする。
data = np.array([tensor[0].numpy().flatten()
for tensor in zero_one_dataset], dtype=float)
labels = [tensor[1] for tensor in zero_one_dataset]
print(data.shape)
(12665, 784)
以下で PCA を実行。可視化したいのでコンポーネント数を 2 とする。
%%time
pca = PCA(n_components=2)
pca.fit(data)
data_pca = pca.transform(data)
CPU times: user 1.06 s, sys: 202 ms, total: 1.26 s
Wall time: 680 ms
ほぼ一瞬で完了。結果を可視化する。
x_z = []
y_z = []
x_o = []
y_o = []
for i, (dat, label) in enumerate(zip(data_pca, labels)):
if i >= 100:
break
if label == 0:
x_z.append(dat[0])
y_z.append(dat[1])
else:
x_o.append(dat[0])
y_o.append(dat[1])
plt.figure()
plt.scatter(x_z, y_z, c="r")
plt.scatter(x_o, y_o, c="b")
plt.xlabel("component-1")
plt.ylabel("component-2")
plt.show()
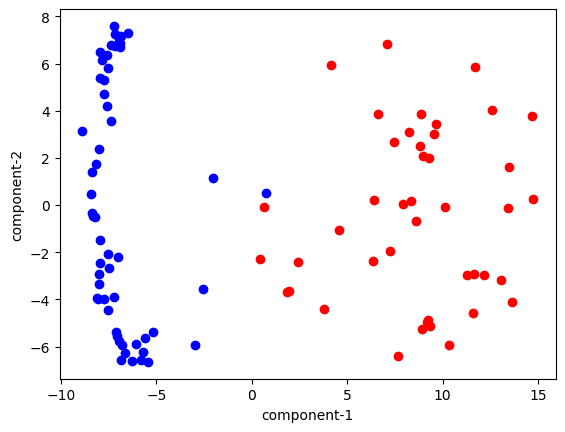
特に驚きもない結果を得た。
GPU 版
まず、データを PyTorch の Tensor にする。
data2 = torch.asarray(data, device="cuda", dtype=torch.float32)
次に 11.1. Array API support (experimental) を参考に PCA を使う。
(with
svd_solver="full",svd_solver="randomized"andpower_iteration_normalizer="QR")
と書いてあるのにでこれに注意する。svd_solver="randomized" のほうが速いのでこれを使ってみた。
%%time
with config_context(array_api_dispatch=True):
pca2 = PCA(n_components=2, svd_solver="randomized",
power_iteration_normalizer="QR")
pca2.fit(data2)
data_pca2 = pca2.transform(data2)
CPU times: user 17.9 ms, sys: 0 ns, total: 17.9 ms
Wall time: 18.7 ms
CPU で 680 ms だったものが、20 ms 前後になったので、他のケースでも速くなりそうな気がする。
念のために可視化する。
x_z = []
y_z = []
x_o = []
y_o = []
for i, (dat, label) in enumerate(zip(data_pca2.cpu().numpy(), labels)):
if i >= 100:
break
if label == 0:
x_z.append(dat[0])
y_z.append(dat[1])
else:
x_o.append(dat[0])
y_o.append(dat[1])
plt.figure()
plt.scatter(x_z, y_z, c="r")
plt.scatter(x_o, y_o, c="b")
plt.xlabel("component-1")
plt.ylabel("component-2")
plt.show()

CPU 版との違いは目視では分からない。
まとめ
scikit-learn が一部 GPU に対応したぞ、やった~!という内容。
manifold.TSNE はまだらしい。tsne-cuda というのがあってこれも使いやすかったのだが、
We only support
n_components=2. We currently have no plans to support more dimensions as this requires significant changes to the code to accomodate.
とあるので注意が必要である。
Discussion