AlphaFoldでタンパク質-環状ペプチド複合体構造予測する方法
こちらの記事は創薬 (dry) Advent Calendar 2023 7日目の記事です。
今回の記事ではAlphaFoldを用いて、従来不可能とされていたタンパク質-環状ペプチド複合体構造を予測可能にした手法について記事を書きます。
はじめてZennを使うので、見づらい部分があるかもしれませんがご容赦ください。
AlphaFoldが何かを知らない方も含めてわかるように歴史も含めて書きます。
2023-12-23追記:偶数奇数を考慮したoffsetのベンチマーク関連
2023-12-23追記:上記は間違いの実装でした。修正中
2023-12-23追記:修正したので、 ベンチマークの最終結果を載せました
AlphaFoldとは
AlphaFoldは、2021年にNatureにそのアーキテクチャーと詳細のアルゴリズムが公開された[1]。

また、同時にAlphaFold v2.0.0がGitHubに公開され[2] 、世界中の研究者がアミノ酸配列を入力とし、その3次元構造を超高精度に予測することを可能にした。
歴史的にには、Critical Assessment of Structure Prediction (CASP)という、Protein Structure Prediction Centerが主催する、アミノ酸配列からタンパク質の3次元構造を予測技術の現状を明らかにするためのコミュニティコンテストがあり、CASPの参加者は実験的な構造がまだ公開されていないタンパク質の予測を試みるモデルを提出する[3]。 2020年に開催されたCASP14では、DeepMind社のAlphaFold2 teamが用いたモデルが、2位と圧倒的な差をつけたことでも有名である[4]。 なお、他にもBaker labが開発したRoseTTAFold[5]や、Metaが開発したMSAを使わない言語モデルであるESMFold[6] などもタンパク質構造予測モデルである。
単量体しか学習していないAlphaFoldで複合体予測する
AlphaFoldのモデルが公開されたのち、様々な研究者がAlphaFoldを用いて、自身の非公開構造データをとの比較などを行いAlphaFoldの精度の検証がされた。また、タンパク質とタンパク質やペプチドを長いグリシン配列でつなぐこと (G-linker法)で、タンパク質-タンパク質複合体やタンパク質-ペプチド複合体が予測できることをX(旧Twitter)上で議論された。 G-linker法でタンパク質-タンパク質複合体を世界で初めて発信したYoshitaka Moriwakiさんのこの投稿である。
そして、G-linker法でタンパク質-ペプチド複合体については、Masahito Ohueさんのこの投稿である。
さらに、Minkyung Baekさんによって、入力特徴の一つである入力配列のインデックスであるresidue_indexに比較的大きなギャップを設定すると"Chain break"と呼ばれる、タンパク質のChainが分断される現象が起こることも示された。
この方法は、G-linker法に比べて見た目も美しく、後に、誰でも簡単にWebUIで構造予測ができるColabFold[7] やAlphaFold-Multimer[8] にも取り込まれる手法である。
その後の研究で、AlphaFoldは単量体しか学習しないにもかかわらず、タンパク質複合体やタンパク質-ペプチド複合体を予測できることがいくつかの論文で示された[9] [10] [11]。
その後、AlphaFoldは一本鎖予測だけでなく、タンパク質複合体予測にも一般化できることが示され、AlphaFoldの改良版であるAlphaFold-Multimerがタンパク質複合体予測のためにリリースされた。
環状ペプチドとは
ペプチドとは2~50のアミノ酸がつながったポリアミドであるが、その長さの閾値の厳密な定義は存在しない。
基本的にはペプチドにはタンパク質と同様、N末端とC末端が存在するが、この末端同士がペプチド結合し、環状化したペプチドのことを環状ペプチドと呼ぶ。以下は3AV9のLEDGF peptideであり、末端同士が結合しているため環状構造を作っていることがわかる。
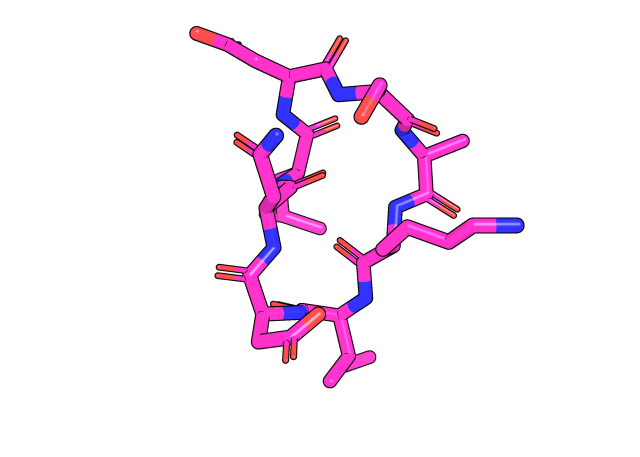
他にもシステインのS同士のジスルフィド結合によって環状化したものも環状ペプチドと呼ばれる。今回この記事で、取り扱うのは前者のHead-to-tail型の環状ペプチドの構造予測である。(ただし、N末端とC末端以外の部分のシステイン同士がジスルフィド結合しているものは含む)
AlphaFoldでは、直鎖タンパク質・直鎖ペプチドを前提しているため、このような環状ペプチドについては予測できなかったが、2023年に、Cyclic offsetと呼ばれる特殊なoffsetを用いることで、環状ペプチドをAlphaFoldで予測できるという報告があった[12]。 この論文のFigure 1を引用する。
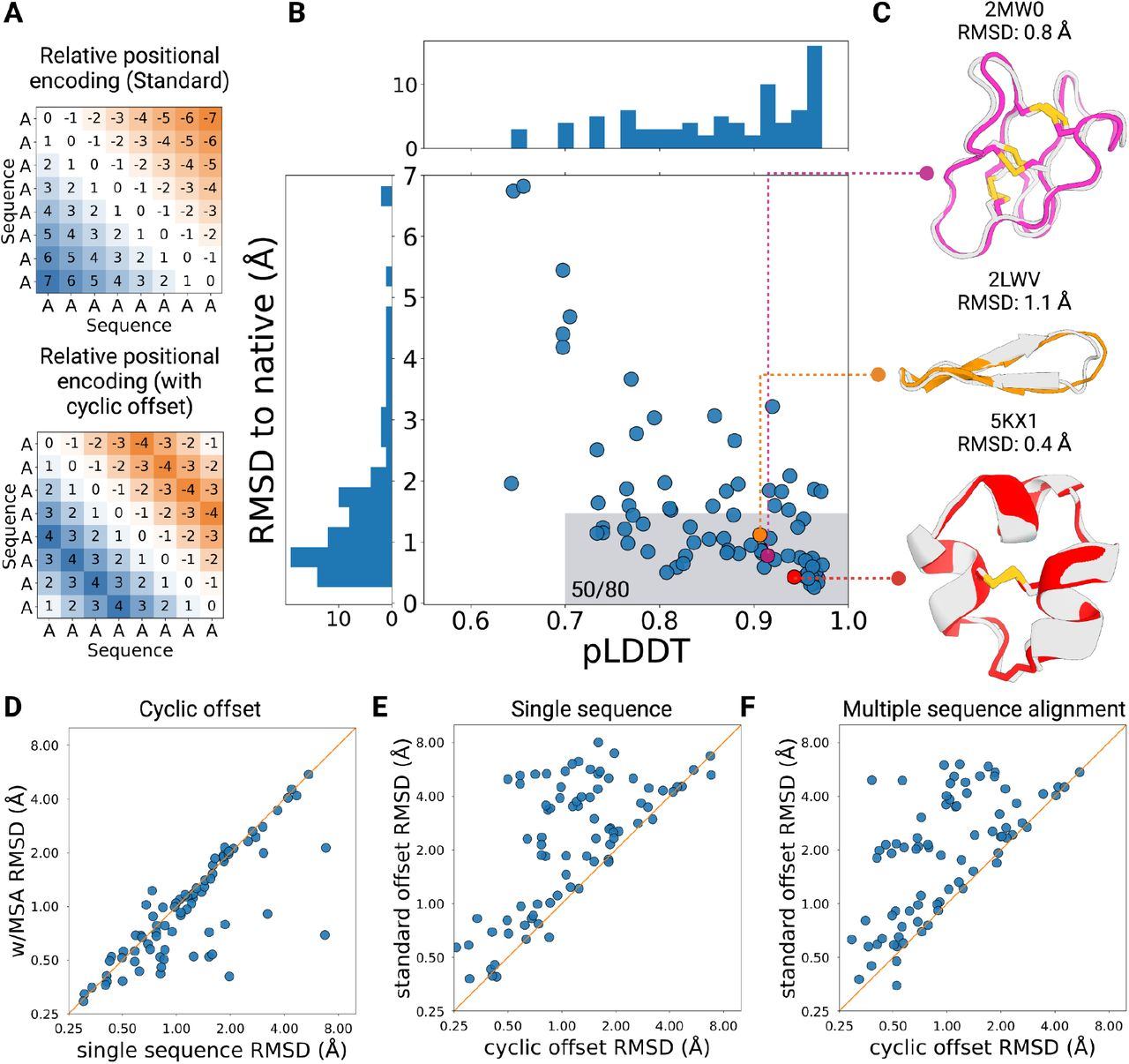
アルゴリズムとしては、入力配列のインデックスから作られる2次元特徴量である、offset配列を、循環的にすることで、AlphaFoldがN末端とC末端をつなげることができたということである。 AlphaFoldの特徴量に何かしらの細工をするにはEvoformerにデータが入る手前の部分であればできる(経験的にそうなだけで実はできるのかもしれない)。

しかし、このCyclic offsetは後にCyclic permutation test (つまり、環状ペプチドなので、予測する際の入力の文字の開始位置に依存せず、同一になるかどうかのテスト)を行うと、ズレてしまうことがわかり、改良された。
Cyclic peptide complex offset
これまでの章で、本題のタンパク質-環状ペプチド複合体構造予測に関する予備知識を書き記した。
次に、これまでの技術を用いて、どの様にタンパク質-環状ペプチド複合体構造の予測を行うのかを考えてみよう。
私が、真っ先に思ったことは、複合体予測におけるoffset配列は対角成分に目的のタンパク質及びペプチドのoffsetが作られる。このoffsetの内ペプチド部分だけを環状ペプチドにすることで、タンパク質-環状ペプチド複合体構造予測ができるのではないか?という仮説に基づき、AlphaFoldの実装の改変を試みた。
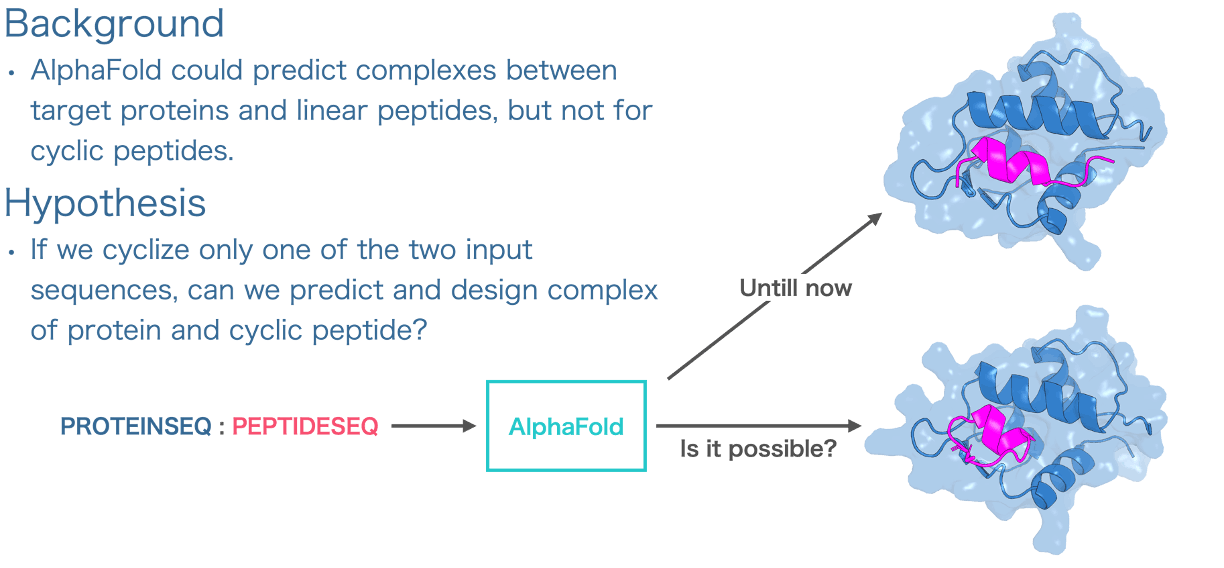
アイディアとしてはとてもシンプルであるため、非常にわかりやすい。開発したoffsetとしては以下の図のように、AlphaFoldのgap法で複合体を作成する際のoffsetのペプチド部分をcyclic offsetに変更するだけである。 私の論文ではCyclic peptide complex offsetと名付けている[13]。
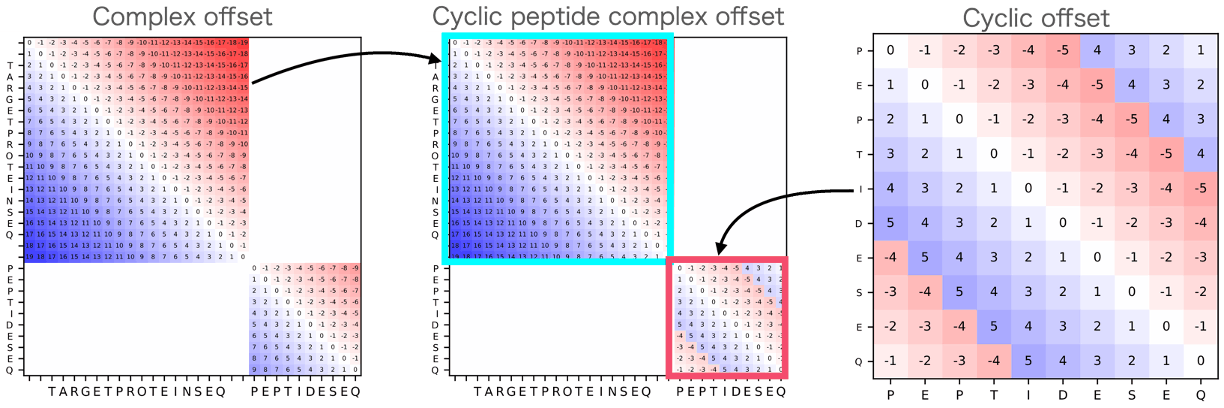
Cyclic peptide complex offsetを導入し、タンパク質-環状ペプチド複合体構造予測を可能にし、
PROTEINSEQ:PEPTIDESEQという入力方式でタンパク質-環状ペプチド複合体を予測できるようにしたColabFoldがColabFold-cycpep-dockである。 興味がある方は、Colabで実行可能なので、是非試していただきたい。
タンパク質-環状ペプチド複合体構造予測
実際にタンパク質-環状ペプチド複合体構造予測をしてみると、かなり精度が良いことがわかった。
ここでは、AlphaFoldには不利な条件であるが、タンパク質-環状ペプチド複合体のinterface情報を用いるドッキングツールであるAutoDock CrankPep (ADCP)を比較相手としている[14]。

使っているモデルは、alphafold_multimer_v2である。これはptmモデルとMultimerモデルを比較すると圧倒的にMultimerモデルの方が精度よいため、Multimerモデルを選択した。(下図は私の論文のsupplementary Figure S2である。)

alphafold2_multimer_v3が最新モデルではあるがalphafold2_multimer_v2をつかっている。これは、v2までは、学習範囲として、タンパク質の長さが16残基以上だったが、v3から学習範囲が変わって、4残基以上のタンパク質が学習範囲になったということをAlphaFold teamに確認したためである。
interface情報があるADCPよりも基本的には良い成果であることが確認できる。ただ、Head-to-tail型のタンパク質-環状ペプチドの例は数えるほどしかなく、これらはそのうちのいくつかである。当然ADCPの方が制度が良いものもあるが今回は載せていない(論文には載っている)。
Cyclic offsetはまだ改良の余地があった
そして、私が先日Xで投稿した内容ではあるが、Cyclic permutation testという観点では、Cyclic offsetは実はまだ不完全であることが判明した。
具体的にどういうことかと言うと、bugfixされたCyclic offsetは「奇数」の長さの環状ペプチドに関してはCyclic permutation testを通るが、「偶数」の長さの環状ペプチドに関してはCyclic permutation testが通らないということである。
まず単量体から説明する
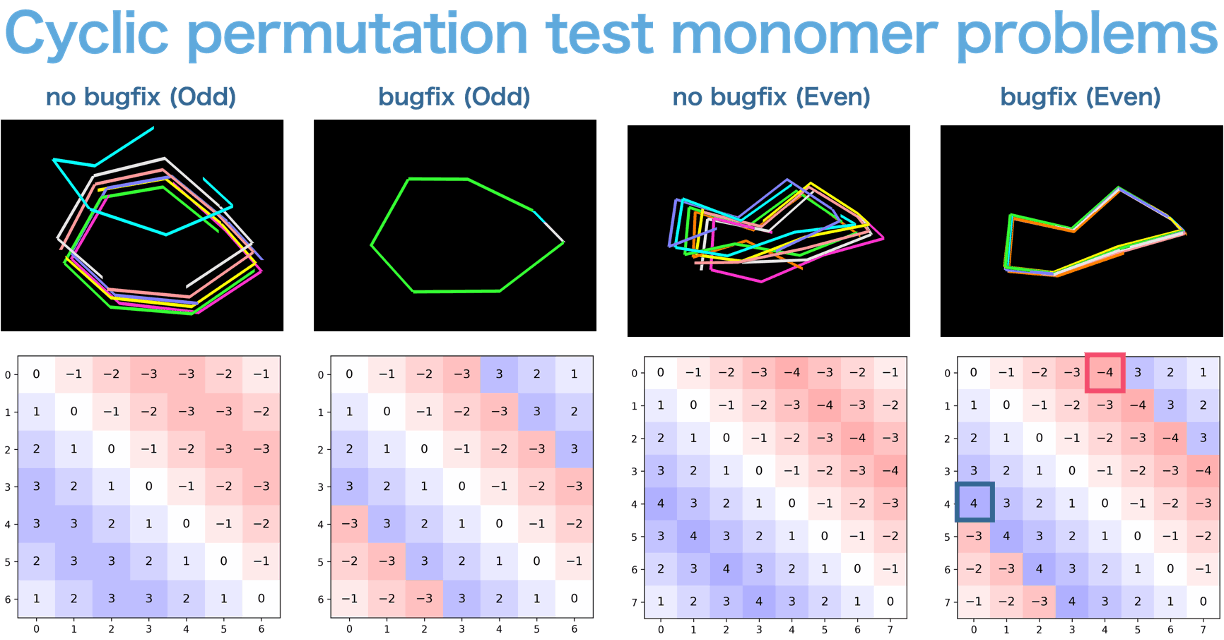
上図は単体の環状ペプチドにCyclic permutation testを行ったものだが、奇数のものは、bugfix offsetであれば、同一になるが、偶数の長さにすると、bugfix offsetでも、ごくわずかにズレが生じる(これは計算誤差よりも大きい。)
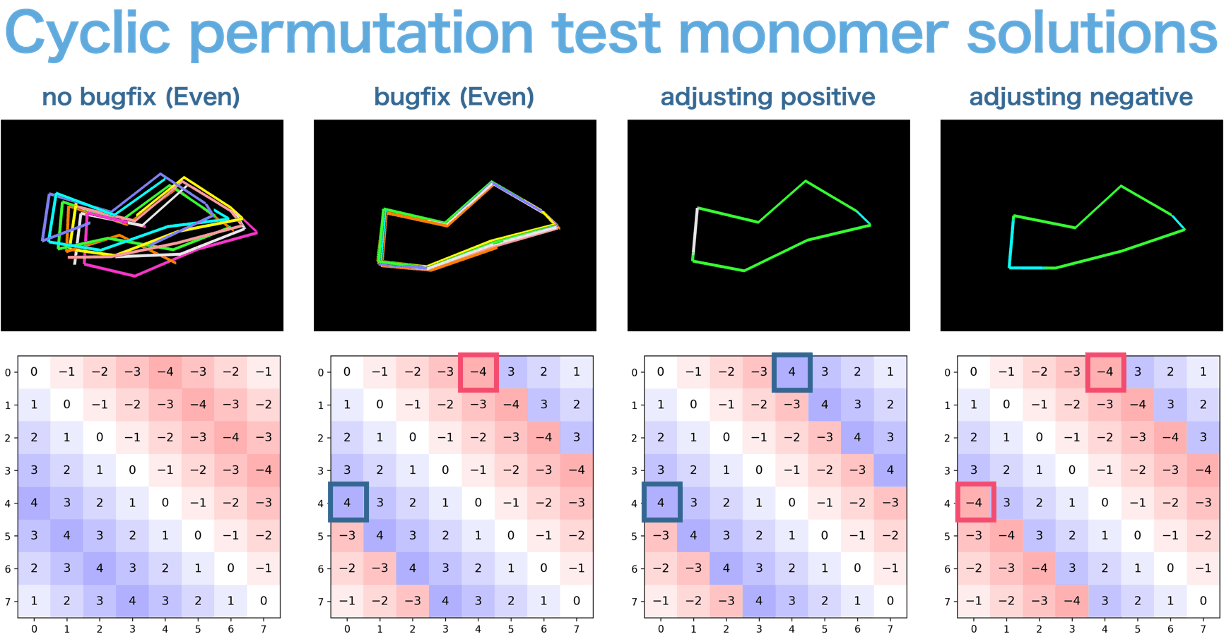
上図は、単量体のペプチドの長さが偶数のときのCyclic offsetをどの様に変更すれば良いかを示した図である。この問題は、偶数の真ん中を整数値で取ることができないことに起因しており、枠でくくった部分を開始点取して右下方向へ符号を揃える(正 or 負)完全に同一になることがわかった。
次にこれが複合体においても適用できるのかを検討した。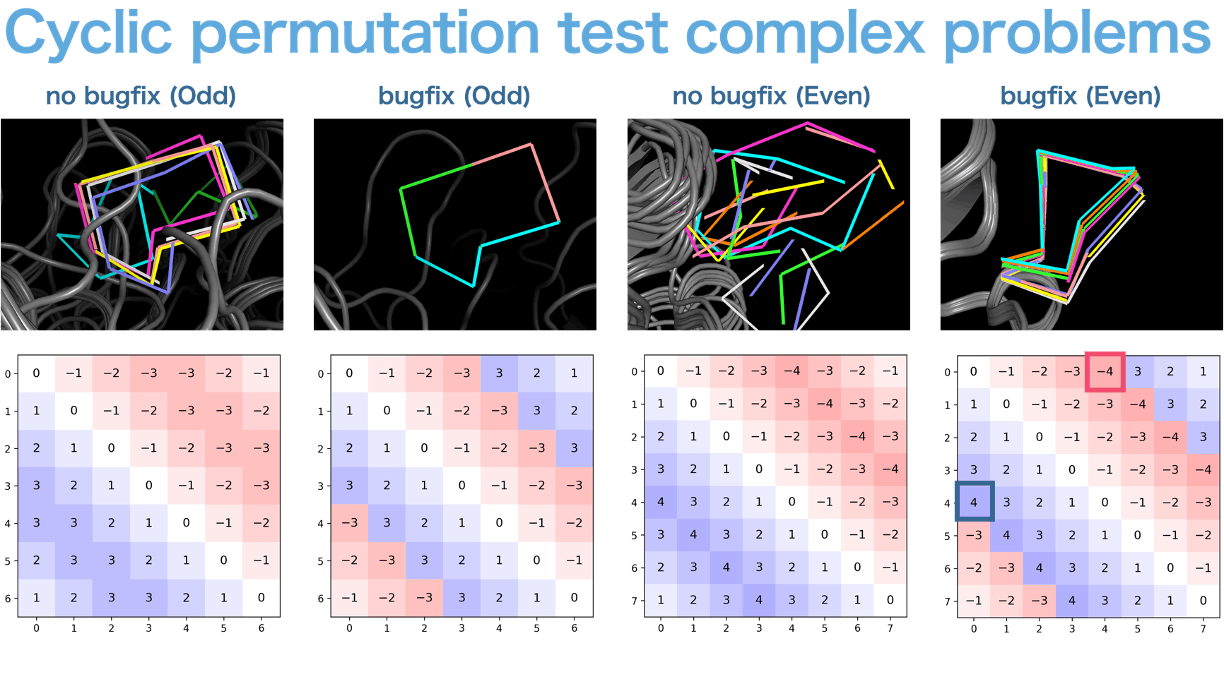
上図は複合体の環状ペプチドにCyclic permutation testを行ったものだが、奇数のものは、bugfix offsetであれば、同一になるが、偶数の長さにすると、bugfixしても、明らかにズレが生じる。
(bugfixがしないものが鎖が切れているのはありえない構造の配置になっているからと思ってもらえればよい。このCyclic permutation testを行うには、mlmをdisableしたり、bfloat16をdisableしたり、recycleする前の構造を取得する必要があるが、この記事では説明を割愛する。)
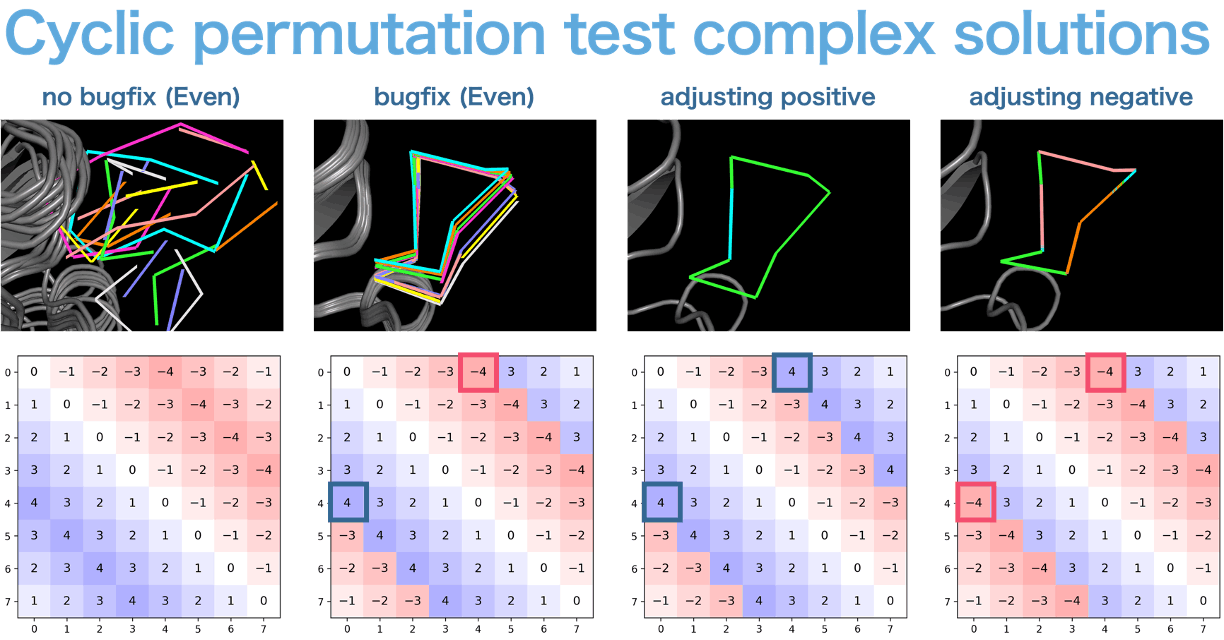
そしてこれを同様に解決できるかを検討した。
上図は、複合体のペプチドの長さが偶数のときのCyclic offsetをどの様に変更すれば良いかを示した図である。単量体のときと同じく、枠でくくった部分を開始点取して右下方向へ符号を揃える(正 or 負)完全に同一になることがわかった。(わずかに色がズレて見えるのはおそらく計算誤差であるfloat16の限界)
以上により、どんな長さの環状ペプチドであっても、Cyclic permutation testを通すことが可能なCyclic offsetを示すことができた。
しかし、実はこのCyclic permutation testを通るかどうかと、実際の予測精度が良くなるのかどうかということは別問題のようで、ベンチマークをとると精度はそれほど変わらないということをColabFoldコミュニティから聞いている。(私は検討していない。)
まとめ
- AlphaFoldは高精度のタンパク質構造予測モデルである。
- AlphaFoldは単量体しか学習してないモデルでも複合体が予測できる。
- AlphaFoldは直鎖タンパク質・直鎖ペプチドしか予測できなかったが、環状ペプチドも予測できるようになった。
- 私が開発したCyclic peptide complex offsetを用いるとタンパク質-環状ペプチド複合体を予測することができるようになった
- 単量体・複合体共に、どんな長さの環状ペプチドであってもCyclic permutation testを通すことができるCyclic offsetの検証ができた
- タンパク質-環状ペプチド複合体を予測をしたい方はぜひこちらのColabFold-cycpep-dockを使ってほしい(オプションを選べば環状ペプチド単体も実行できる)
- **2023−12−23 追記:ベンチマークをとると、bugfixの有無で精度が変わらないという結果が出た。(AlphaFoldは細かいことは吸収してしまうのか・・・) **
- positiveとbugfix, negativeとbugfixもほぼ差がない。
追記
入力配列長の偶奇を考慮したoffsetのベンチマーク
せっかくなのでやってみた。
データは元論文で使われていた、80の環状ペプチドのNMRデータを正解構造として、AlphaFoldで予測した構造を1つNMRの全モデルに対してRMSDを測定し、平均化値を今回の1つの点のRMSDとして散布図を描いた。
そうすると驚愕の事実がわかる。グラフをおいておくので各自この現象について考えよう。
(私の計算が間違っている可能性はある)
まず、bugfixされたoffsetを基準で考えることとする(positive, negativeはそもそもbugfixありきであるため)
これは、bugfixの有無の比較である。

Cyclic permutation testなんてどうでもいいらしい。bugfix offsetを使おうが最初のcyclic offsetのままだろうが、精度に関係ないことが分かる。
そして、positiveとnegativeをそれぞれ行うと、これもほぼ影響がない。少しくらい良くなってくれたらいいのにと思うが、誤差の範囲と言えるレベルだろう。
これがpositiveの結果

これがnegativeの結果
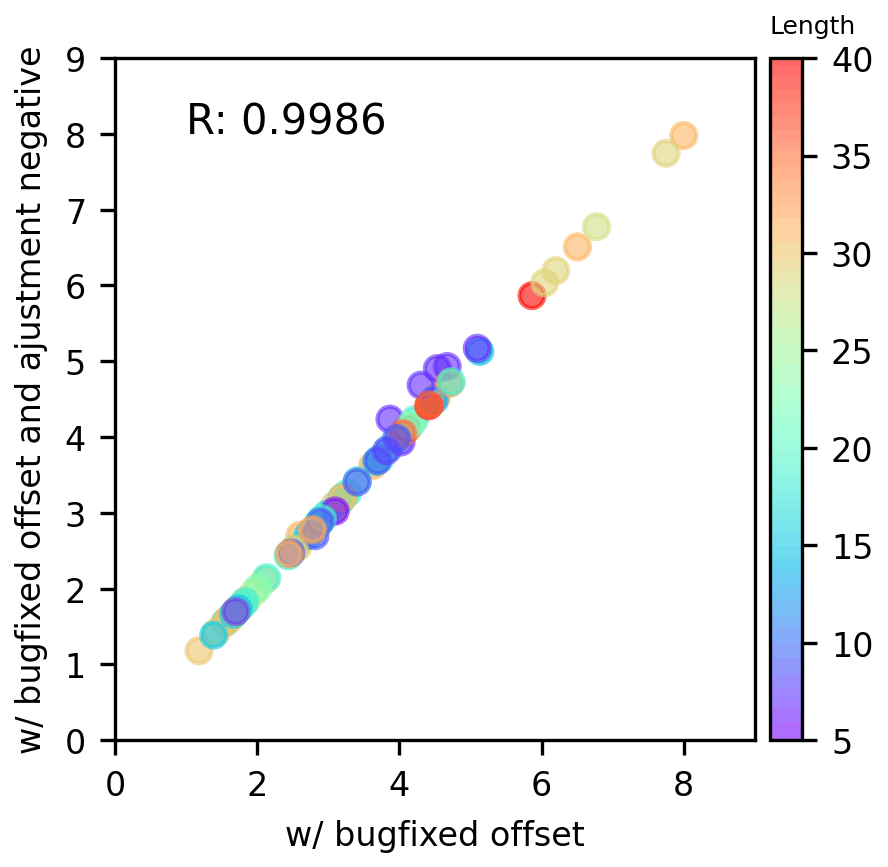
ということは、cyclic permutationがidenticalであることは精度にほぼ影響がないということだろうか・・・
以上で検討は終わり。本当に精度をきちんと見るのであれば、散布図ではなくRMSDが下がっているかどうかの箱ひげなどにするべきだろうが、差があまりに小さすぎて判別できない。
驚くべきは、bugfixの有無で精度差が無いことだ。AlphaFoldはoffsetの微弱な影響すら吸収してしまうのだろうか・・・
みなさんの意見を待つ。
勢いでやって間違ってたやつ
参考文献
-
Jumper, J. et al. Highly accurate protein structure prediction with AlphaFold. Nature 2021, 596, 583–589. https://doi.org/10.1038/s41586-021-03819-2 ↩︎
-
AlphaFold:Available online: https://github.com/google-deepmind/alphafold ↩︎
-
Kryshtafovych, A. et al. Critical assessment of methods of protein structure prediction (CASP)―Round XIV. Proteins: Structure, Function, and Bioinformatics 2021, 89(12), 1607–1617. doi: 10.1002/prot.26237. url: https://doi.org/10.1002/prot.26237 ↩︎
-
Jumper, J. et al. Applying and improving AlphaFold at CASP14. Proteins: Structure, Function, and Bioinformatics 2021, 89(12), 1711–1721. https://doi.org/10.1002/prot.26257 ↩︎
-
Baek, M. et al. Accurate prediction of protein structures and interactions using a three-track neural network. Science 2021, 373, 871–876. https://doi.org/10.1126/science.abj8754 ↩︎
-
Lin, Z. et al. Evolutionary-scale prediction of atomic-level protein structure with a language model. Science 2023, 379, 1123-1130. https://doi.org/10.1126/science.ade2574 ↩︎
-
Mirdita, M. et al. ColabFold—Making Protein Folding Accessible to All. Nat. Methods 2022, 19, 679–682. https://doi.org/10.1038/s41592-022-01488-1 ↩︎
-
Evans, R. et al. Protein Complex Prediction with AlphaFold-Multimer. bioRxiv 2022. https://doi.org/10.1101/2021.10.04.463034. ↩︎
-
Xiong, H. et al. Evaluating the Reliability of AlphaFold 2 for Unknown Complex Structures with Deep Learning. bioRxiv 2022, https://doi.org/10.1101/2022.07.08.499384 ↩︎
-
Bryant, P. et al. Improved Prediction of Protein-Protein Interactions Using AlphaFold2. Nature Communications 2022, 13, 1265, https://doi.org/10.1038/s41467-022-28865-w ↩︎
-
Tsaban, T. et al. Harnessing Protein Folding Neural Networks for Peptide–Protein Docking. Nature Communications 2022, 13, 176, https://doi.org/10.1038/s41467-021-27838-9 ↩︎
-
Rettie, S.A. et al. Cyclic Peptide Structure Prediction and Design Using AlphaFold. bioRxiv 2023. https://doi.org/10.1101/2023.02.25.529956 ↩︎
-
Kosugi, T. and Ohue, M. Design of Cyclic Peptides Targeting Protein–Protein Interactions Using AlphaFold Int. J. Mol. Sci. 2023, 24, 13257. https://doi.org/10.3390/ijms241713257 ↩︎
-
Zhang, Y. and Sanner, M.F. Docking Flexible Cyclic Peptides with AutoDock CrankPep. J. Chem. Theory Comput. 2019, 15, 5161–5168. https://doi.org/10.1021/acs.jctc.9b00557 ↩︎
Discussion