【LiNGAM】複数グループのデータセットをまとめて因果探索する(Python)
はじめに
こんにちは、因果探索アプリケーション Causalas 開発の井手です。
今回は、統計的因果探索の手法であるDirectLiNGAMを複数グループのデータセットに拡張したアルゴリズムについて紹介し、Pythonで実際に複数グループのデータセットをまとめて因果探索する方法を説明します。
Python パッケージ
複数グループのデータセットに拡張したDirectLiNGAMのアルゴリズムを利用される方は、LiNGAMのPythonパッケージのMultiGroupDirectLiNGAMを参照してください。これに合わせて、以降の説明でもアルゴリズム名をMultiGroupDirectLiNGAMと表記します。
動作環境
- OS: Windows 11
- Python: v3.12.3
- lingam: v1.9.0
MultiGroupDirectLiNGAM
MultiGroupDirectLiNGAM[1]の最大の特徴は、複数のグループにまたがるデータセットに対して、各グループが同じ因果順序を共有するという仮定をおき、各グループ同時に因果順序を推定していることです。
利点
MultiGroupDirectLiNGAMを使う利点は以下の通りです。
-
複数グループのデータをまとめて因果探索することで精度が上がる
異なる実験条件や異なるデータソース(例:病院Aと病院B)のデータをまとめて因果探索することで、個別に因果探索するよりも精度が向上します。ただし、これは因果順序が同じという仮定に基づいているため、異なる因果構造のデータをまとめることはできません。 -
グループ毎の違いが明確になり、解釈が容易に
因果順序は同じですが、グループによって因果効果の値が異なるため、因果効果を比較することでグループ間の共通点と相違点が明確になり、結果の解釈がしやすくなります。 -
データの不足を補う
因果探索に使用するデータを増やすことで、データの不足を補うことができます。グループ間で因果構造が共通であるため、外れ値やノイズデータがあるデータへの頑健性も向上します。
MultiGroupDirectLiNGAMの計算方法
DirectLiNGAMのデータ生成モデルとMultiGroupDirectLiNGAMのデータ生成モデルを以下に示します。
またDirectLiNGAMやMultiGroupDirectLiNGAMでは、変数の中から外生変数(親がいない変数)を見つけて因果順序のトップに置くことで順に因果順序を推定しているのですが、この外生変数を見つけるために、変数とその残差の独立性を評価しています。
このとき、MultiGroupDirectLiNGAMの独立性評価の計算式は以下のようになります。
MultiGroupDirectLiNGAMでは複数のグループに共通する外生変数を見つける必要があるのですが、各グループのデータの重要度を考慮するために独立性指標の重み付き和を計算しています。
この重み
このように、各グループの独立性評価を重み付けして合計することで、全グループを統合した因果順序を決定しています。
複数グループのデータセットを因果探索する
では、実際にPythonを使って複数グループのデータセットを因果探索してみます。
使用するデータセット
本記事では、アイリスデータセットを使用します。
(データセットの配布元:https://archive.ics.uci.edu/dataset/53/iris)
以下のように3種類のアイリスの花(Setosa、Versicolor、Virginica)の特徴量(がく片の長さ、がく片の幅、花弁の長さ、花弁の幅)が含まれています。
| sepal_length (cm) | sepal_width (cm) | petal_length (cm) | petal_width (cm) | species |
|---|---|---|---|---|
| 5.1 | 3.5 | 1.4 | 0.2 | setosa |
| 4.9 | 3.0 | 1.4 | 0.2 | setosa |
| 4.7 | 3.2 | 1.3 | 0.2 | setosa |
| 4.6 | 3.1 | 1.5 | 0.2 | setosa |
| 5.0 | 3.6 | 1.4 | 0.2 | setosa |
アイリスの花の品種(species)はカテゴリ変数で、3種類×50行のデータがありますので、花の品種をグループとして因果探索していきます。
Python コード
比較のためDirectLiNGAMとMultiGroupDirectLiNGAMの両方を用いて因果探索を実行するPythonコードを示します。
最初にデータセットを読み込みます。DirectLiNGAMはnumpy配列、MultiGroupDirectLiNGAMはグループごとにnumpy配列に分割し、リストに格納しています。
import lingam
from lingam.utils import make_dot
import seaborn as sns
# アイリスデータセットを読み込み
df = sns.load_dataset('iris')
labels = list(df.columns)[:-1]
X = df.drop('species', axis=1).to_numpy()
# 品種ごとに分割
setosa_array = df[df['species'] == 'setosa'].drop('species', axis=1).to_numpy()
versicolor_array = df[df['species'] == 'versicolor'].drop('species', axis=1).to_numpy()
virginica_array = df[df['species'] == 'virginica'].drop('species', axis=1).to_numpy()
X_list = [setosa_array, versicolor_array, virginica_array]
次にDirectLiNGAMを実行し、結果を因果グラフで表示します。
# 因果探索(DirectLiNGAM)
model_d = lingam.DirectLiNGAM()
model_d.fit(X)
print(f'Causal order: {model_d.causal_order_}')
# Causal order: [2, 3, 1, 0]
# 因果グラフ
make_dot(model_d.adjacency_matrix_, labels=labels)
次にMultiGroupDirectLiNGAMを実行し、結果を因果グラフで表示します。
# 因果探索(MultiGroupDirectLiNGAM)
model_group = lingam.MultiGroupDirectLiNGAM()
model_group.fit(X_list)
print(f'Causal order: {model_group.causal_order_}')
# Causal order: [3, 2, 1, 0]
# 因果グラフ
make_dot(model_group.adjacency_matrices_[0], labels=labels)
make_dot(model_group.adjacency_matrices_[1], labels=labels)
make_dot(model_group.adjacency_matrices_[2], labels=labels)
結果
上記のコードを実行すると、DirectLiNGAMでは全データセットで因果探索した推定結果が、MultiGroupDirectLiNGAMでは各グループに対する推定結果が得られます。
推定結果として因果順序(causal_order_)と隣接行列(adjacency_matrix_またはadjacency_matrices_)を取得しています。また、隣接行列はmake_dot関数で因果グラフとして確認しています。
推定された因果順序は以下の通りです。petal_lengthとpetal_widthの順序が逆になっています。
- DirectLiNGAM: petal_length → petal_width → sepal_width → sepal_length
- MultiGroupDirectLiNGAM: petal_width → petal_length → sepal_width → sepal_length
DirectLiNGAMで推定された因果グラフは以下の通りです。
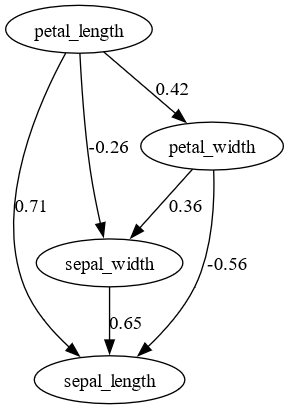
DirectLiNGAMの実行結果
MultiGroupDirectLiNGAMで推定された因果グラフは以下の通りです。
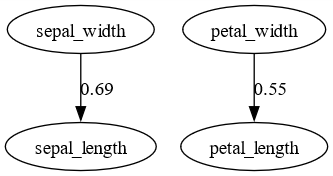
MultiGroupDirectLiNGAMの実行結果 (品種:'setosa')

MultiGroupDirectLiNGAMの実行結果 (品種:'versicolor')

MultiGroupDirectLiNGAM の実行結果 (品種:'virginica')
petal_length→petal_widthの因果の方向は逆ですが、それ以外の方向は一致しているようです。
また、MultiGroupDirectLiNGAMの因果グラフは、グループ間で因果効果や因果の有無が異なることが分かります。
| 因果 | setosa | versicolor | virginica |
|---|---|---|---|
| petal_width → petal_length | 0.55 | 1.87 | 0.65 |
| petal_width → sepal_width | 1.05 | 0.54 | |
| petal_length → sepal_width | 0.15 | ||
| petal_length → sepal_length | 0.83 | 1.00 | |
| sepal_width → sepal_length | 0.69 |
これにより、DirectLiNGAMでは分からなかったグループ毎の因果構造の細かな違いを確認することができます。
宣伝:Causalas 複数データセットの因果探索機能
弊社が開発している Causalas にも、「複数データセットの因果探索」という機能があります(v1.1以降)。
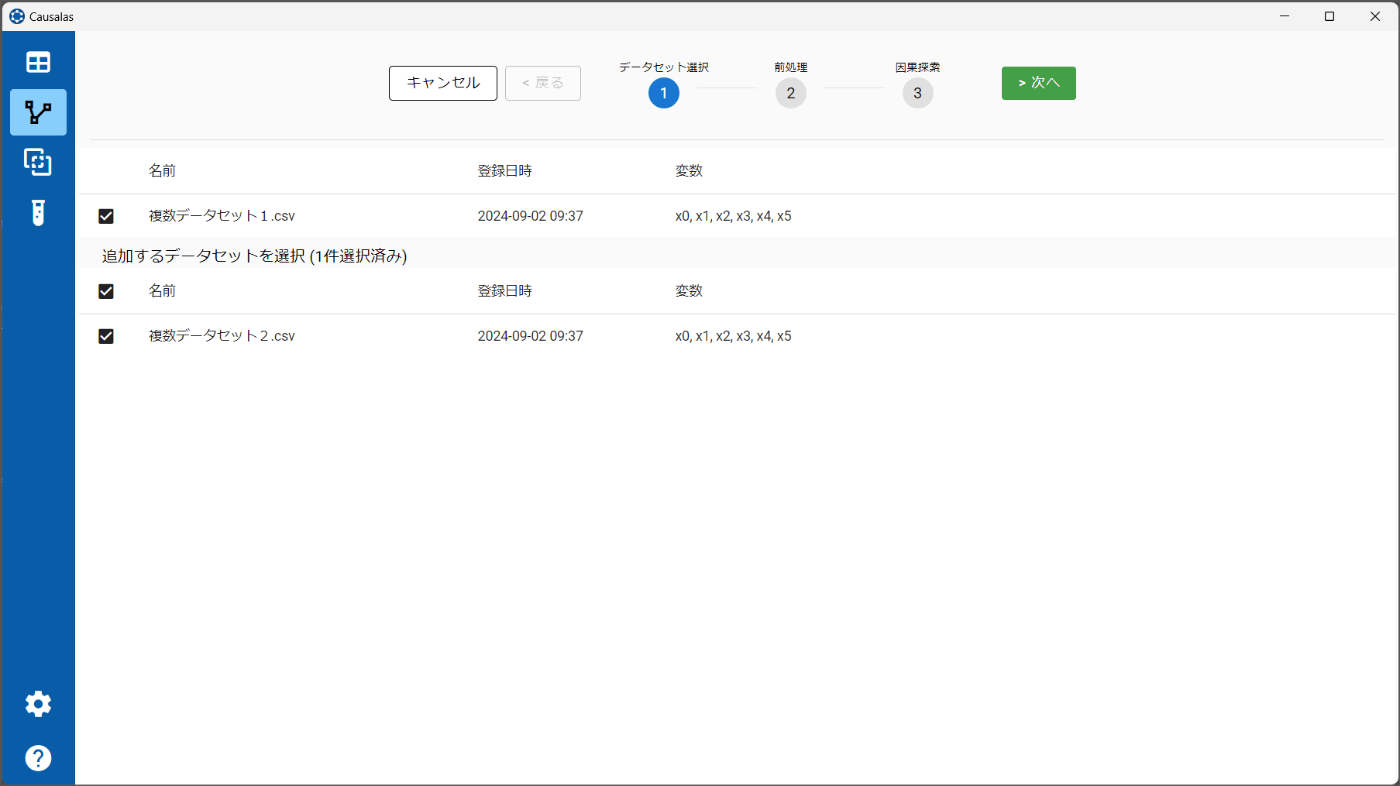
因果探索したいデータセットを複数選択すると、MultiGroupDirectLiNGAMによる因果探索を実行することができます。
また、因果グラフの比較機能を使用することで2つのグループの因果グラフを比較することができます。[差分を強調]スイッチをオンにすると、増減したエッジや方向の違うエッジが強調表示されます。

おわりに
本記事では、MultiGroupDirectLiNGAMを用いて複数グループのデータセットをまとめて因果探索する方法を紹介しました。MultiGroupDirectLiNGAMを使用することによって、複数のグループで因果順序を共有したまま同時に推定することができ、因果効果や因果の有無の違いなどの詳細をグループ毎に確認することができます。
-
S. Shimizu. Joint estimation of linear non-Gaussian acyclic models. Neurocomputing, 81: 104-107, 2012. ↩︎
Discussion