フルスタックエンジニア的に活動してきた僕がアンプラットにインターンにきた その1

この記事の要約
- フルスタックエンジニア的に活動していた人材がアンプラットにインターンにきた。
- 基礎知識の学習〜実際の解析まで確認してみたら、エンジニア的なスキルだけでは難しい部分も多かった。
- コマンド操作を用いた解析はハードルが高かったので、ひとまず、自社開発の解析プラットフォーム「ANCAT」をもちいて、RNA-seq解析をやってみた。
- エンジニアがバイオインフォマティシャンになっていく過程をZennでお見せしていこうと思う。
はじめに
皆さんこんにちは。
アンプラットです。
8/5付けで、当社に新しいインターン生がやってきました。
すでにフルスタックエンジニア的に活動されていた方ということで、当社の期待も高まっています!!
とはいえ、生物学と情報学の複合学問であるバイオインフォマティクスはすぐにできるものでもないですし、
何より、当社の場合、最先端の技術を取り入れている部分もあるので、学んでもらえることも多いでしょう。
バイオインフォマティシャンは、バイオを学んでいた人が、コマンド操作、プログラミング技術等を習得するのが王道だと思いますが、社会的な待遇の違いもあり、エンジニアがバイオインフォに入ってくるのはさほど多くないので、この記事では「エンジニアがバイオインフォマティシャンっぽくなっていく過程」をお見せできればと思います。
まずは、エンジニアが初めてRNA-seqに触れたときの反応を御覧ください!
この記事では、当社のインターン生「Oさん」が、当社に入社して初めてバイオインフォを学び、RNA-seq解析を行うまでのドキュメンタリーです。
※ 記事は当社社員、役員でも確認をしており、ある程度の信頼性は担保できていますが、完全ではないので、そこはご認識の上、ご覧くださいませ。
ANPLATでのインターン
インターン参加経緯
自分は過去にwebエンジニアとしてフロントエンドやバックエンド開発をしていました。大学での選考は合成生物学やゲノミクスで、バイオインフォマティクスに興味があり、今回インターンに参加させていただくことになりました。
バイオインフォマティクスの基礎知識を学んで
インターンの初期段階で、バイオインフォマティクスの基礎知識を学びました。これまでの開発経験や大学での勉強の経験が役立つと思っていましたが、実際には新しい概念や技術が多く、難しいながらもとても興味深かったです。
バイオインフォマティクスを体系的に学ぶにつれ、データの解析方法が多岐にわたることを知りました。特に、DNAやRNAの配列データを扱う際の専門用語や手法の理解が難しかったです。
RNA-seqの理解を目指して
RNA-seq(RNAシーケンシング)は、遺伝子発現を解析するための手法です。用意していただいた勉強資料を活用し、RNA-seqの基礎を学びました。
学習内容
-
RNA-seqの基本概念: RNA-seqは、次世代シーケンシング(NGS)技術を用いてmRNAやmiRNAなどを抽出し、その発現量を調べる解析手法です。近年では、cDNAへの逆転写を行わずに直接RNAを読むDirect RNA解析も行われるようになっています。
-
データ解析の流れ: RNA-seq解析は、以下のステップで行われます。
-
Quality Control: 配列データの品質を確認し、低品質な塩基や短すぎる配列を除去します。TrimmomaticやCutadaptなどのツールが用いられます。
-
poly-A tailの除去: 実験的に付加されたpoly-A tailを除去します。これもTrimmomaticやCutadaptが使用されます。
-
Mapping: 配列データを参照ゲノムにマッピングします。HISAT2やSTARなどのツールが一般的です。
-
Read Count: 各遺伝子にマッピングされたリードの数をカウントします。featureCountsやStringTieが使用されます。
-
感想
RNA-seqの解析フローを学ぶことで、データがどのように処理され、最終的な遺伝子発現データが得られるのかを理解することができました。特に、データのクオリティコントロールやマッピングのステップが正確に行われることで信頼性の高いデータ解析が可能になるため、その重要性を実感しました。
さらに、RNA-seq解析の各ステップにおいて、使用するツールの選定やパラメータ設定が結果に大きく影響することを学びました。特に、Mappingの際に使用するツールやその設定は、最終的な解析結果に直結するため、慎重に選ぶ必要があります。このあたりの詳しい知識が自分にはまだないため、これから学んでいきたいと思います。
ANCATでRNA-seqを実行
次に、ANCATを用いてRNA-seqを実行しました。
ANCATを使うとGUIで解析を行えるので、自分で解析を行うのに比べて非常に簡単に解析を行うことができました。
実行時
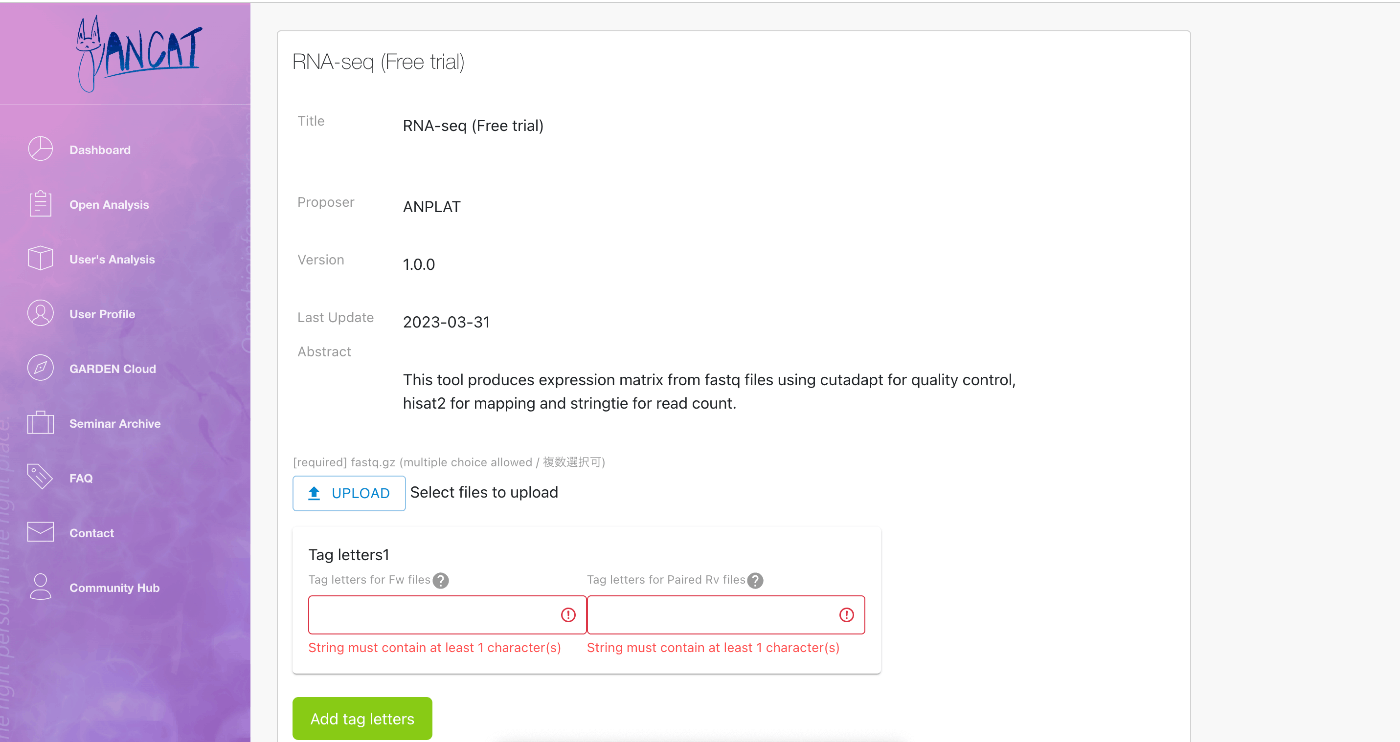

結果

感想
RNA-seqの実行を通じて、データ解析の流れを体験することができました。特に、結果の解釈において、どの遺伝子がどのように発現しているのかを視覚化するプロセスが非常に興味深かったです。
次回の記事では、コマンドレベルで解析を行う過程をお見せします。お楽しみに!
さいごに
いかがでしたか?
Oさんは、エンジニアとして活動してきたといえど、
生物学的な教養も深く、スムーズに解析自体の理解に取り組めていましたね。
次回はコマンドで解析し、Pathway解析くらいまでいく姿が見れるかな?
次回をお楽しみに!!

Discussion