Adversarial Random ForestsによるテーブルデータのAugmentation・モックデータ生成
はじめに
こんにちは。株式会社アイデミーデータサイエンティストの中沢(@shnakazawa_ja)です。
本記事ではAdversarial Random Forestsを使ったテーブルデータの生成について、RおよびPythonでの実装を紹介します。
Adversarial Random Forests (ARF) とは
ARFは2023年にProceedings of The 26th International Conference on Artificial Intelligence and Statisticsに採択された論文で提案された、テーブルデータに対して密度推定と生成モデリングを行う高速な手法です[1]。
その名の通りGAN[2]とRandom Forestを組み合わせた手法で、生成と識別を交互に繰り返すことで元データの特性を学習し、元のテーブルデータと類似したデータを生成できます。
テーブルデータの生成は
- (画像データで行われるような) データの増幅/Data Augmentation
- 個人情報・秘匿情報をマスクしたモックデータの生成
といった場面での活用可能性があり、動作も非常に軽快であるため、適切に用いれば強力な武器になることが期待されます。(懸念点は「おわりに」で議論)
Rでの実装
生成元となるデータとして、本稿ではIrisを用います[3]。
機械学習のド定番、アヤメ分類のデータセットですね。
まずはパッケージのインストール。
install.packages("arf")
そして、データを生成します。
library(arf)
arf <- adversarial_rf(iris) # ARFモデルの訓練。元となるデータを渡す
psi <- forde(arf, iris) # 訓練されたモデルから密度推定
synth_data <- forge(psi, n_synth=1000) # 推定された密度からデータを生成。n_synthで生成するデータサイズを指定
たった4行で完了です!簡単!
出力されたデータを見てみると、ラベル含め確かにそれっぽい値が割り振られています。

より正確に、データの分布を可視化してみましょう。
library(ggplot2)
library(gridExtra)
# 分布のプロット
cols_to_plot <- names(iris)[1:4] # アヤメの形態についてのカラムをプロットする
plots_list <- list()
for (col in cols_to_plot) {
# それぞれの形態情報についてdensity plotを作成。オレンジ:元データ、青:合成データ
p <- ggplot() +
geom_density(aes(x=!!sym(col)), data=iris, fill="orange", alpha=0.3) +
geom_density(aes(x=!!sym(col)), data=synth_data, fill="skyblue", alpha=0.3) +
facet_wrap(~Species, scales="free") +
labs(title=paste(col), x=col, y='Density')
plots_list[[col]] <- p
}
# プロットを並べる
do.call(grid.arrange, c(plots_list, ncol=2))
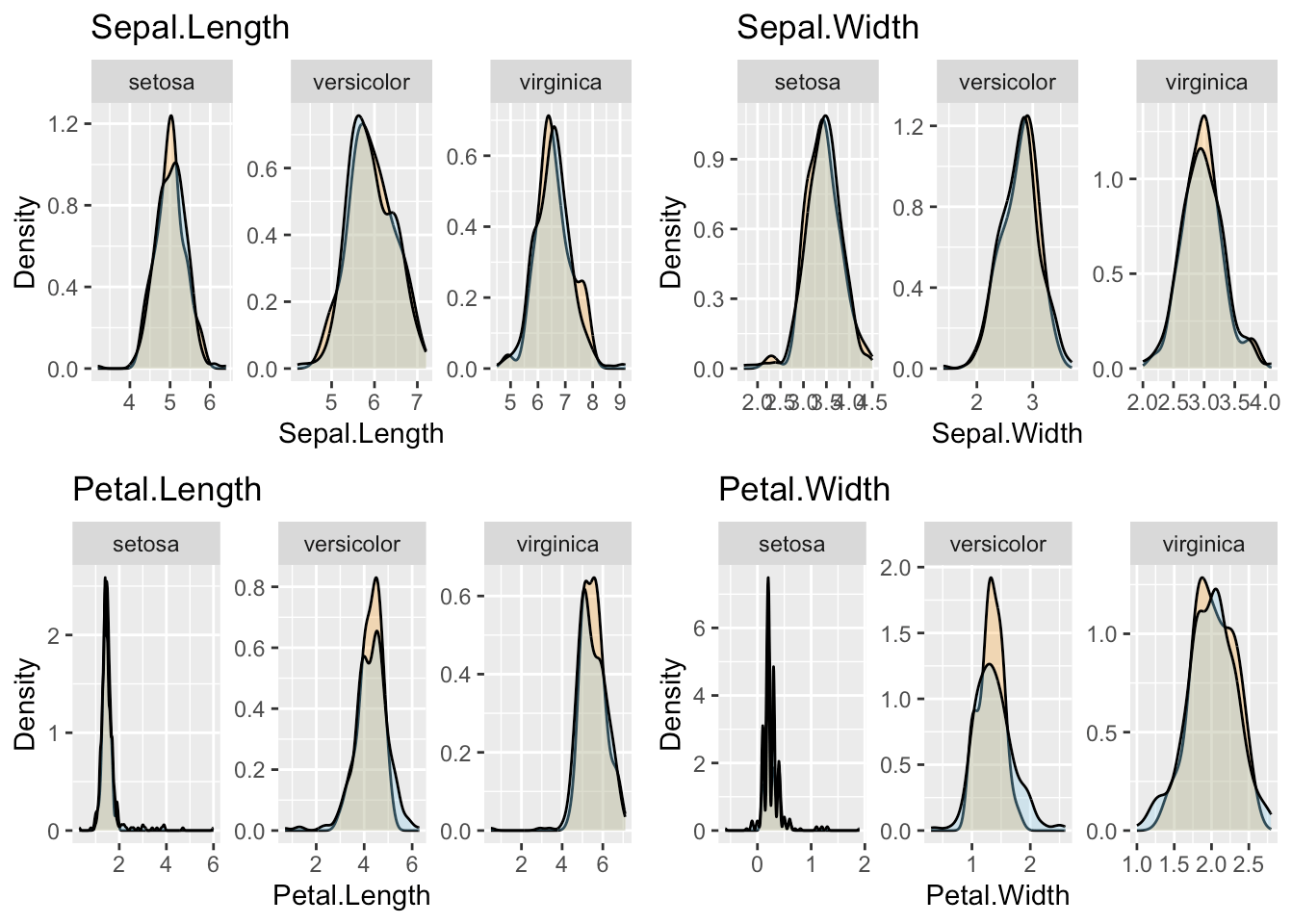
オレンジ:元データ、青:合成データです。確かに元のデータと似た分布の模擬データを生成できていることが確認できました。
Pythonでの実装
Pythonでも実装をしてみましょう。まずはパッケージのインストール。"arf" は別パッケージなので注意してください。
pip install arfpy
モジュールとデータを読み込みます。
from sklearn.datasets import load_iris
from arfpy import arf
import pandas as pd
iris = load_iris()
df = pd.DataFrame(data=iris['data'], columns=iris['feature_names'])
df["species"] = iris.target_names[iris.target]
df["species"] = df["species"].astype("category")
df
これを元とし、模擬データを生成してみましょう。
my_arf = arf.arf(x=df) # ARFモデルの訓練。元となるデータを渡す
my_arf.forde() # 訓練されたモデルから密度推定
synth_data = my_arf.forge(n=1000) # 推定された密度からデータを生成。nで生成するデータサイズを指定
synth_data
こちらも数行で完了です!簡単!
最後に、データの分布を可視化して、生成データが元データと似ているかを確認します。
import matplotlib.pyplot as plt
import seaborn as sns
plt.style.use('ggplot')
fig, axes = plt.subplots(nrows=4, ncols=3, figsize=(10, 15))
axes = axes.flatten()
for i, col in enumerate(df.columns[:4]):
for j, species in enumerate(df['species'].unique()):
ax_idx = i * 3 + j
subset_iris = df[df['species'] == species]
subset_synth = synth_data[synth_data['species'] == species]
sns.kdeplot(subset_iris[col], ax=axes[ax_idx], label=f'Original', fill=True, alpha=0.3, color='orange')
sns.kdeplot(subset_synth[col], ax=axes[ax_idx], label=f'Synth', fill=True, alpha=0.3, color='skyblue')
axes[ax_idx].set_title(f'{species} - {col}')
axes[ax_idx].set_xlabel(col)
axes[ax_idx].set_ylabel('Density')
if j == 0:
axes[ax_idx].legend()
plt.tight_layout()
plt.show()
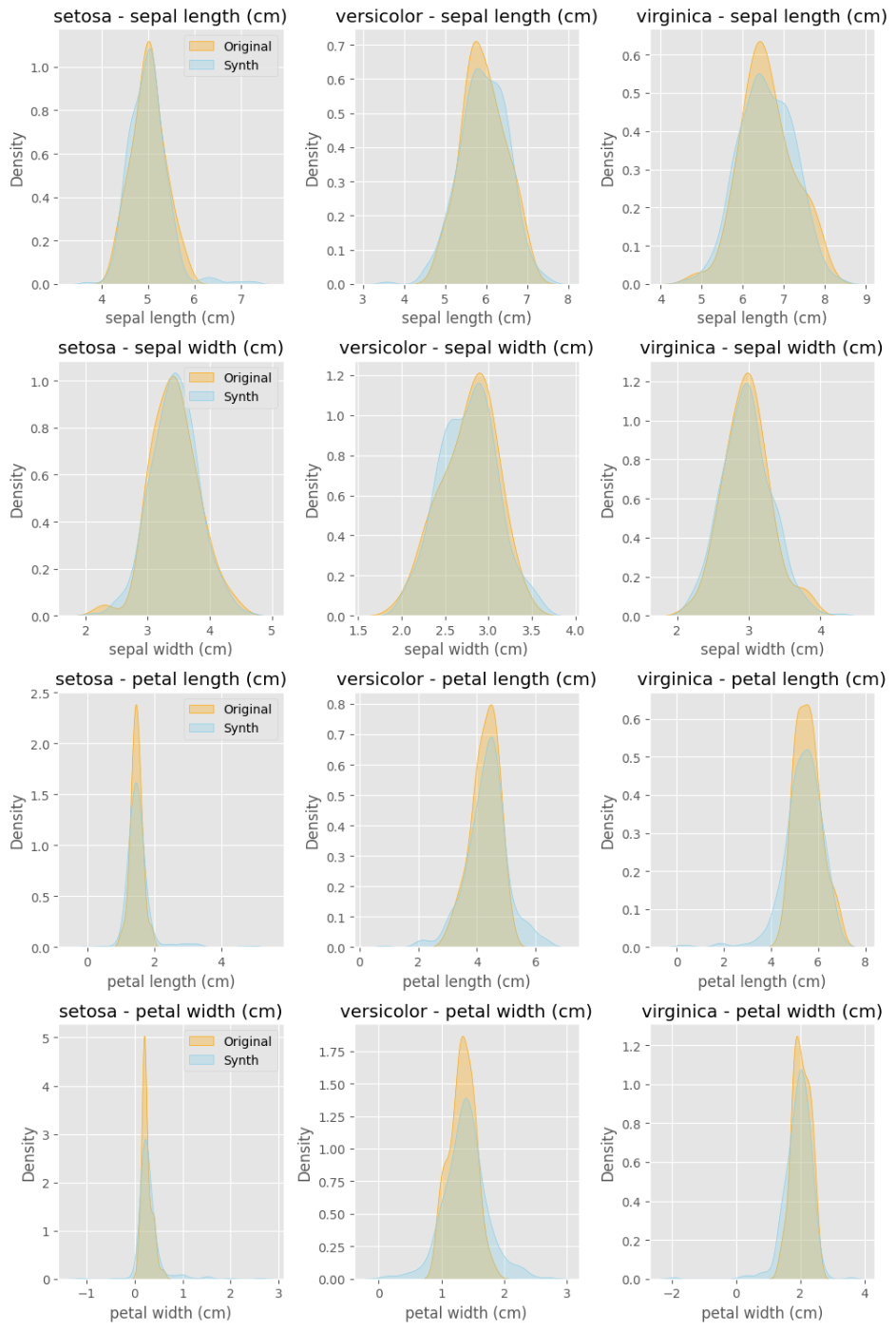
Pythonでも、元データと似た分布の模擬データの生成が確認できました。
おわりに
本稿ではARFを用いたテーブルデータの生成方法を紹介しました。冒頭で述べたように、テーブルデータのAugmentationやconfidential情報をマスクした模擬データの生成などに活用が期待されます。一方で、使用に当たっては明確に注意が必要な手法でもあります。
まず、元のデータに含まれる特徴をそのまま再現・増幅してしまう点が(いいところでもあるのですが)注意すべきところです。元のデータが偏っていると、その偏りごと再現してしまうため、元データの(本来着目したくない)特徴にオーバーフィットしてしまう懸念があります。そのため、根本的なデータサイズ不足の解消にはなりません。
また、どれだけ似ているといってもフェイクデータであることに変わりはありません。Augmentation目的で使うのであれば、訓練データのみを増幅し、評価は本物のデータで行う、といった(画像のAugmentation同様の)取り扱いも必要です。
そして、モックデータ生成に用いる場合は、本当に隠すべき情報が隠せているのか?隠した情報が再現されてしまわないか? に十二分に注意しないといけません。例えば、実験条件と結果の組み合わせはピンポイントの再現でなくても十分に意味のある情報です。「分布がわかるだけでも個人と紐付けられてしまう」状況もありえるでしょう。
しかし、こうした課題に注意を払っていれば非常に簡便に使える、テーブルデータ向けの軽量な生成モデルということで、応用の範囲はとても広いと想定されます。
本稿が皆様のデータ分析のお役に立てば幸いです。
-
論文の解説はこちらで → https://github.com/TeamAidemy/ds-paper-summaries/issues/25 ↩︎
-
生成器は訓練されないので、厳密にはGANとは異なります。 ↩︎
-
Rには多くのデータセットが同梱されています。 https://www.math.chuo-u.ac.jp/~sakaori/Rdata.html ↩︎
Discussion