DynamicVizで次元削減から恣意性を除く
はじめに
こんにちは。株式会社アイデミーデータサイエンティストの中沢(@shnakazawa_ja)です。
弊社のデータサイエンスグループでは論文の日本語サマリーを作る取り組みを行っています。本稿ではそちらで以前に紹介した論文 "DynamicViz" を実際に使ってみよう、というお話をします。
また、本記事に記載したコードはNotebookにまとめGitHubにて公開しています。
DynamicVizとは
DynamicVizとは2022年5月にbioRxivにプレプリントとして公開、同年12月にNature Computational Science誌に受理された論文で提案されたフレームワークで、一言でまとめると「t-SNEやUMAPといった既存の次元削減手法を拡張するツール」です[1][2]。
手法選定やハイパラ調整プロセスへの数値指標導入や、解釈に至る道筋からの恣意性の削減など、既存手法の弱みのカバーを試みており、結果・結論の信頼性を高める一つのアプローチとして用いうると考えられます。
論文の詳細については日本語サマリーで取り扱っていますので、本稿では使い方にフォーカスしていきます。
環境構築
pip install dynamicvizで一発です。
この後のコードは Python 3.8.16, dynamicviz 0.0.3 を用いて実行します。
データの準備
結果の比較のため、本稿では以前執筆した次元削減に関する記事と同じく2022年のプロ野球投手データを題材として用います。
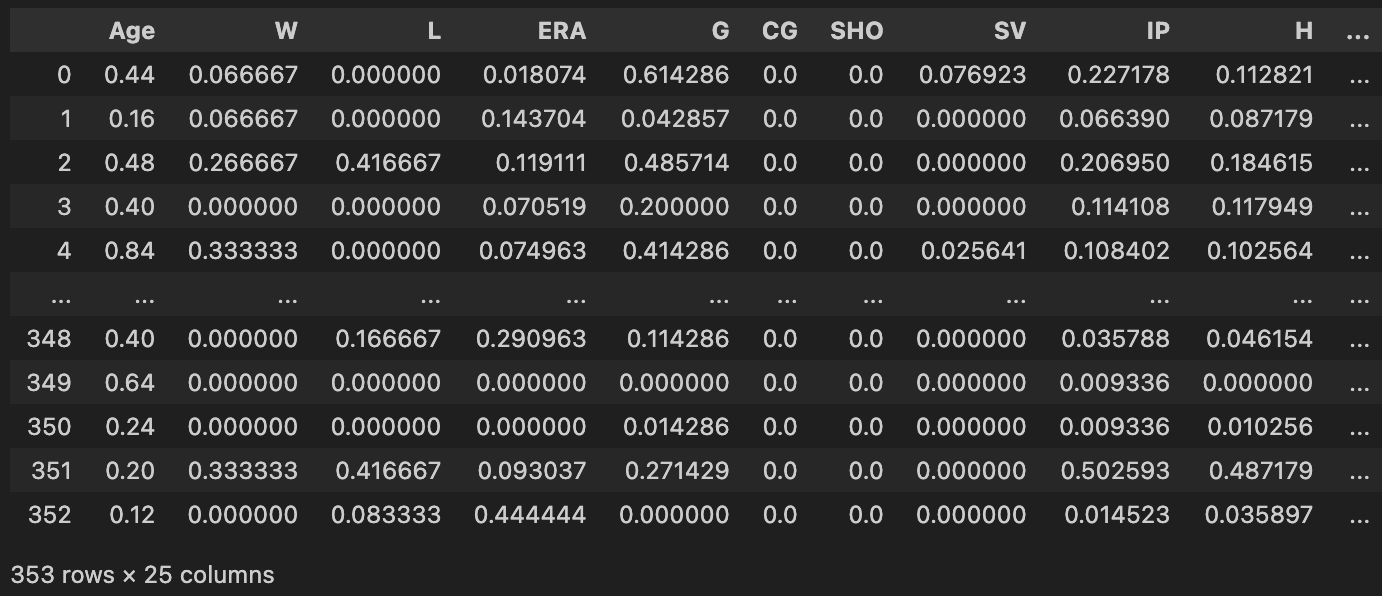
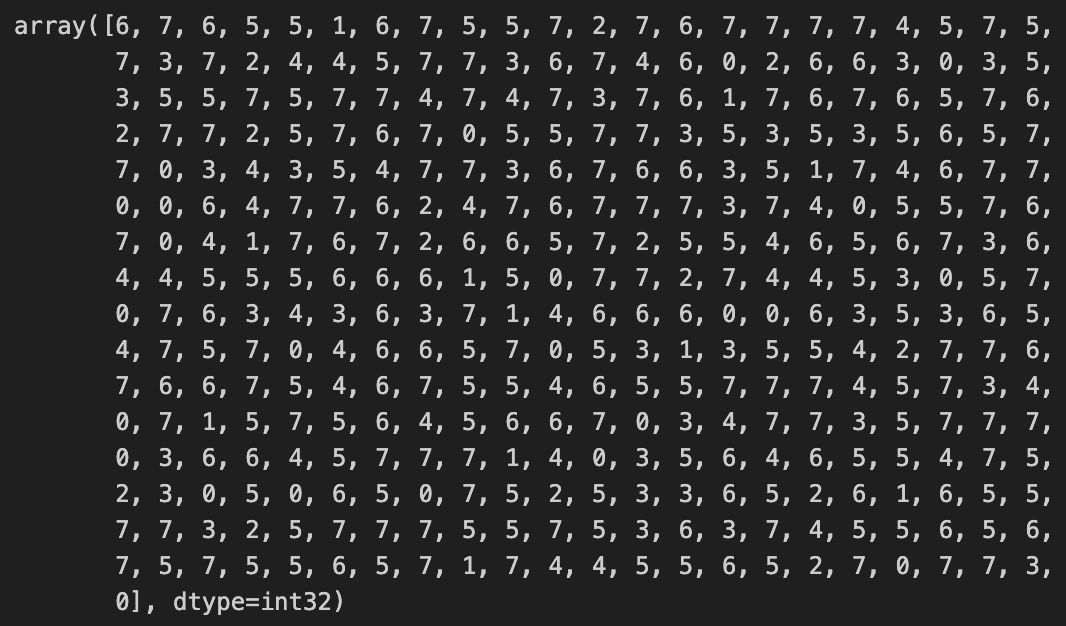
前処理とクラスタリングまでが終わった状態であるである(すなわち、df_features_mmに前処理を終わらせた投手成績と、clustersにクラスタリングの結果が格納されている)としてこの先進めていきます。
df_features_mm
clusters
基本的な使い方
まず、DynamicVizの基本的な使い方をさらいましょう。本家のtutorial.ipynbを参考にしながら進めます[3]。
モジュールのインポートとデータの変形
まずはモジュールのインポートとデータの変形を行います。
from dynamicviz import boot, viz, score
X = df_features_mm.to_numpy()
y = pd.DataFrame(clusters, columns=['Cluster'])
Bootstrap法よるサンプリング
DynamicVizではBootstrap法で実データから再サンプリングを行います。
out = boot.generate(
X, # データ。numpy.ndarray
Y=y, # グループのラベル
method='umap', # 可視化手法
B=4, # ブートストラップ回数
save='bootstrap.csv', # ブートストラップ結果の保存先
random_seed=452, random_state=452 # 乱数シード
)
out
Static Visualization
次に、Static Visualizationを行います[4]。Bootstrapで増やしたデータを重ね合わせ、図示のバイアスを減らそうという発想です。
fig = viz.stacked(
out, # ブートストラップしたデータ
'Cluster', # グループラベルのカラム名
show=True, # 図を表示するか
save='StaticViz.png', # 保存先
# 以下、図を作成するときのパラメータ
alpha=0.2, title='Static Visualization', width=5, height=4,
xlabel='UMAP 1', ylabel='UMAP 2', dpi=150, marker='x', s=20,
show_legend=True, solid_legend=True, legend_fontsize=12,
colors=['green','mediumpurple','orange','yellow','blue','magenta','brown','gray']
)
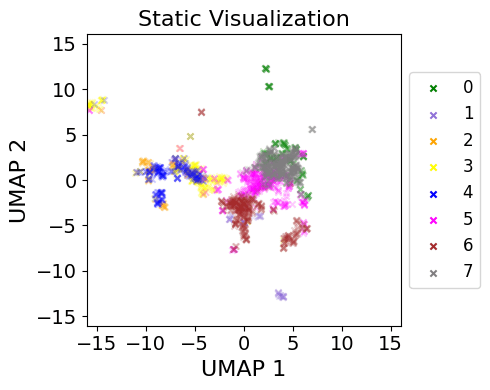
Bootstrap回数がB=4と小さいため、少し歪なかたちになっています。論文での使用例を見ても B=20くらいは指定したほうが良さそうです。
Variance Scoreの計算
Variance Scoreが「クラスターの安定性を評価する数字」とされており、どの次元削減手法が最適かの定量指標となります(0に近いほどよい)。
Global, Local, Predefined neighborhoodsの3種類のオプションがあります。Globalは全ての観測データ間で、Localは指定したk個の近くの観測データ間で、Predefined...はユーザーが指定した観測データ間でスコアを計算します[5]。
variance_scores = score.variance(
out, # ブートストラップしたデータ
method='global' # 評価方法
)
# Global Variance Scoreの平均値を表示
print('Global variance scores ', variance_scores.mean())
# Global Variance Scoreのバラツキを可視化
plt.hist(variance_scores)
plt.show()
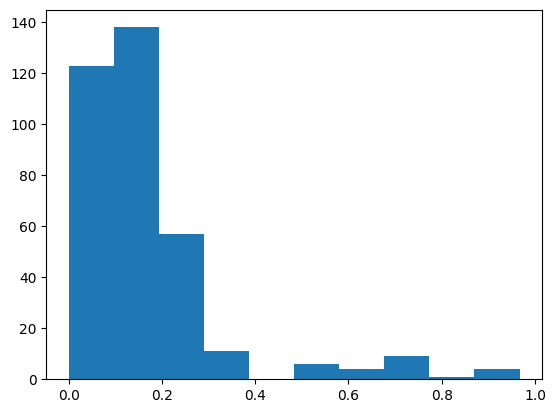
約350件のデータの計算に(筆者の環境では)7秒弱と、結構な時間がかかるのが本手法の難点ですね。
Globalでは全てのデータ間で計算が行われるため長い時間がかかってしまっています。そこで別オプションとしてmethod='random', k=50のような指定も用意されています。これはランダムに選んだk個のサンプル間との計算になるので、ある程度の精度を保ちつつ高速化することができます[6]。
実践的な使い方
さて、ここからはDynamicVizを実戦投入していきます。手元のデータに対して最適な次元削減手法とそのハイパーパラメータを探してみましょう。
モジュールのインポートとデータの変形
同上。
from dynamicviz import boot, viz, score
X = df_features_mm.to_numpy()
y = pd.DataFrame(clusters, columns=['Cluster'])
最適な次元削減手法を定量的に決定する
まずはどの手法が最適かを探索します。DynamicVizは t-SNE, MDS, LLE, MLLE, Isomap, UMAP の6手法に対応しています。
それぞれの手法のデフォルト設定で、Global Variance Scoreを比較してみます。
論文での使用例にあわせ、B=20としています。
methods = ['tsne', 'mds', 'lle', 'mlle', 'isomap', 'umap']
scores = []
for method in methods:
print(f'===={method}====')
if method == 'mlle': # MLLEは eigen_solver 引数を指定しないと動かないため別処理
out = boot.generate(X, Y=y, method=method, B=20, random_seed=452, random_state=452, eigen_solver="dense")
elif method == 'isomap': # isomapではrandom_stateが引数として存在しない
out = boot.generate(X, Y=y, method=method, B=20, random_seed=452)
else:
out = boot.generate(X, Y=y, method=method, B=20, random_seed=452, random_state=452)
variance_scores = score.variance(out, method='global')
scores.append(variance_scores)
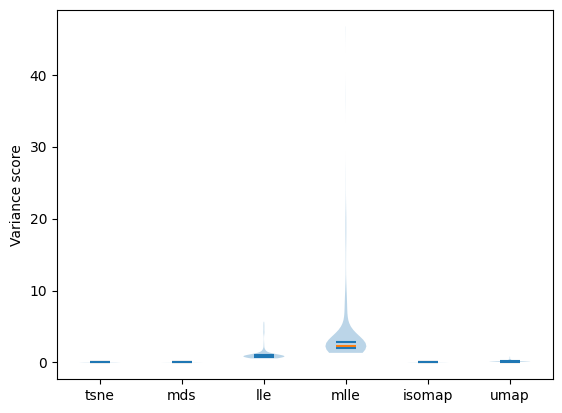
# violin plotでmethodsごとのvariance scoreを表示
fig=plt.violinplot(scores, showmedians=True, showextrema=False, quantiles= [[0.25, 0.75]]*len(methods))
# plt.ylim(0, 0.2)
plt.xticks(range(1, len(methods)+1), methods)
plt.ylabel('Variance score')
fig['cmedians'].set_color('C1')
plt.show()
縦軸の倍率を変えて再プロット↓
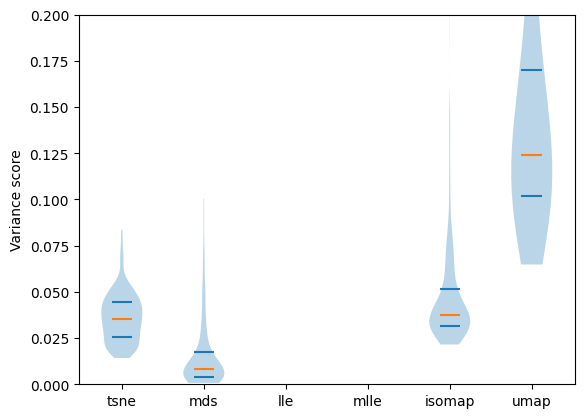
ここでは中央値をオレンジ、25,75パーセンタイルを青線でマークしています。
Variance Scoreは小さいほど良いスコアであるため、今回のデータにはMDSが(Variance Scoreを指標とすると)最適であると考えられます。
最適なハイパーパラメータを定量的に決定する
次にMDSのハイパーパラメータ調整を行います。こちらもGlobal Variance Scoreでの比較を行います。
ここでは簡単のために max_iter だけを振ってみましょう[7]。
params = [50,150,300,500,1000]
scores = []
for param in params:
print(f'==== max_iter:{param} ====')
out = boot.generate(X, Y=y, method='mds', B=20, random_seed=452, random_state=452, max_iter=param)
variance_scores = score.variance(out, method='global')
scores.append(variance_scores)
# 折れ線グラフでmax_iterごとのvariance scoreの中央値と25,75パーセンタイルを表示
import numpy as np
plt.plot(params, [np.median(x) for x in scores], marker='o')
plt.fill_between(params, [np.quantile(x, 0.25) for x in scores], [np.quantile(x, 0.75) for x in scores], alpha=0.3)
plt.xlabel('max_iter')
plt.ylabel('Variance score')
plt.show()
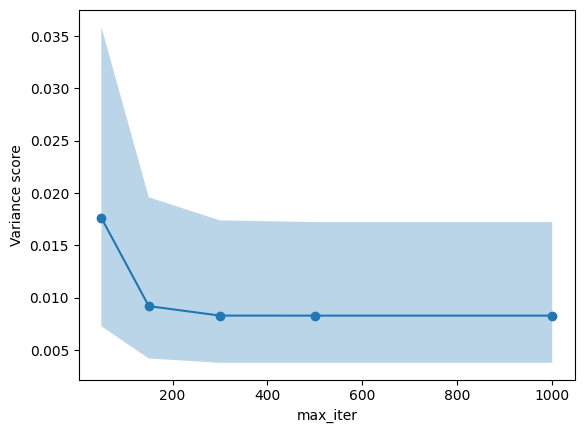
以上より、max_iterは300で最適なスコアに到達するには十分であると考えられます。
Static Visualizationで次元削減の結果を解釈する
最後に、選択された手法、ハイパーパラメータを使ってStatic Visualizationを作ります。
out = boot.generate(X, Y=y, method='mds', B=20, save=f'bootstrap_mds_optimized.csv', random_seed=452, random_state=452, max_iter=300)
fig = viz.stacked(
out, # ブートストラップしたデータ
'Cluster', # グループラベルのカラム名
show=True, # 図を表示するか
save='StaticViz.png', # 保存先
# 以下、図を作成するときのパラメータ
alpha=0.2, title='MDS Static Visualization', width=5, height=4,
xlabel='MDS 1', ylabel='MDS 2', dpi=150, marker='o', s=20, show_legend=True, solid_legend=True, legend_fontsize=12,
colors=['green','mediumpurple','orange','yellow','blue','magenta','brown','gray']
)
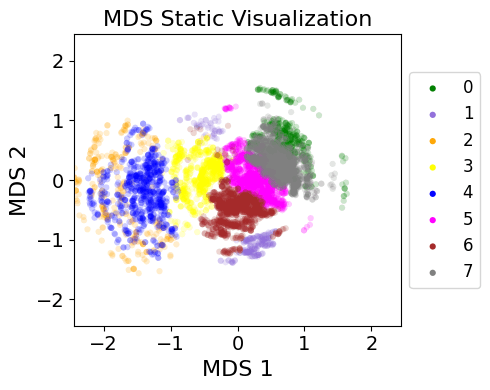
過去記事のケースと比較してみましょう。

パッと見た印象として、
- 全体的な位置関係は保たれている。
- 成績の連続的な変化が表現できている。
- 擬似的な時系列変化とみなした分析も引き続き行えるだろう。
- 先発とリリーフでの断絶がなくなった。
- 連続的な成績変化であることを反映している。
- 先発・中継ぎの両方を担う投手も実際に存在するため、MDSのプロットがより現実を反映しているかもと考えられる。
- クローザーのクラスター(紫: 1)が2つに分かれている。
- もしかしたらクローザーの中でも2タイプに分かれるのかもしれない。
- そもそも k-means によるクラス分けが不適切な可能性もある。
といったことが考えられます。
さいごに
DynamicVizはこれまで恣意的に(ややもすると見栄えが最も良くなるように)選択されていた次元削減の設定の諸々を体系的に処理できるようにする一つの指標を提案し、データの解釈からの恣意性の削減を目指しています。
一方で、random_seed次第でvariance scoreの順位が入れ替わることもあり、必ずしも最適な設定が一意に定まるわけではないという点には留意が必要です[8][9]。
Variance Scoreが常に適切な指標なのか?という点も注意すべきですが、データを可視化して理解するための明快な道筋として、まずやってみる手法としては悪くないのではないでしょうか。
本記事が皆様のデータ分析のお役に立てば幸いです。
-
Nat. Comput. Sci.へのアクセス権がないので、本稿はbioRxiv版に準拠した理解で進めます。 ↩︎
-
GitHubはこちら。https://github.com/sunericd/dynamicviz ↩︎
-
本稿ではこのうちの一部しか紹介しません。 ↩︎
-
ver.0.0.3だと
cmapが正しく動作しないため、colorsで個別に指定しています。 ↩︎ -
特別な理由がない場合は基本的にはGlobalを使っておけば間違いないかと思いますが、LocalやPredefined...を使うことでクラスター内の安定性をより重視できるようです。 ↩︎
-
k=50とすると、同じデータでも約1秒で計算が完了します。globalとrandomのVariance Scoreの相関係数は0.9程度。 ↩︎ -
MDSはハイパラ調整がt-SNEやUMAPほど劇的な影響を与えないので、記事の題材としては正直微妙なものが選ばれたなと思っています。 ↩︎
-
random_seedを変えるとスコアが劇的に変わるという状況は観測範囲では見られていないので、こういうときは「いずれのハイパラを選んでも大した違いがないのでどちらでも良い」と解釈すべきでしょう。 ↩︎
-
random_seedの選定に本手法を使っちゃだめですよ。 ↩︎
Discussion